数据处理和分析专家拥有许多用于创建分类模型的工具。开发此类模型的最流行,最可靠的方法之一是使用随机森林(RF)算法。为了尝试改善使用RF算法构建的模型的性能,可以使用模型超参数的优化(Hyperparameter Tuning,HT)。
此外,存在一种广泛的方法,根据该方法,使用主成分分析处理数据,然后再将数据传输到模型中。 ,PCA)。但是值得使用吗?射频算法的主要目的不是帮助分析师解释特征的重要性吗?是的,在分析RF模型的“特征重要性”时,使用PCA算法可能会使每个“特征”的解释稍微复杂一些。但是,PCA算法减小了特征空间的尺寸,这可能导致RF模型需要处理的特征数量减少。请注意,计算量是随机森林算法的主要缺点之一(也就是说,完成模型可能需要很长时间)。 PCA算法的应用可能是建模的一个非常重要的部分,尤其是在它们具有数百甚至数千个功能的情况下。因此,如果最重要的事情就是简单地创建最有效的模型,并且同时牺牲了确定属性重要性的准确性,那么PCA可能值得一试。现在到了重点。我们将使用乳腺癌数据集-Scikit-learn“乳腺癌”。我们将创建三个模型并比较它们的有效性。即,我们正在谈论以下模型:
,PCA)。但是值得使用吗?射频算法的主要目的不是帮助分析师解释特征的重要性吗?是的,在分析RF模型的“特征重要性”时,使用PCA算法可能会使每个“特征”的解释稍微复杂一些。但是,PCA算法减小了特征空间的尺寸,这可能导致RF模型需要处理的特征数量减少。请注意,计算量是随机森林算法的主要缺点之一(也就是说,完成模型可能需要很长时间)。 PCA算法的应用可能是建模的一个非常重要的部分,尤其是在它们具有数百甚至数千个功能的情况下。因此,如果最重要的事情就是简单地创建最有效的模型,并且同时牺牲了确定属性重要性的准确性,那么PCA可能值得一试。现在到了重点。我们将使用乳腺癌数据集-Scikit-learn“乳腺癌”。我们将创建三个模型并比较它们的有效性。即,我们正在谈论以下模型:- 基于RF算法的基本模型(我们将简称为RF模型)。
- 与1号模型相同,但其中一种模型是使用主成分方法(RF + PCA)来缩小特征空间的尺寸。
- 与2号模型相同,但使用超参数优化(RF + PCA + HT)构建。
1.导入数据
首先,加载数据并创建Pandas数据框。由于我们使用了Scikit-learn中预先清除的“玩具”数据集,因此之后我们就可以开始建模过程了。但是,即使使用此类数据,也建议您始终通过使用以下应用于数据框(df)的命令对数据进行初步分析来开始工作:df.head() -查看新数据框,看其是否符合预期。df.info()-找出数据类型和列内容的特征。在继续之前,可能需要执行数据类型转换。df.isna()-确保数据中没有值NaN。相应的值(如果有)可能需要以某种方式处理,或者,如果有必要,可能有必要从数据帧中删除整行。df.describe() -找出列中指标的最小,最大值,平均值,找出列中均方根和可能偏差的指标。
在我们的数据集中,一列cancer(癌症)是目标变量,我们希望使用模型预测其值。0表示“没有疾病”。1-《病的存在》。import pandas as pd
from sklearn.datasets import load_breast_cancer
columns = ['mean radius', 'mean texture', 'mean perimeter', 'mean area', 'mean smoothness', 'mean compactness', 'mean concavity', 'mean concave points', 'mean symmetry', 'mean fractal dimension', 'radius error', 'texture error', 'perimeter error', 'area error', 'smoothness error', 'compactness error', 'concavity error', 'concave points error', 'symmetry error', 'fractal dimension error', 'worst radius', 'worst texture', 'worst perimeter', 'worst area', 'worst smoothness', 'worst compactness', 'worst concavity', 'worst concave points', 'worst symmetry', 'worst fractal dimension']
dataset = load_breast_cancer()
data = pd.DataFrame(dataset['data'], columns=columns)
data['cancer'] = dataset['target']
display(data.head())
display(data.info())
display(data.isna().sum())
display(data.describe())
. . , cancer, , . 0 « ». 1 — « »2.
现在,使用Scikit-learn函数拆分数据train_test_split。我们希望为模型提供尽可能多的训练数据。但是,我们需要掌握足够的数据来测试模型。通常,我们可以说,随着数据集中的行数增加,可以认为具有教育意义的数据量也会增加。例如,如果有数以百万计的行,则可以通过突出显示训练数据的90%和测试数据的10%来拆分集合。但是测试数据集仅包含569行。而且对于训练和测试模型而言,还不算什么。因此,为了公平对待培训和验证数据,我们将集合分为两个相等的部分-50%-培训数据和50%-验证数据。我们安装stratify=y 以确保训练和测试数据集与原始数据集的比率分别为0和1。from sklearn.model_selection import train_test_split
X = data.drop('cancer', axis=1)
y = data['cancer']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.50, random_state = 2020, stratify=y)
3.数据缩放
在进行建模之前,您需要通过缩放数据来“居中”和“标准化”数据。由于不同数量以不同单位表示,因此执行缩放。通过此过程,您可以在标牌之间进行“公平竞争”以确定其重要性。此外,我们将y_trainPandas数据类型Series转换为NumPy数组,以便以后模型可以与相应的目标一起使用。import numpy as np
from sklearn.preprocessing import StandardScaler
ss = StandardScaler()
X_train_scaled = ss.fit_transform(X_train)
X_test_scaled = ss.transform(X_test)
y_train = np.array(y_train)
4.基本模型的训练(RF 1号模型)
现在创建型号1。在其中,我们记得只有随机森林算法被使用。它使用所有功能并使用默认值进行配置(有关这些设置的详细信息,请参见sklearn.ensemble.RandomForestClassifier的文档)。初始化模型。之后,我们将对她进行规模化数据培训。可以在训练数据上测量模型的准确性:from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import recall_score
rfc = RandomForestClassifier()
rfc.fit(X_train_scaled, y_train)
display(rfc.score(X_train_scaled, y_train))
如果我们想知道哪些特征对于RF模型预测乳腺癌最重要,我们可以通过查看以下属性来可视化和量化特征严重性指标feature_importances_:feats = {}
for feature, importance in zip(data.columns, rfc_1.feature_importances_):
feats[feature] = importance
importances = pd.DataFrame.from_dict(feats, orient='index').rename(columns={0: 'Gini-Importance'})
importances = importances.sort_values(by='Gini-Importance', ascending=False)
importances = importances.reset_index()
importances = importances.rename(columns={'index': 'Features'})
sns.set(font_scale = 5)
sns.set(style="whitegrid", color_codes=True, font_scale = 1.7)
fig, ax = plt.subplots()
fig.set_size_inches(30,15)
sns.barplot(x=importances['Gini-Importance'], y=importances['Features'], data=importances, color='skyblue')
plt.xlabel('Importance', fontsize=25, weight = 'bold')
plt.ylabel('Features', fontsize=25, weight = 'bold')
plt.title('Feature Importance', fontsize=25, weight = 'bold')
display(plt.show())
display(importances)
可视化标志的“重要性”重要性指标5.主成分法
现在,让我们问一下如何改善基本的RF模型。使用减小特征空间尺寸的技术,可以通过较少的变量显示初始数据集,同时减少确保模型运行所需的计算资源量。使用PCA,您可以研究这些特征的累积样本方差,以便了解哪些特征可以解释数据中的大多数方差。我们初始化PCA(pca_test)对象,指出需要考虑的组件(功能)数量。我们将此指标设置为30,以便在决定所需的组件数量之前查看所有生成的组件的解释方差。然后我们转移到pca_test比例数据X_train使用方法pca_test.fit()。之后,我们将数据可视化。import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.decomposition import PCA
pca_test = PCA(n_components=30)
pca_test.fit(X_train_scaled)
sns.set(style='whitegrid')
plt.plot(np.cumsum(pca_test.explained_variance_ratio_))
plt.xlabel('number of components')
plt.ylabel('cumulative explained variance')
plt.axvline(linewidth=4, color='r', linestyle = '--', x=10, ymin=0, ymax=1)
display(plt.show())
evr = pca_test.explained_variance_ratio_
cvr = np.cumsum(pca_test.explained_variance_ratio_)
pca_df = pd.DataFrame()
pca_df['Cumulative Variance Ratio'] = cvr
pca_df['Explained Variance Ratio'] = evr
display(pca_df.head(10))
在使用的组件数超过10后,组件数的增加不会大大增加解释的方差该数据框包含诸如累积方差比(数据的解释方差的累积大小)和解释方差比(每个成分对解释方差的总体积的贡献)之类的指标如果您查看上述数据框,就会发现使用PCA从30个变量转变为10个变量组件允许解释95%的数据分散。其他20个组件所占比例不到5%,这意味着我们可以拒绝它们。按照这一逻辑,我们用PCA从30减少元件数量,以10X_train和X_test。我们在中X_train_scaled_pca和中写入这些人为创建的“降维”数据集X_test_scaled_pca。pca = PCA(n_components=10)
pca.fit(X_train_scaled)
X_train_scaled_pca = pca.transform(X_train_scaled)
X_test_scaled_pca = pca.transform(X_test_scaled)
每个组件都是源变量与相应“权重”的线性组合。通过创建数据框,我们可以看到每个组件的这些“权重”。pca_dims = []
for x in range(0, len(pca_df)):
pca_dims.append('PCA Component {}'.format(x))
pca_test_df = pd.DataFrame(pca_test.components_, columns=columns, index=pca_dims)
pca_test_df.head(10).T
组件信息数据框6.在对数据应用主成分法之后训练基本的RF模型(模型2,RF + PCA)
现在,我们可以继续传递另一个基本的RF模型数据X_train_scaled_pca,y_train并找出该模型发出的预测的准确性是否有所提高。rfc = RandomForestClassifier()
rfc.fit(X_train_scaled_pca, y_train)
display(rfc.score(X_train_scaled_pca, y_train))
型号比较如下。7.优化超参数。第一轮:RandomizedSearchCV
使用主成分方法处理数据后,可以尝试使用模型超参数的优化,以提高RF模型产生的预测的质量。可以将超参数视为模型的“设置”。最适合一个数据集的设置将不适用于另一个数据集-这就是为什么您需要对其进行优化。您可以从RandomizedSearchCV算法开始,该算法使您可以大致探索各种值。可以在此处找到有关RF模型的所有超参数的说明。在工作过程中,我们生成一个实体param_dist,其中包含每个超参数的一系列需要测试的值。接下来,我们初始化对象。rs使用函数RandomizedSearchCV(),将其传递给RF模型param_dist,迭代次数和需要执行的交叉验证次数。超参数verbose使您可以控制模型在其操作期间显示的信息量(例如在模型训练期间的信息输出)。超参数n_jobs允许您指定需要使用多少个处理器核心以确保模型正常工作。将其设置n_jobs为值-1将导致更快的模型,因为它将使用所有处理器内核。我们将从事以下超参数的选择:n_estimators -“随机森林”中“树”的数量。max_features -选择分割的特征数量。max_depth -树木的最大深度。min_samples_split -树节点分裂所需的最少对象数。min_samples_leaf -叶子中的最小对象数。bootstrap -用于构造带有返回的子样本树。
from sklearn.model_selection import RandomizedSearchCV
n_estimators = [int(x) for x in np.linspace(start = 100, stop = 1000, num = 10)]
max_features = ['log2', 'sqrt']
max_depth = [int(x) for x in np.linspace(start = 1, stop = 15, num = 15)]
min_samples_split = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
min_samples_leaf = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
bootstrap = [True, False]
param_dist = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
rs = RandomizedSearchCV(rfc_2,
param_dist,
n_iter = 100,
cv = 3,
verbose = 1,
n_jobs=-1,
random_state=0)
rs.fit(X_train_scaled_pca, y_train)
rs.best_params_
利用参数n_iter = 100和的值cv = 3,我们创建了300个RF模型,随机选择上述超级参数的组合。我们可以参考该属性best_params_ 以获取有关一组参数的信息,这些参数使您可以创建最佳模型。但是在现阶段,这可能无法为我们提供关于参数范围的最有趣的数据,这些数据值得在下一轮优化中探索。为了找出值得继续搜索的值范围,我们可以轻松地获得一个包含RandomizedSearchCV算法结果的数据框。rs_df = pd.DataFrame(rs.cv_results_).sort_values('rank_test_score').reset_index(drop=True)
rs_df = rs_df.drop([
'mean_fit_time',
'std_fit_time',
'mean_score_time',
'std_score_time',
'params',
'split0_test_score',
'split1_test_score',
'split2_test_score',
'std_test_score'],
axis=1)
rs_df.head(10)
RandomizedSearchCV算法的结果现在我们将创建条形图,在该条形图的X轴上是超参数值,Y轴上是模型显示的平均值。这将使您有可能了解平均而言,超参数的哪些值显示出最佳性能。fig, axs = plt.subplots(ncols=3, nrows=2)
sns.set(style="whitegrid", color_codes=True, font_scale = 2)
fig.set_size_inches(30,25)
sns.barplot(x='param_n_estimators', y='mean_test_score', data=rs_df, ax=axs[0,0], color='lightgrey')
axs[0,0].set_ylim([.83,.93])axs[0,0].set_title(label = 'n_estimators', size=30, weight='bold')
sns.barplot(x='param_min_samples_split', y='mean_test_score', data=rs_df, ax=axs[0,1], color='coral')
axs[0,1].set_ylim([.85,.93])axs[0,1].set_title(label = 'min_samples_split', size=30, weight='bold')
sns.barplot(x='param_min_samples_leaf', y='mean_test_score', data=rs_df, ax=axs[0,2], color='lightgreen')
axs[0,2].set_ylim([.80,.93])axs[0,2].set_title(label = 'min_samples_leaf', size=30, weight='bold')
sns.barplot(x='param_max_features', y='mean_test_score', data=rs_df, ax=axs[1,0], color='wheat')
axs[1,0].set_ylim([.88,.92])axs[1,0].set_title(label = 'max_features', size=30, weight='bold')
sns.barplot(x='param_max_depth', y='mean_test_score', data=rs_df, ax=axs[1,1], color='lightpink')
axs[1,1].set_ylim([.80,.93])axs[1,1].set_title(label = 'max_depth', size=30, weight='bold')
sns.barplot(x='param_bootstrap',y='mean_test_score', data=rs_df, ax=axs[1,2], color='skyblue')
axs[1,2].set_ylim([.88,.92])
axs[1,2].set_title(label = 'bootstrap', size=30, weight='bold')
plt.show()
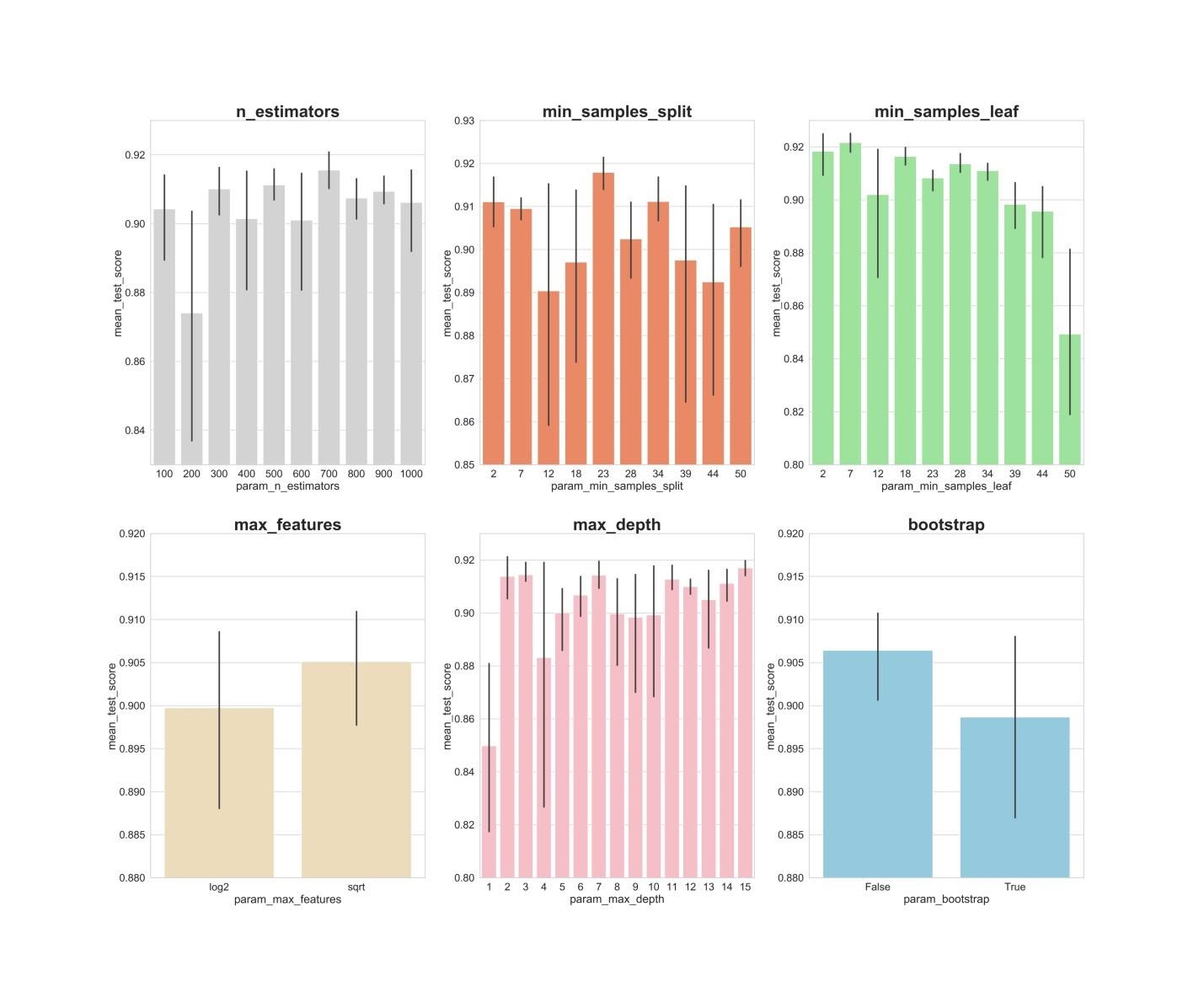
n_estimators:显然,300、500、700的值显示出最佳的平均结果。min_samples_split:像2和7这样的小值似乎显示出最好的结果。值23看起来也不错。您可以检查此超参数超过2的几个值,以及约23的几个值。min_samples_leaf:感觉这个超参数的较小值会给出更好的结果。这意味着我们可以体验2到7之间的值。max_features:选项sqrt给出最高的平均结果。max_depth:超参数的值与模型的结果之间没有明确的关系,但是有一种感觉,值2、3、7、11、15看起来不错。bootstrap:该值False显示最佳的平均结果。
现在,使用这些发现,我们可以继续进行第二轮超参数优化。这将缩小我们感兴趣的价值范围。8.优化超参数。第2轮:GridSearchCV(模型3的参数的最终准备,RF + PCA + HT)
应用RandomizedSearchCV算法后,我们将使用GridSearchCV算法对超参数的最佳组合进行更精确的搜索。这里研究了相同的超参数,但现在我们正在对其进行更好的“彻底”搜索。使用GridSearchCV算法,检查超参数的每种组合。当我们独立设置搜索迭代次数时,这比使用RandomizedSearchCV算法需要更多的计算资源。例如,为3个块中的交叉验证为6个超参数中的每一个研究10个值将需要10 x 3或300万个模型训练课程。这就是为什么我们在应用RandomizedSearchCV之后缩小研究参数的取值范围之后使用GridSearchCV算法的原因。因此,使用我们在RandomizedSearchCV的帮助下发现的内容,我们检查了表现出最佳状态的超参数的值:from sklearn.model_selection import GridSearchCV
n_estimators = [300,500,700]
max_features = ['sqrt']
max_depth = [2,3,7,11,15]
min_samples_split = [2,3,4,22,23,24]
min_samples_leaf = [2,3,4,5,6,7]
bootstrap = [False]
param_grid = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
gs = GridSearchCV(rfc_2, param_grid, cv = 3, verbose = 1, n_jobs=-1)
gs.fit(X_train_scaled_pca, y_train)
rfc_3 = gs.best_estimator_
gs.best_params_
在这里,我们针对540(3 x 1 x 5 x 6 x 6 x 1)个模型训练课程在3个块中应用交叉验证,这提供了1620个模型训练课程。现在,在我们使用RandomizedSearchCV和GridSearchCV之后,我们可以转到属性best_params_以找出哪些超参数值可以使模型与正在研究的数据集最佳配合使用(这些值可以在上一个代码块的底部看到) 。这些参数用于创建型号3。9.根据验证数据评估模型的质量
现在,您可以根据验证数据评估创建的模型。即,我们正在谈论材料开始时描述的这三个模型。查看以下模型:y_pred = rfc.predict(X_test_scaled)
y_pred_pca = rfc.predict(X_test_scaled_pca)
y_pred_gs = gs.best_estimator_.predict(X_test_scaled_pca)
为模型创建误差矩阵,并找出每个模型对乳腺癌的预测能力:from sklearn.metrics import confusion_matrix
conf_matrix_baseline = pd.DataFrame(confusion_matrix(y_test, y_pred), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_baseline_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_pca), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_tuned_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_gs), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
display(conf_matrix_baseline)
display('Baseline Random Forest recall score', recall_score(y_test, y_pred))
display(conf_matrix_baseline_pca)
display('Baseline Random Forest With PCA recall score', recall_score(y_test, y_pred_pca))
display(conf_matrix_tuned_pca)
display('Hyperparameter Tuned Random Forest With PCA Reduced Dimensionality recall score', recall_score(y_test, y_pred_gs))
三种模型的工作结果在此评估度量“完整性”(召回)。事实是我们正在诊断癌症。因此,我们对最小化模型发布的假阴性预测非常感兴趣。鉴于此,我们可以得出结论,基本RF模型给出了最佳结果。完整率达94.97%。在测试数据集中,有179名癌症患者的记录。该模型找到了其中的170个。摘要
这项研究提供了重要的观察。有时,使用主成分方法和超参数的大规模优化的RF模型可能无法像标准设置的最普通模型那样工作。但这不是将自己仅限于最简单的模型的原因。如果不尝试不同的模型,就不可能说哪个模型将显示最佳结果。在用于预测患者癌症存在的模型的情况下,我们可以说模型越好-可以挽救更多的生命。亲爱的读者们!您使用机器学习方法解决了哪些任务?