Olá Habr! Hoje, quero compartilhar um pequeno exemplo de como conduzir a análise de cluster. Neste exemplo, o leitor não encontrará redes neurais e outras direções da moda. Este exemplo pode servir como um ponto de referência para fazer uma análise de cluster pequena e completa para outros dados. Qualquer pessoa interessada - bem-vindo ao gato.
Faça uma reserva imediatamente, este artigo de forma alguma afirma ser acadêmico em sua totalidade, exclusividade dos resultados obtidos ou abrangência da cobertura do problema. O artigo pretende demonstrar as etapas básicas da análise clássica de agrupamentos, que podem ser usadas para um estudo simples e significativo (possivelmente precedendo um estudo mais detalhado). Quaisquer correções, comentários e acréscimos de mérito são bem-vindos.
Os dados são uma amostra do consumo de álcool por país per capita, por tipo de bebidas alcoólicas (cerveja, vinho, bebidas espirituosas, etc.) para 2010 como uma porcentagem do consumo per capita de álcool. Os dados também contêm: consumo médio diário de álcool per capita em gramas de álcool puro e todo o consumo de álcool (registrado + não contabilizado) per capita (apenas bebedores em litros de álcool puro).
Ao mesmo tempo, cada país pertence condicionalmente a um dos grupos geográficos: leste, centro e oeste. A divisão é muito arbitrária e muito controversa por várias razões, mas prosseguiremos com o que temos. Fonte de dados - Relatório de status global sobre álcool e saúde 2014, S. 289-364
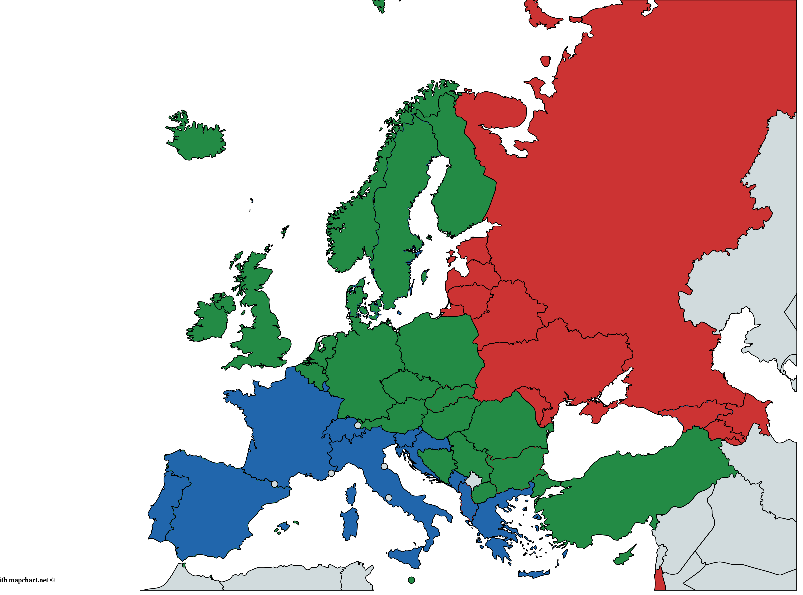
(Pintado à mão, pode haver erros, mas a idéia geral, eu acho, é compreensível)
Análise preliminar
Conecte as bibliotecas usadas.
library(rgl)
library(heplots)
library(MVN)
library(klaR)
library('Morpho')
library(caret)
library(mclust)
library(ggplot2)
library(GGally)
library(plyr)
library(psych)
library(GPArotation)
library(ggpubr)
, .
#
data <- read.table("alcohol_data.csv", header=TRUE, sep=",")
#
rownames(data) <- make.names(data[,1], unique = TRUE)
# ,
data <- data[,-1]
data <- na.omit(data)
#
head(data)
summary(data)
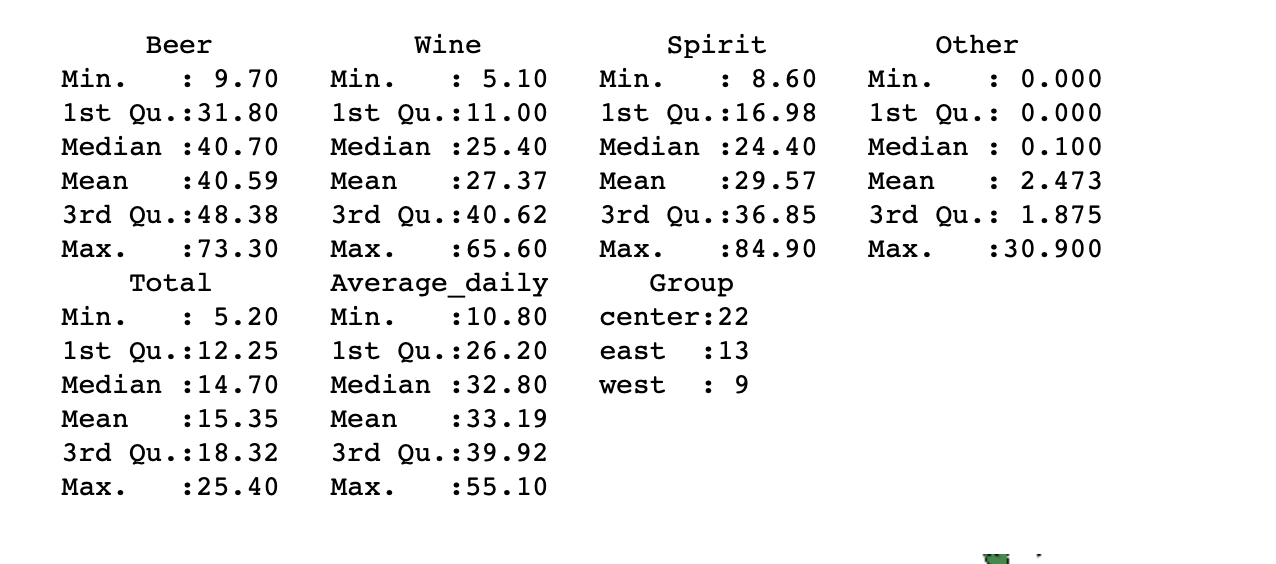
, . , Other , , , , . , , , , . , . - .
, , , .
options(rgl.useNULL=TRUE)
open3d()
mfrow3d(2,2)
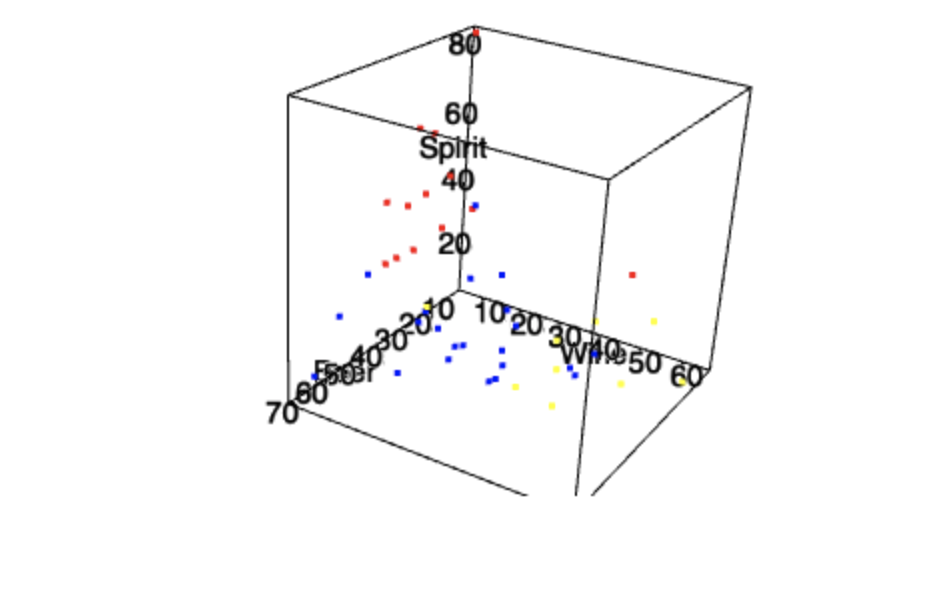
levelColors <- c('west'='blue', 'east'='red', 'center'='yellow')
plot3d(data$Beer, data$Wine, data$Spirit, xlab="Beer", ylab="Wine", zlab="Spirit", col = levelColors[data$Group], size=3)
widget <- rglwidget()
widget
, . , .
ggpairs(
data,
mapping = ggplot2::aes(color = data$Group),
upper = list(continuous = wrap("cor", alpha = 0.5), combo = "box"),
lower = list(continuous = wrap("points", alpha = 0.3), combo = wrap("dot", alpha = 0.4)),
diag = list(continuous = wrap("densityDiag",alpha = 0.5)),
title = "Alcohol"
)
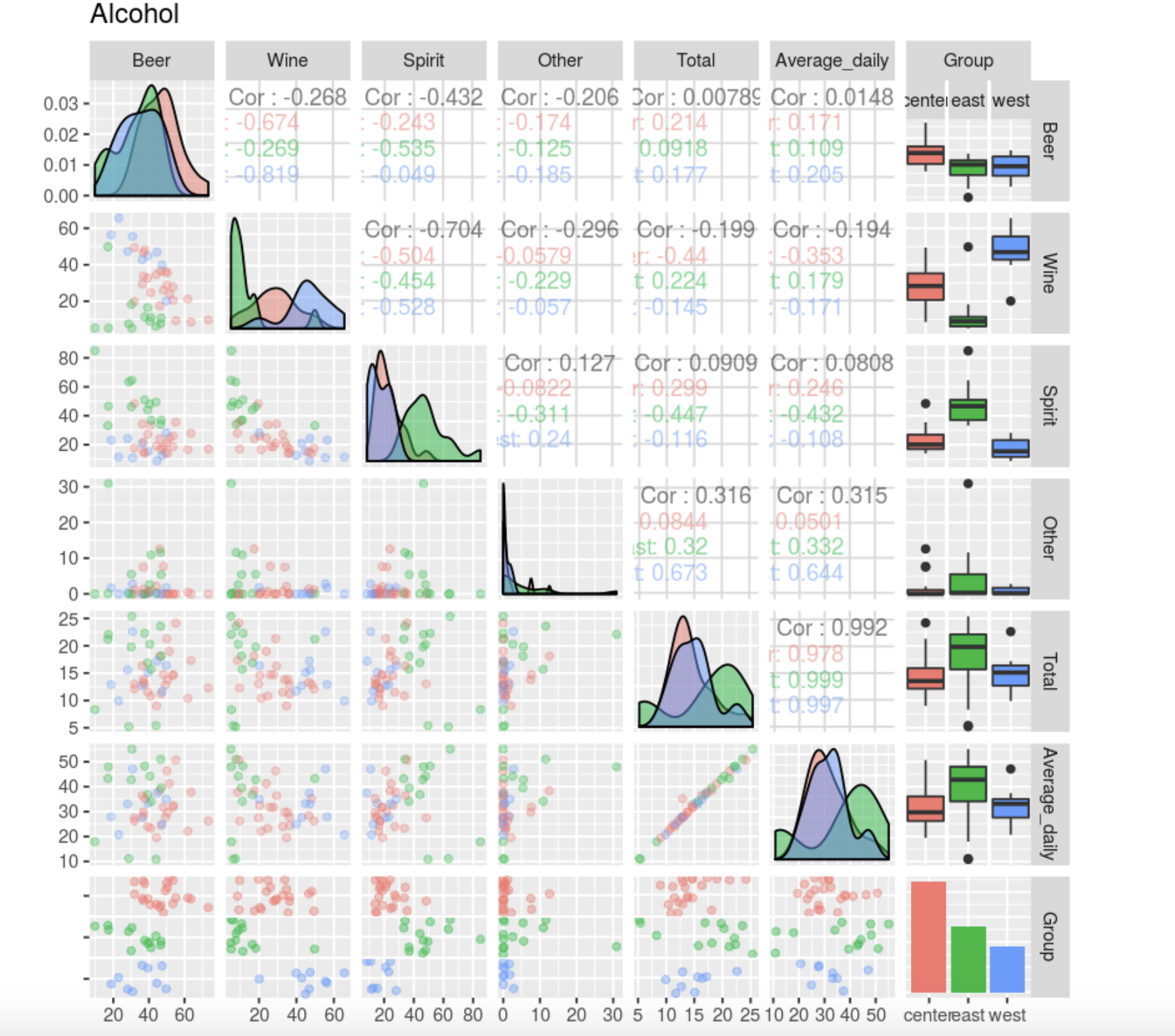
Average Total , Average.
data <- data[, -6]
, , , , . .
data[data$Wine>60,]
, , , , - , , .
data[data$Spirit>70,]
data[data$Spirit<10,]
, , .
,
split(data[,1:5],data$Group)
$center
$east
$west
ggpairs(
data,
mapping = ggplot2::aes(color = data$Group),
diag=list(continuous="bar", alpha=0.4)
)
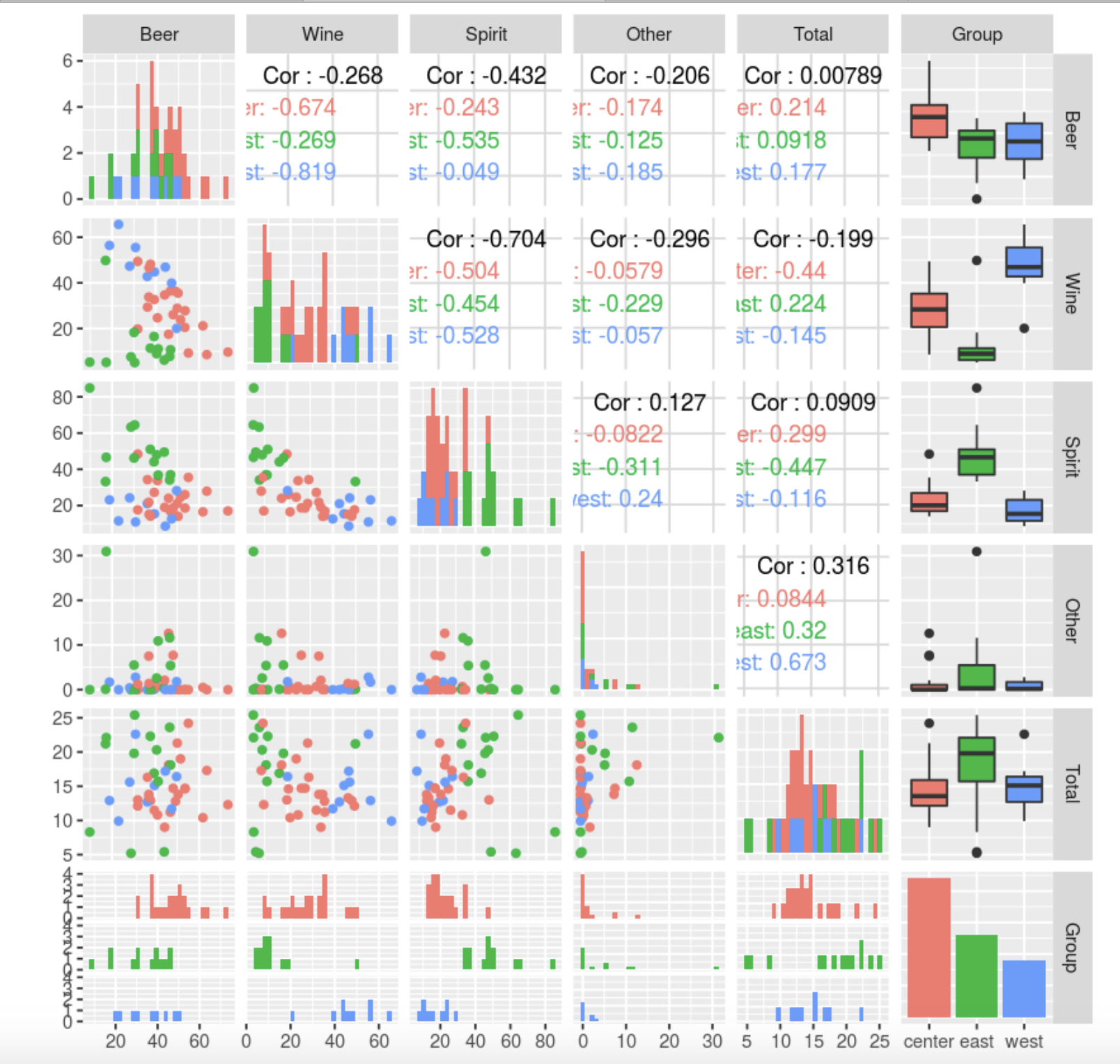
, , . Other, : , , , ( 10-12 , 45, , ). . , , , (). , , . Other .
, , — , — . , — , .
Total Other, . .
, Beer, Spirit Wine . , , , . , , , , , .
Total. , — .
data.group = data[,5]
data <- data[,-5]
data<- data[,-4]
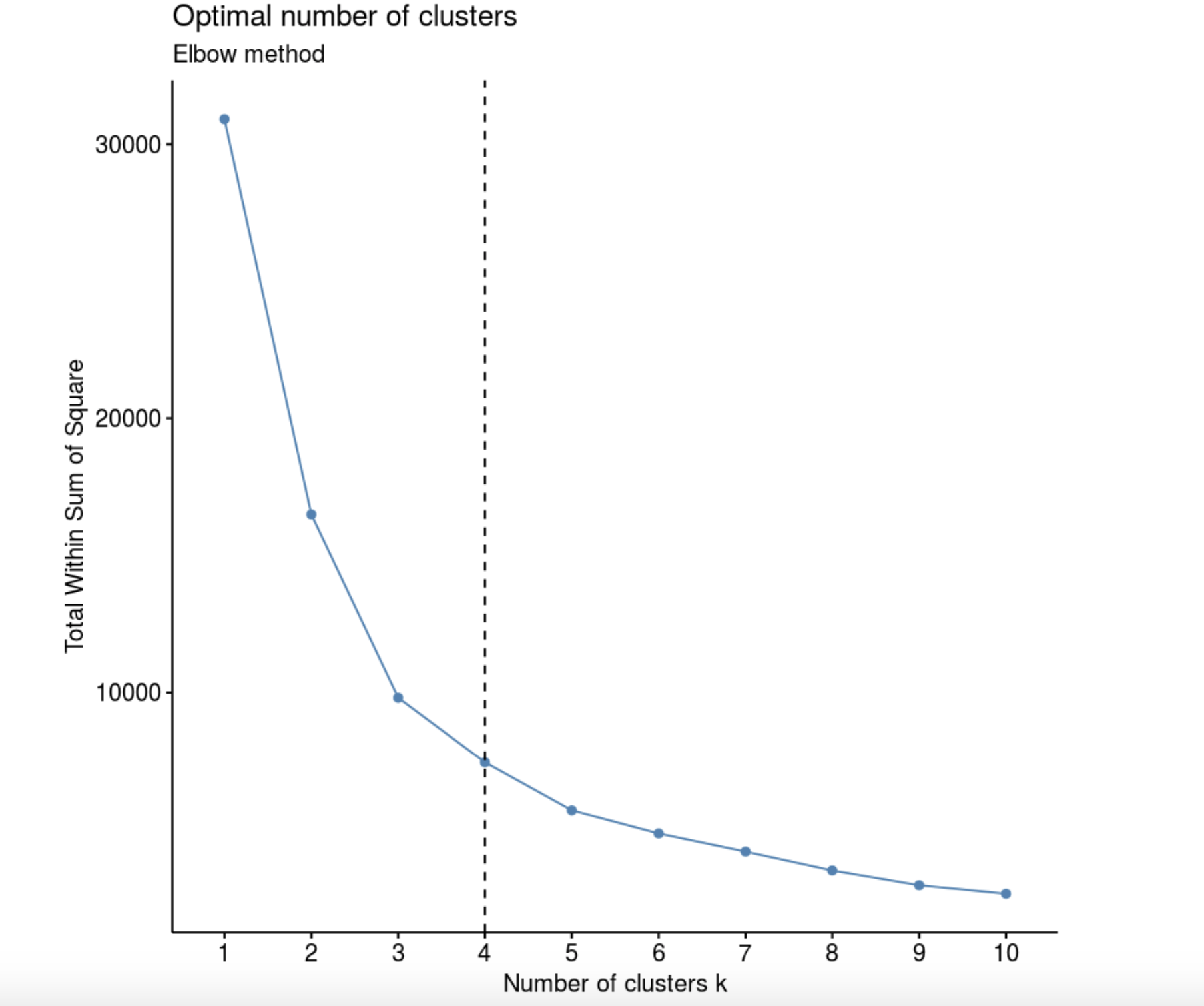
Elbow method (“ ”, “ ”). , k, – W(K), .
library(factoextra)
fviz_nbclust(data, kmeans, method = "wss") +
labs(subtitle = "Elbow method") +
geom_vline(xintercept = 4, linetype = 2)
data.dist <- dist((data))
hc <- hclust(data.dist, method = "ward.D2")
plot(hc, cex = 0.7)
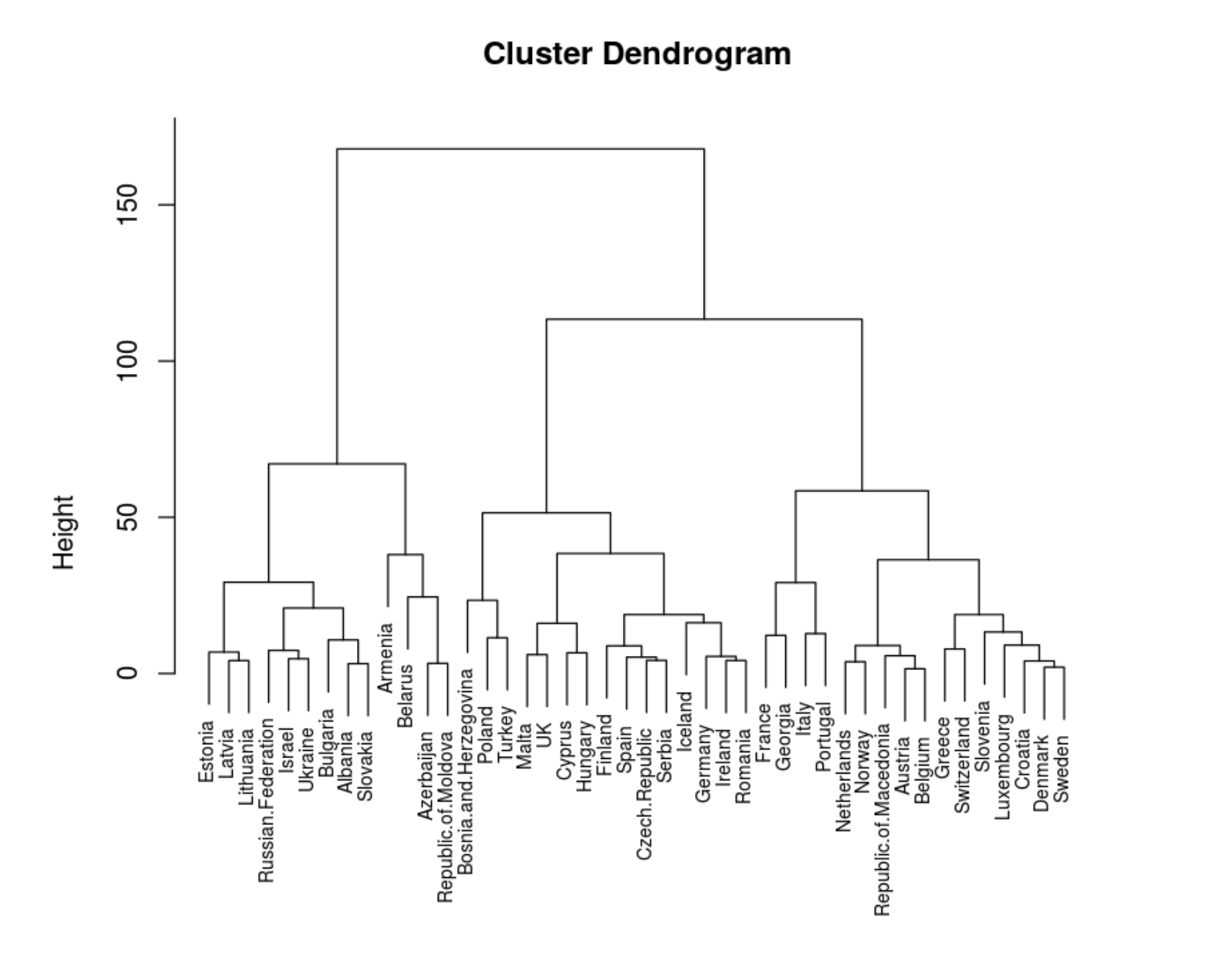
. .
colors=c('green', 'red', 'blue')
hcd = as.dendrogram(hc)
clusMember = cutree(hc, 4)
colLab <- function(n) {
if (is.leaf(n)) {
a <- attributes(n)
labCol <- colors[data.group[n]]
attr(n, "nodePar") <- c(a$nodePar, lab.col = labCol)
}
n
}
clusDendro = dendrapply(hcd, colLab)
plot(clusDendro, main = "Cool Dendrogram", type = "triangle")
rect.hclust(hc, k = 4)
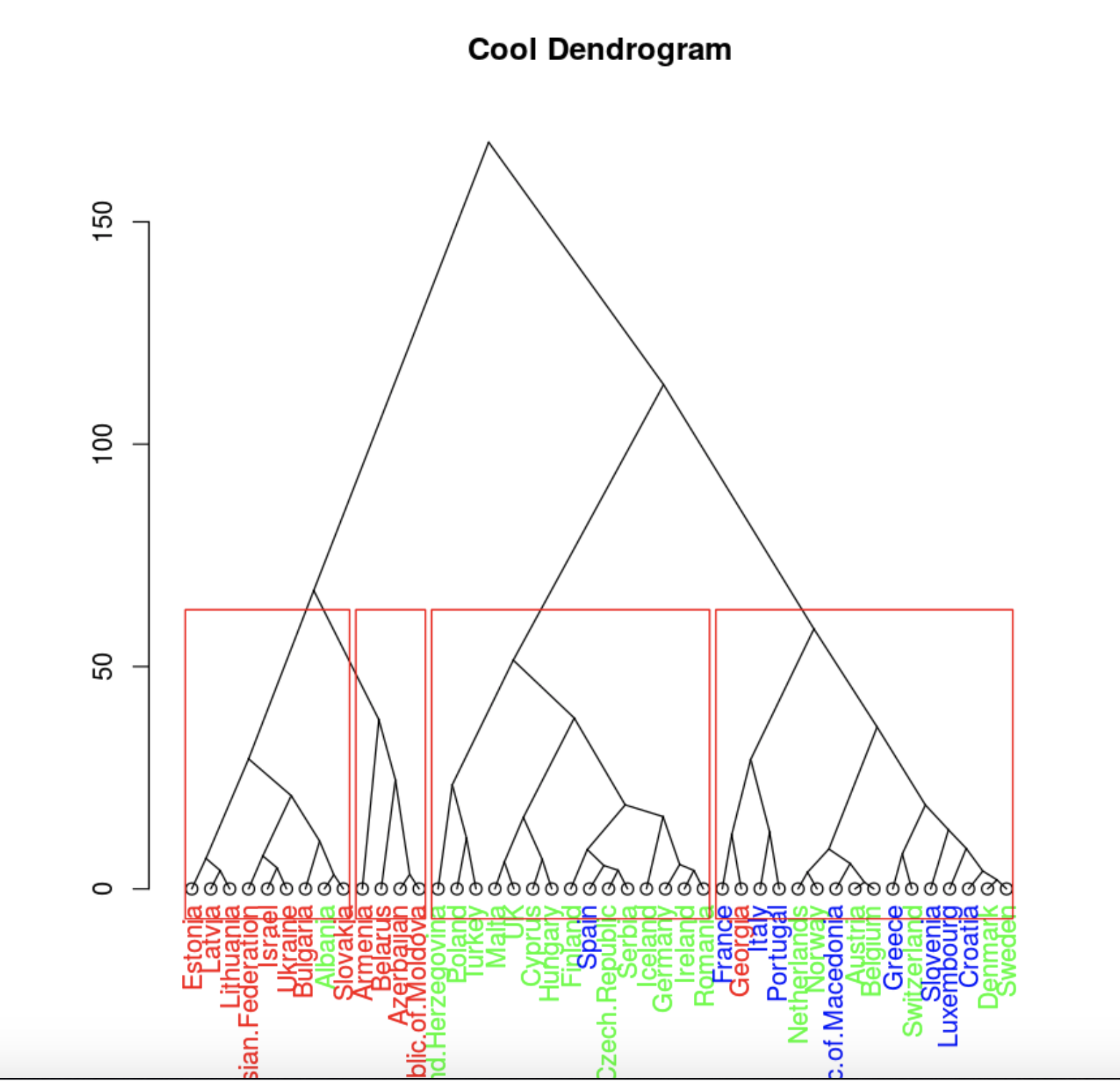
. , .
, , , 4 .
plot(clusDendro, main = "Cool Dendrogram", type = "triangle")
data.hclas_group <- factor(cutree(hc, k = 3))
rect.hclust(hc, k = 3)
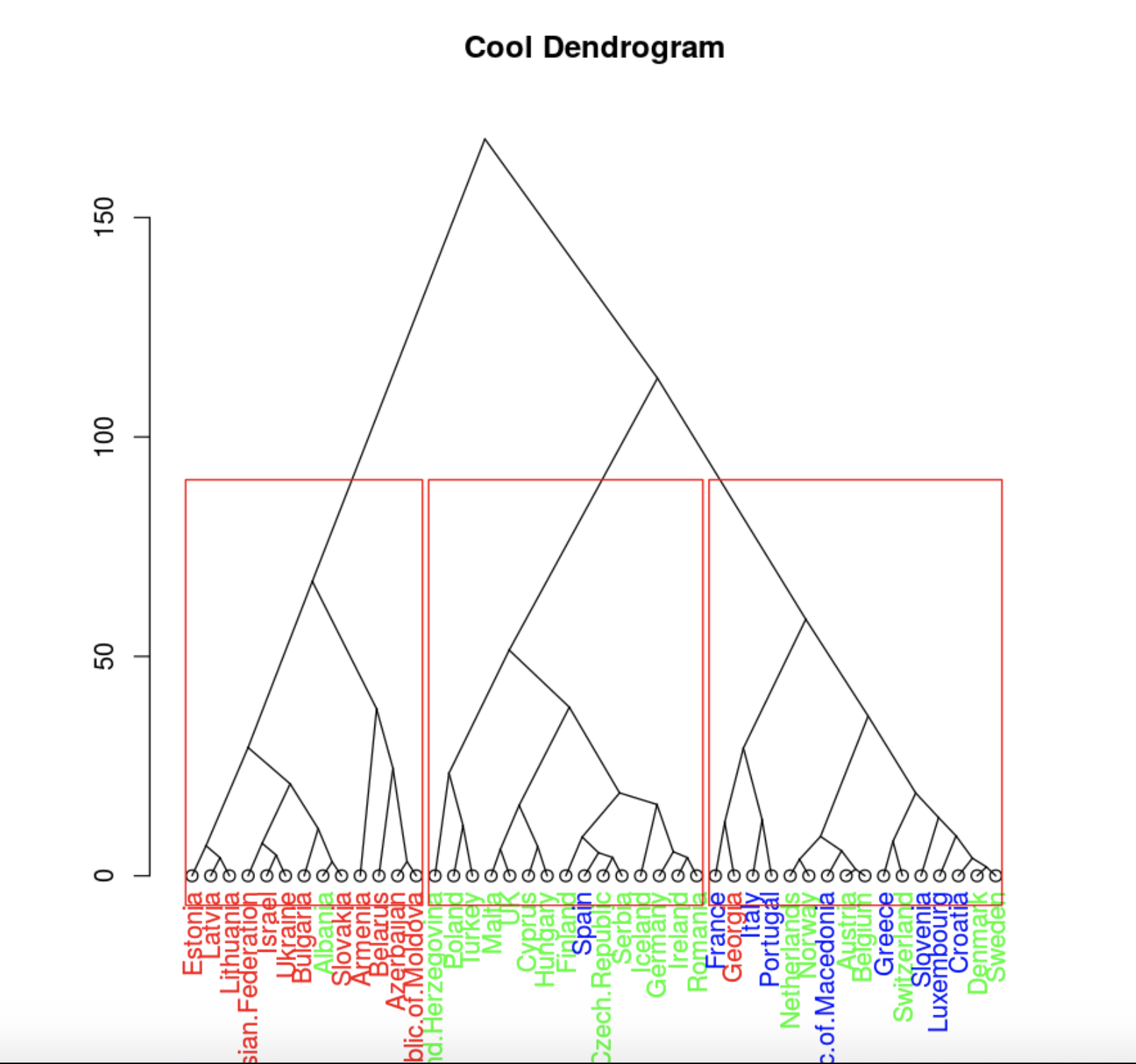
, , .
library(FactoMineR)
res.pca <- PCA(data,scale.unit=T, graph = F)
fviz_pca_biplot(res.pca,
col = colors[data.hclas_group], palette = "jco",
label = "var",
ellipse.level = 0.8,
addEllipses = T,
col.var = "black",
legend.title = "groups4")
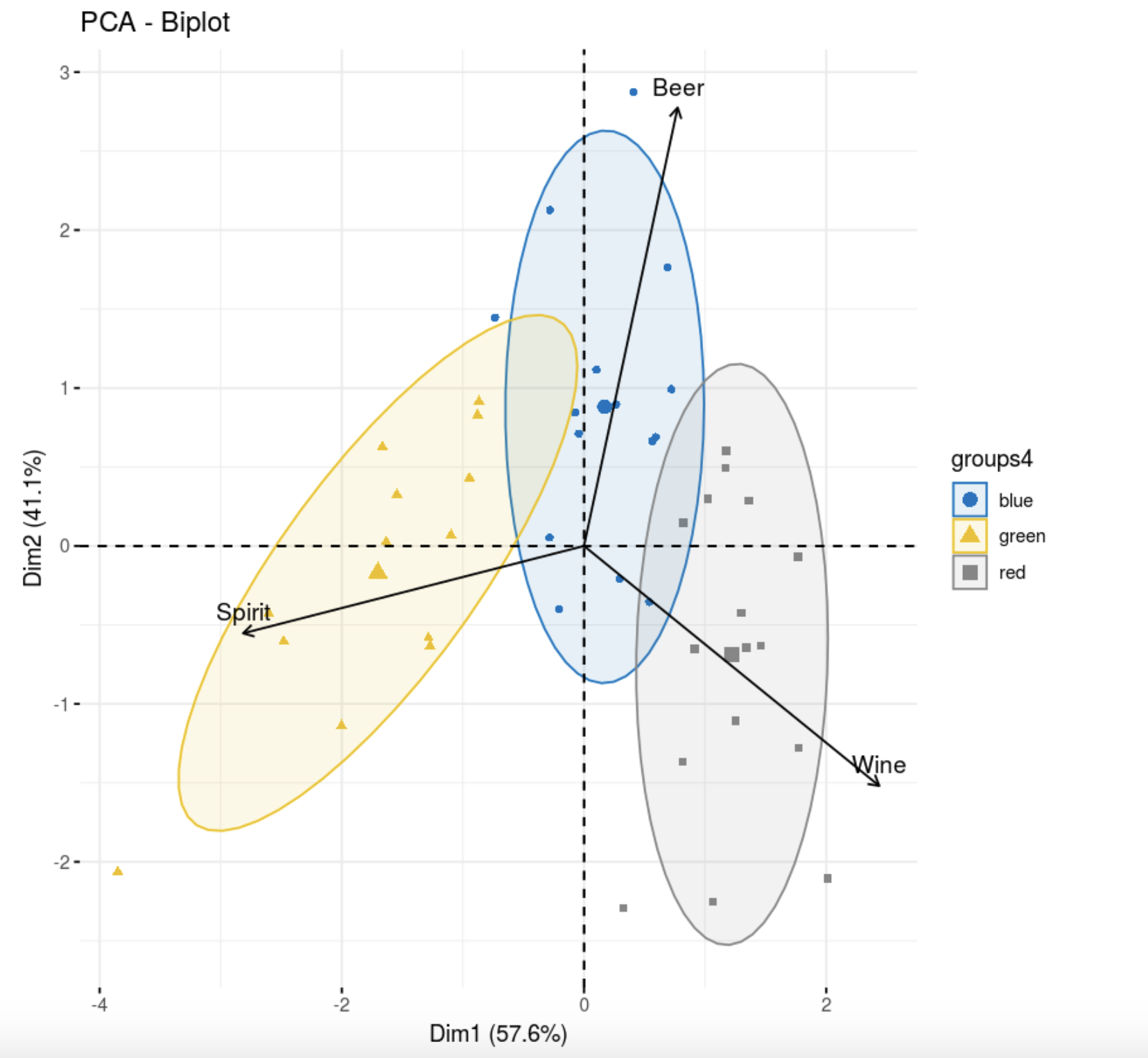
, , . , , , , . , , , k-++.
library(flexclust)
data.kk <- kcca(data, k=3, family=kccaFamily("kmeans"),
control=list(initcent="kmeanspp"))
fviz_pca_biplot(res.pca,
col.ind =as.factor(data.kk@cluster), palette = "jco",
label = "var",
ellipse.level = 0.8,
addEllipses = T,
col.var = "black", repel = TRUE,
legend.title = "clusters")
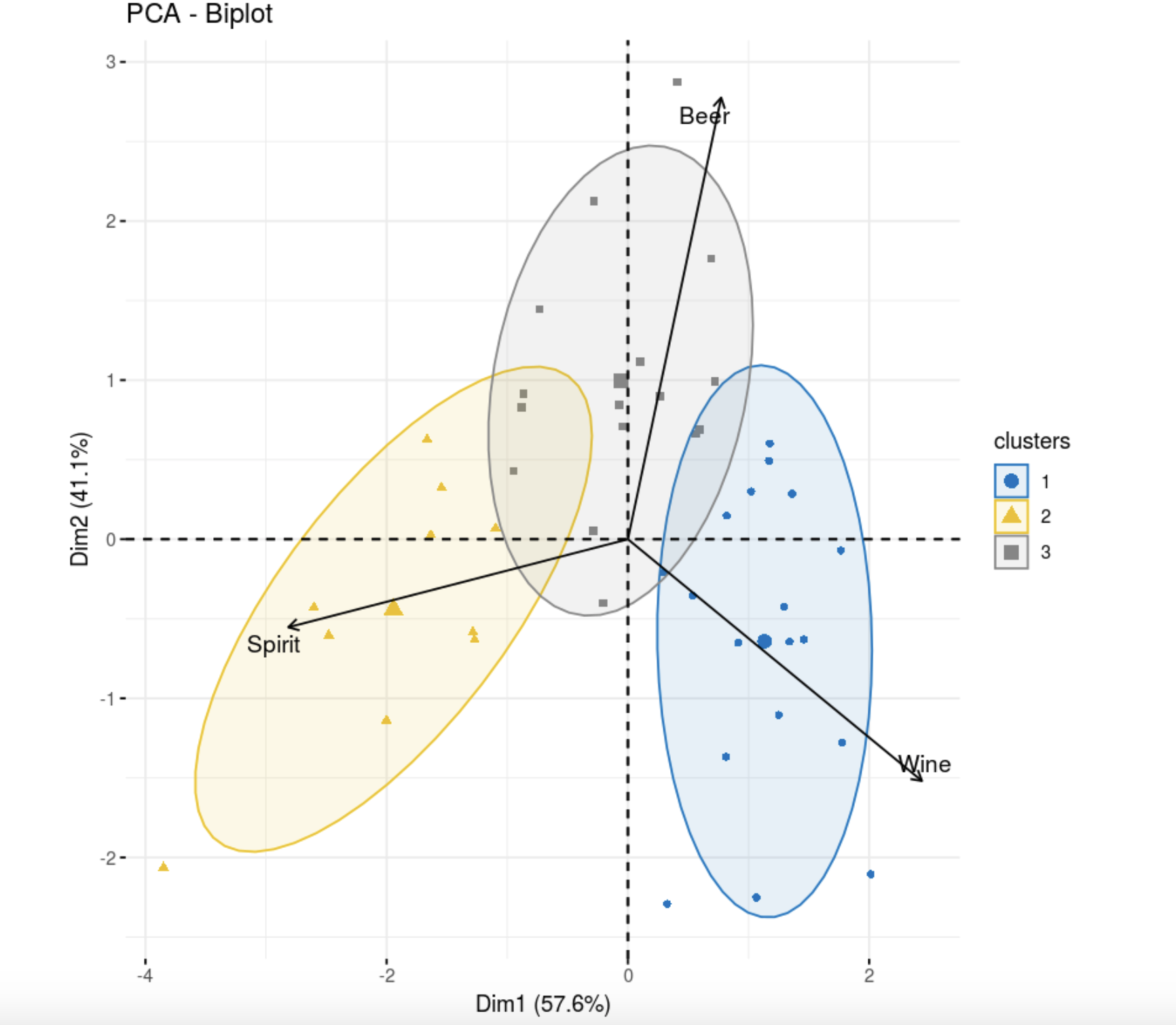
, k- . , , .
, , hclust. .

, , . . , .
. . , , , . , , . , .
Seria possível realizar o clustering com base na suposição de modelos de cluster usando critérios de informação ( aqui está a descrição ) e também tentar a análise discriminante clássica para esse conjunto de dados. Se este artigo foi útil, pretendo publicar uma sequência.