Os especialistas em processamento e análise de dados têm muitas ferramentas para criar modelos de classificação. Um dos métodos mais populares e confiáveis para desenvolver esses modelos é usar o algoritmo Random Forest (RF). Para tentar melhorar o desempenho de um modelo construído usando o algoritmo de RF , você pode usar a otimização do hiperparâmetro do modelo ( Hyperparameter Tuning , HT). Além disso, existe uma abordagem ampla segundo a qual os dados, antes de serem transferidos para o modelo, são processados usando a Análise de componentes principais , PCA). Mas vale a pena usar? O principal objetivo do algoritmo de RF não é ajudar o analista a interpretar a importância das características?Sim, o uso do algoritmo PCA pode levar a uma ligeira complicação da interpretação de cada "recurso" na análise da "importância dos recursos" do modelo de RF. No entanto, o algoritmo PCA reduz a dimensão do espaço de recurso, o que pode levar a uma diminuição no número de recursos que precisam ser processados pelo modelo de RF. Observe que o volume de cálculos é uma das principais desvantagens do algoritmo de floresta aleatória (ou seja, pode levar muito tempo para concluir o modelo). A aplicação do algoritmo PCA pode ser uma parte muito importante da modelagem, especialmente nos casos em que eles trabalham com centenas ou mesmo milhares de recursos. Como resultado, se o mais importante é simplesmente criar o modelo mais eficaz e, ao mesmo tempo, você pode sacrificar a precisão de determinar a importância dos atributos, então vale a pena tentar o PCA.Agora ao ponto. Trabalharemos com um conjunto de dados de câncer de mama - Scikit-learn "câncer de mama" . Criaremos três modelos e compararemos sua eficácia. Ou seja, estamos falando dos seguintes modelos:
, PCA). Mas vale a pena usar? O principal objetivo do algoritmo de RF não é ajudar o analista a interpretar a importância das características?Sim, o uso do algoritmo PCA pode levar a uma ligeira complicação da interpretação de cada "recurso" na análise da "importância dos recursos" do modelo de RF. No entanto, o algoritmo PCA reduz a dimensão do espaço de recurso, o que pode levar a uma diminuição no número de recursos que precisam ser processados pelo modelo de RF. Observe que o volume de cálculos é uma das principais desvantagens do algoritmo de floresta aleatória (ou seja, pode levar muito tempo para concluir o modelo). A aplicação do algoritmo PCA pode ser uma parte muito importante da modelagem, especialmente nos casos em que eles trabalham com centenas ou mesmo milhares de recursos. Como resultado, se o mais importante é simplesmente criar o modelo mais eficaz e, ao mesmo tempo, você pode sacrificar a precisão de determinar a importância dos atributos, então vale a pena tentar o PCA.Agora ao ponto. Trabalharemos com um conjunto de dados de câncer de mama - Scikit-learn "câncer de mama" . Criaremos três modelos e compararemos sua eficácia. Ou seja, estamos falando dos seguintes modelos:- O modelo básico baseado no algoritmo de RF (vamos abreviar esse modelo de RF).
- O mesmo modelo que o nº 1, mas no qual uma redução na dimensão do espaço de feição é aplicada usando o método do componente principal (RF + PCA).
- O mesmo modelo que o nº 2, mas construído usando a otimização de hiperparâmetros (RF + PCA + HT).
1. Importar dados
Para começar, carregue os dados e crie um quadro de dados do Pandas. Como usamos um conjunto de dados "de brinquedo" pré-limpo do Scikit-learn, depois disso já podemos iniciar o processo de modelagem. Porém, mesmo ao usar esses dados, é recomendável que você sempre comece a trabalhar realizando uma análise preliminar dos dados usando os seguintes comandos aplicados ao quadro de dados ( df):df.head() - para dar uma olhada no novo quadro de dados e ver se parece com o esperado.df.info()- para descobrir os recursos dos tipos de dados e do conteúdo da coluna. Pode ser necessário executar a conversão do tipo de dados antes de continuar.df.isna()- para garantir que não haja valores nos dados NaN. Os valores correspondentes, se houver, podem precisar ser processados de alguma forma ou, se necessário, pode ser necessário remover linhas inteiras do quadro de dados.df.describe() - descobrir os valores mínimos, máximos e médios dos indicadores nas colunas, descobrir os indicadores do quadrado médio e desvio provável nas colunas.
Em nosso conjunto de dados, uma coluna cancer(câncer) é a variável de destino cujo valor queremos prever usando o modelo. 0significa "sem doença". 1- "a presença da doença".import pandas as pd
from sklearn.datasets import load_breast_cancer
columns = ['mean radius', 'mean texture', 'mean perimeter', 'mean area', 'mean smoothness', 'mean compactness', 'mean concavity', 'mean concave points', 'mean symmetry', 'mean fractal dimension', 'radius error', 'texture error', 'perimeter error', 'area error', 'smoothness error', 'compactness error', 'concavity error', 'concave points error', 'symmetry error', 'fractal dimension error', 'worst radius', 'worst texture', 'worst perimeter', 'worst area', 'worst smoothness', 'worst compactness', 'worst concavity', 'worst concave points', 'worst symmetry', 'worst fractal dimension']
dataset = load_breast_cancer()
data = pd.DataFrame(dataset['data'], columns=columns)
data['cancer'] = dataset['target']
display(data.head())
display(data.info())
display(data.isna().sum())
display(data.describe())
. . , cancer, , . 0 « ». 1 — « »2.
Agora divida os dados usando a função Scikit-learn train_test_split. Queremos fornecer ao modelo o máximo de dados de treinamento possível. No entanto, precisamos ter dados suficientes à disposição para testar o modelo. Em geral, podemos dizer que, à medida que o número de linhas no conjunto de dados aumenta, também aumenta a quantidade de dados que podem ser considerados educacionais.Por exemplo, se houver milhões de linhas, você pode dividir o conjunto destacando 90% das linhas para dados de treinamento e 10% para dados de teste. Mas o conjunto de dados de teste contém apenas 569 linhas. E isso não é muito para treinar e testar o modelo. Como resultado, para sermos justos em relação aos dados de treinamento e verificação, dividiremos o conjunto em duas partes iguais - 50% - dados de treinamento e 50% - dados de verificação. Nós instalamosstratify=y para garantir que os conjuntos de dados de treinamento e teste tenham a mesma proporção de 0 e 1 que o conjunto de dados original.from sklearn.model_selection import train_test_split
X = data.drop('cancer', axis=1)
y = data['cancer']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.50, random_state = 2020, stratify=y)
3. Escala de dados
Antes de prosseguir com a modelagem, você precisa “centralizar” e “padronizar” os dados, escalando-os . A escala é realizada devido ao fato de que quantidades diferentes são expressas em unidades diferentes. Este procedimento permite que você organize uma "luta justa" entre os sinais para determinar sua importância. Além disso, convertemos y_traindo tipo de dados Pandas Seriespara o array NumPy para que mais tarde o modelo possa trabalhar com os destinos correspondentes.import numpy as np
from sklearn.preprocessing import StandardScaler
ss = StandardScaler()
X_train_scaled = ss.fit_transform(X_train)
X_test_scaled = ss.transform(X_test)
y_train = np.array(y_train)
4. Treinamento do modelo básico (modelo nº 1, RF)
Agora crie o número do modelo 1. Nele, lembramos que apenas o algoritmo Random Forest é usado. Ele usa todos os recursos e é configurado com os valores padrão (detalhes sobre essas configurações podem ser encontrados na documentação para sklearn.ensemble.RandomForestClassifier ). Inicialize o modelo. Depois disso, vamos treiná-la em dados dimensionados. A precisão do modelo pode ser medida nos dados de treinamento:from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import recall_score
rfc = RandomForestClassifier()
rfc.fit(X_train_scaled, y_train)
display(rfc.score(X_train_scaled, y_train))
Se estivermos interessados em aprender quais recursos são os mais importantes para o modelo de RF na previsão do câncer de mama, podemos visualizar e quantificar os indicadores da importância dos sinais consultando o atributo feature_importances_:feats = {}
for feature, importance in zip(data.columns, rfc_1.feature_importances_):
feats[feature] = importance
importances = pd.DataFrame.from_dict(feats, orient='index').rename(columns={0: 'Gini-Importance'})
importances = importances.sort_values(by='Gini-Importance', ascending=False)
importances = importances.reset_index()
importances = importances.rename(columns={'index': 'Features'})
sns.set(font_scale = 5)
sns.set(style="whitegrid", color_codes=True, font_scale = 1.7)
fig, ax = plt.subplots()
fig.set_size_inches(30,15)
sns.barplot(x=importances['Gini-Importance'], y=importances['Features'], data=importances, color='skyblue')
plt.xlabel('Importance', fontsize=25, weight = 'bold')
plt.ylabel('Features', fontsize=25, weight = 'bold')
plt.title('Feature Importance', fontsize=25, weight = 'bold')
display(plt.show())
display(importances)
Visualização da "importância" dos signosIndicadores de significância5. O método dos componentes principais
Agora vamos perguntar como podemos melhorar o modelo básico de RF. Utilizando a técnica de reduzir a dimensão do espaço de recurso, é possível apresentar o conjunto de dados inicial por meio de menos variáveis e, ao mesmo tempo, reduzir a quantidade de recursos de computação necessários para garantir a operação do modelo. Usando o PCA, você pode estudar a variação cumulativa da amostra desses recursos para entender quais recursos explicam a maior parte da variação nos dados.Inicializamos o objeto PCA ( pca_test), indicando o número de componentes (recursos) que precisam ser considerados. Definimos esse indicador como 30 para ver a variação explicada de todos os componentes gerados antes de decidir quantos componentes precisamos. Depois transferimos para os pca_testdados escaladosX_trainusando o método pca_test.fit(). Depois disso, visualizamos os dados.import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.decomposition import PCA
pca_test = PCA(n_components=30)
pca_test.fit(X_train_scaled)
sns.set(style='whitegrid')
plt.plot(np.cumsum(pca_test.explained_variance_ratio_))
plt.xlabel('number of components')
plt.ylabel('cumulative explained variance')
plt.axvline(linewidth=4, color='r', linestyle = '--', x=10, ymin=0, ymax=1)
display(plt.show())
evr = pca_test.explained_variance_ratio_
cvr = np.cumsum(pca_test.explained_variance_ratio_)
pca_df = pd.DataFrame()
pca_df['Cumulative Variance Ratio'] = cvr
pca_df['Explained Variance Ratio'] = evr
display(pca_df.head(10))
Depois que o número de componentes usados excede 10, o aumento no número não aumenta muito a variação explicadaEste quadro de dados contém indicadores como o Índice Acumulado Variação (tamanho cumulativo da variância explicada dos dados) e Relação de variância explicada (contribuição de cada componente para o volume total da variância explicada)Se você olhar para o quadro de dados acima, verifica-se que o uso do PCA para se deslocar de 30 variáveis para 10 aos componentes permite explicar 95% da dispersão dos dados. Os outros 20 componentes representam menos de 5% da variação, o que significa que podemos recusá-los. Seguindo essa lógica, usamos o PCA para reduzir o número de componentes de 30 para 10 paraX_traineX_test. Escrevemos esses conjuntos de dados de "dimensão reduzida" criados artificialmente dentroX_train_scaled_pcae dentroX_test_scaled_pca.pca = PCA(n_components=10)
pca.fit(X_train_scaled)
X_train_scaled_pca = pca.transform(X_train_scaled)
X_test_scaled_pca = pca.transform(X_test_scaled)
Cada componente é uma combinação linear de variáveis de origem com os "pesos" correspondentes. Podemos ver esses "pesos" para cada componente criando um quadro de dados.pca_dims = []
for x in range(0, len(pca_df)):
pca_dims.append('PCA Component {}'.format(x))
pca_test_df = pd.DataFrame(pca_test.components_, columns=columns, index=pca_dims)
pca_test_df.head(10).T
Informações sobre o componente Dataframe6. Treinar o modelo básico de RF após aplicar o método dos componentes principais aos dados (modelo nº 2, RF + PCA)
Agora podemos passar para outro dados básicos RF-modelo X_train_scaled_pcae y_traine pode descobrir sobre se há uma melhoria na precisão das previsões emitidas pelo modelo.rfc = RandomForestClassifier()
rfc.fit(X_train_scaled_pca, y_train)
display(rfc.score(X_train_scaled_pca, y_train))
Os modelos são comparados abaixo.7. Otimização de hiperparâmetros. Rodada 1: RandomizedSearchCV
Depois de processar os dados usando o método do componente principal, você pode tentar usar a otimização dos hiperparâmetros do modelo para melhorar a qualidade das previsões produzidas pelo modelo de RF. Os hiperparâmetros podem ser considerados como algo como "configurações" do modelo. As configurações perfeitas para um conjunto de dados não funcionarão para outro - é por isso que você precisa otimizá-las.Você pode começar com o algoritmo RandomizedSearchCV, que permite explorar bastante uma ampla gama de valores. Descrições de todos os hiperparâmetros para modelos de RF podem ser encontradas aqui .No decorrer do trabalho, geramos uma entidade param_distque contém, para cada hiperparâmetro, um intervalo de valores que precisam ser testados. Em seguida, inicializamos o objeto.rsusando a função RandomizedSearchCV(), passando o modelo de RF param_dist, o número de iterações e o número de validações cruzadas que precisam ser executadas.O hiperparâmetro verbosepermite controlar a quantidade de informações exibidas pelo modelo durante sua operação (como a saída de informações durante o treinamento do modelo). O hiperparâmetro n_jobspermite especificar quantos núcleos de processador você precisa usar para garantir a operação do modelo. Definir esse n_jobsvalor -1levará a um modelo mais rápido, pois isso usará todos os núcleos do processador.Estaremos envolvidos na seleção dos seguintes hiperparâmetros:n_estimators - o número de "árvores" na "floresta aleatória".max_features - o número de recursos para selecionar a divisão.max_depth - profundidade máxima das árvores.min_samples_split - o número mínimo de objetos necessários para a divisão de um nó da árvore.min_samples_leaf - o número mínimo de objetos nas folhas.bootstrap - use para construir subamostras de árvores com retorno.
from sklearn.model_selection import RandomizedSearchCV
n_estimators = [int(x) for x in np.linspace(start = 100, stop = 1000, num = 10)]
max_features = ['log2', 'sqrt']
max_depth = [int(x) for x in np.linspace(start = 1, stop = 15, num = 15)]
min_samples_split = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
min_samples_leaf = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
bootstrap = [True, False]
param_dist = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
rs = RandomizedSearchCV(rfc_2,
param_dist,
n_iter = 100,
cv = 3,
verbose = 1,
n_jobs=-1,
random_state=0)
rs.fit(X_train_scaled_pca, y_train)
rs.best_params_
Com os valores dos parâmetros n_iter = 100e cv = 3, criamos 300 modelos de RF, escolhendo aleatoriamente combinações dos hiper parâmetros apresentados acima. Podemos consultar o atributo best_params_ para obter informações sobre um conjunto de parâmetros que permitem criar o melhor modelo. Mas, nesse estágio, isso pode não nos fornecer os dados mais interessantes sobre os intervalos de parâmetros que vale a pena explorar na próxima rodada de otimização. Para descobrir em qual faixa de valores vale a pena continuar pesquisando, podemos facilmente obter um quadro de dados contendo os resultados do algoritmo RandomizedSearchCV.rs_df = pd.DataFrame(rs.cv_results_).sort_values('rank_test_score').reset_index(drop=True)
rs_df = rs_df.drop([
'mean_fit_time',
'std_fit_time',
'mean_score_time',
'std_score_time',
'params',
'split0_test_score',
'split1_test_score',
'split2_test_score',
'std_test_score'],
axis=1)
rs_df.head(10)
Resultados do algoritmo RandomizedSearchCVAgora, criaremos gráficos de barras nos quais, no eixo X, são os valores do hiperparâmetro e no eixo Y, os valores médios mostrados pelos modelos. Isso possibilitará entender quais valores dos hiperparâmetros, em média, apresentam seu melhor desempenho.fig, axs = plt.subplots(ncols=3, nrows=2)
sns.set(style="whitegrid", color_codes=True, font_scale = 2)
fig.set_size_inches(30,25)
sns.barplot(x='param_n_estimators', y='mean_test_score', data=rs_df, ax=axs[0,0], color='lightgrey')
axs[0,0].set_ylim([.83,.93])axs[0,0].set_title(label = 'n_estimators', size=30, weight='bold')
sns.barplot(x='param_min_samples_split', y='mean_test_score', data=rs_df, ax=axs[0,1], color='coral')
axs[0,1].set_ylim([.85,.93])axs[0,1].set_title(label = 'min_samples_split', size=30, weight='bold')
sns.barplot(x='param_min_samples_leaf', y='mean_test_score', data=rs_df, ax=axs[0,2], color='lightgreen')
axs[0,2].set_ylim([.80,.93])axs[0,2].set_title(label = 'min_samples_leaf', size=30, weight='bold')
sns.barplot(x='param_max_features', y='mean_test_score', data=rs_df, ax=axs[1,0], color='wheat')
axs[1,0].set_ylim([.88,.92])axs[1,0].set_title(label = 'max_features', size=30, weight='bold')
sns.barplot(x='param_max_depth', y='mean_test_score', data=rs_df, ax=axs[1,1], color='lightpink')
axs[1,1].set_ylim([.80,.93])axs[1,1].set_title(label = 'max_depth', size=30, weight='bold')
sns.barplot(x='param_bootstrap',y='mean_test_score', data=rs_df, ax=axs[1,2], color='skyblue')
axs[1,2].set_ylim([.88,.92])
axs[1,2].set_title(label = 'bootstrap', size=30, weight='bold')
plt.show()
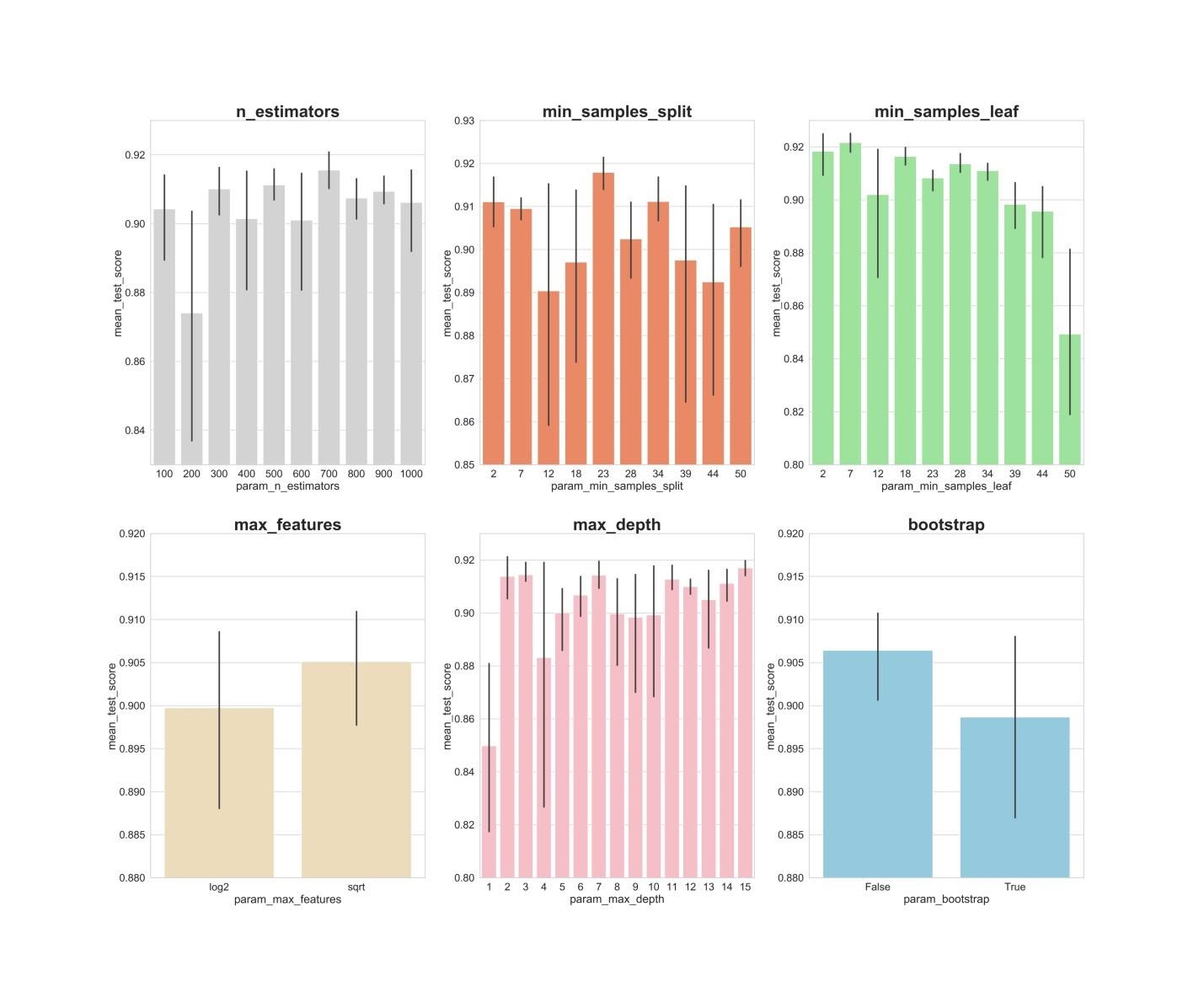
n_estimators: valores de 300, 500, 700, aparentemente, mostram os melhores resultados médios.min_samples_split: Valores pequenos como 2 e 7 parecem mostrar os melhores resultados. O valor 23 também parece bom.Você pode examinar vários valores desse hiperparâmetro além de 2, bem como vários valores de cerca de 23.min_samples_leaf: Há uma sensação de que pequenos valores desse hiperparâmetro fornecem melhores resultados. Isso significa que podemos experimentar valores entre 2 e 7.max_features: opção sqrtfornece o resultado médio mais alto.max_depth: não existe uma relação clara entre o valor do hiperparâmetro e o resultado do modelo, mas há a sensação de que os valores 2, 3, 7, 11, 15 parecem bons.bootstrap: o valor Falsemostra o melhor resultado médio.
Agora, usando essas descobertas, podemos avançar para a segunda rodada de otimização de hiperparâmetros. Isso restringirá o intervalo de valores nos quais estamos interessados.8. Otimização de hiperparâmetros. Rodada 2: GridSearchCV (preparação final dos parâmetros para o modelo nº 3, RF + PCA + HT)
Depois de aplicar o algoritmo RandomizedSearchCV, usaremos o algoritmo GridSearchCV para realizar uma pesquisa mais precisa da melhor combinação de hiperparâmetros. Os mesmos hiperparâmetros são investigados aqui, mas agora estamos aplicando uma pesquisa mais "completa" para sua melhor combinação. Usando o algoritmo GridSearchCV, cada combinação de hiperparâmetros é examinada. Isso requer muito mais recursos computacionais do que usar o algoritmo RandomizedSearchCV quando definimos independentemente o número de iterações de pesquisa. Por exemplo, pesquisar 10 valores para cada um dos 6 hiperparâmetros com validação cruzada em 3 blocos exigirá 10 x 3, ou 3.000.000 de sessões de treinamento em modelo. É por isso que usamos o algoritmo GridSearchCV depois de aplicar o RandomizedSearchCV, reduzimos os intervalos dos valores dos parâmetros estudados.Portanto, usando o que descobrimos com a ajuda do RandomizedSearchCV, examinamos os valores dos hiperparâmetros que se mostraram melhores:from sklearn.model_selection import GridSearchCV
n_estimators = [300,500,700]
max_features = ['sqrt']
max_depth = [2,3,7,11,15]
min_samples_split = [2,3,4,22,23,24]
min_samples_leaf = [2,3,4,5,6,7]
bootstrap = [False]
param_grid = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
gs = GridSearchCV(rfc_2, param_grid, cv = 3, verbose = 1, n_jobs=-1)
gs.fit(X_train_scaled_pca, y_train)
rfc_3 = gs.best_estimator_
gs.best_params_
Aqui, aplicamos a validação cruzada em 3 blocos para 540 (3 x 1 x 5 x 6 x 6 x 1) sessões de treinamento do modelo, o que fornece 1620 sessões de treinamento do modelo. E agora, depois de usarmos RandomizedSearchCV e GridSearchCV, podemos usar o atributo best_params_para descobrir quais valores de hiperparâmetros permitem que o modelo funcione melhor com o conjunto de dados em estudo (esses valores podem ser vistos na parte inferior do bloco de código anterior) . Esses parâmetros são usados para criar o número do modelo 3.9. Avaliação da qualidade dos modelos nos dados de verificação
Agora você pode avaliar os modelos criados nos dados de verificação. Ou seja, estamos falando desses três modelos descritos no início do material.Confira estes modelos:y_pred = rfc.predict(X_test_scaled)
y_pred_pca = rfc.predict(X_test_scaled_pca)
y_pred_gs = gs.best_estimator_.predict(X_test_scaled_pca)
Crie matrizes de erro para os modelos e descubra até que ponto cada um deles é capaz de prever o câncer de mama:from sklearn.metrics import confusion_matrix
conf_matrix_baseline = pd.DataFrame(confusion_matrix(y_test, y_pred), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_baseline_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_pca), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_tuned_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_gs), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
display(conf_matrix_baseline)
display('Baseline Random Forest recall score', recall_score(y_test, y_pred))
display(conf_matrix_baseline_pca)
display('Baseline Random Forest With PCA recall score', recall_score(y_test, y_pred_pca))
display(conf_matrix_tuned_pca)
display('Hyperparameter Tuned Random Forest With PCA Reduced Dimensionality recall score', recall_score(y_test, y_pred_gs))
Resultados do trabalho dos três modelosAqui, a métrica “completude” (recall) é avaliada. O fato é que estamos lidando com um diagnóstico de câncer. Portanto, estamos extremamente interessados em minimizar as previsões negativas falsas emitidas pelos modelos.Diante disso, podemos concluir que o modelo básico de RF apresentou os melhores resultados. Sua taxa de completude foi de 94,97%. No conjunto de dados de teste, houve um registro de 179 pacientes com câncer. O modelo encontrou 170 deles.Sumário
Este estudo fornece uma observação importante. Às vezes, o modelo de RF, que usa o método do componente principal e a otimização em grande escala de hiperparâmetros, pode não funcionar tão bem quanto o modelo mais comum com configurações padrão. Mas esse não é um motivo para se limitar apenas aos modelos mais simples. Sem experimentar modelos diferentes, é impossível dizer qual deles apresentará o melhor resultado. E no caso de modelos usados para prever a presença de câncer em pacientes, podemos dizer que, quanto melhor o modelo - mais vidas podem ser salvas.Queridos leitores! Quais tarefas você resolve usando métodos de aprendizado de máquina?