Halo, Habr! Hari ini saya ingin membagikan contoh kecil bagaimana melakukan analisis klaster. Dalam contoh ini, pembaca tidak akan menemukan jaringan saraf dan arah mode lainnya. Contoh ini dapat berfungsi sebagai titik referensi untuk membuat analisis cluster kecil dan lengkap untuk data lain. Siapa pun yang tertarik - selamat datang di kucing.
Segera melakukan reservasi, artikel ini sama sekali tidak mengklaim akademik secara keseluruhan, keunikan hasil yang diperoleh, atau kelengkapan liputan masalah ini. Artikel ini dimaksudkan untuk menunjukkan langkah-langkah dasar analisis klaster klasik, yang dapat digunakan untuk studi yang sederhana dan bermakna (mungkin sebelum studi yang lebih rinci). Setiap koreksi, komentar, dan penambahan pada jasa diterima.
Data tersebut adalah sampel konsumsi alkohol menurut negara per kapita menurut jenis minuman beralkohol (bir, anggur, alkohol, dll.) Untuk tahun 2010 sebagai persentase dari konsumsi alkohol per kapita. Data juga mengandung: rata-rata konsumsi alkohol harian per kapita dalam gram alkohol murni dan semua (dicatat + tidak dihitung) konsumsi alkohol per kapita (hanya peminum dalam liter alkohol murni).
Pada saat yang sama, setiap negara secara kondisional termasuk dalam salah satu kelompok geografis: timur, tengah dan barat. Divisi ini sangat sewenang-wenang dan sangat kontroversial karena berbagai alasan, tetapi kami akan melanjutkan dari apa yang kami miliki. Sumber Data - Laporan status global tentang alkohol dan kesehatan 2014, S. 289-364
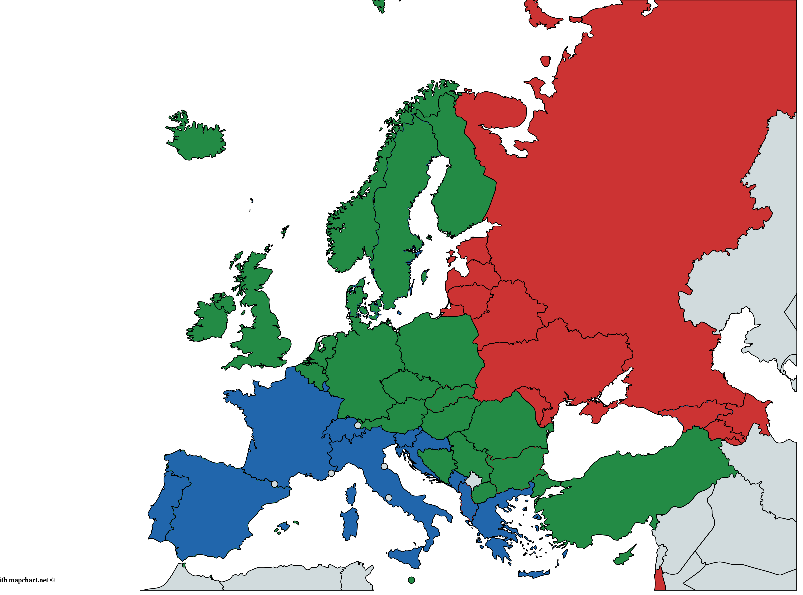
(Dilukis dengan tangan, mungkin ada kesalahan, tetapi ide umum, saya pikir, dapat dimengerti)
Analisis awal
Hubungkan perpustakaan yang digunakan.
library(rgl)
library(heplots)
library(MVN)
library(klaR)
library('Morpho')
library(caret)
library(mclust)
library(ggplot2)
library(GGally)
library(plyr)
library(psych)
library(GPArotation)
library(ggpubr)
, .
#
data <- read.table("alcohol_data.csv", header=TRUE, sep=",")
#
rownames(data) <- make.names(data[,1], unique = TRUE)
# ,
data <- data[,-1]
data <- na.omit(data)
#
head(data)
summary(data)
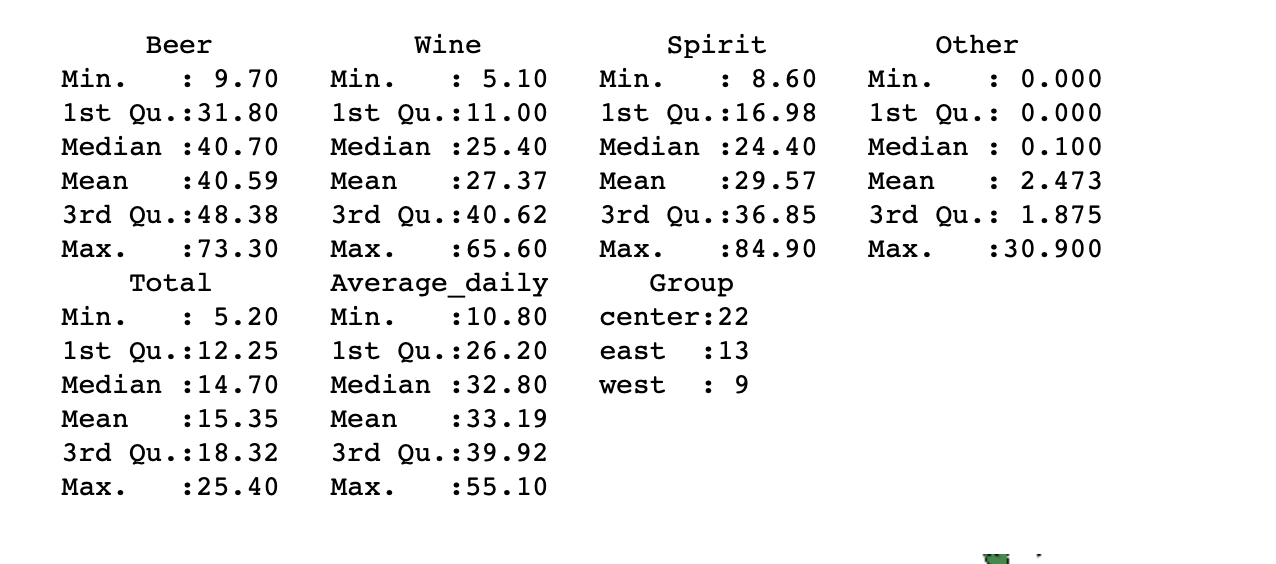
, . , Other , , , , . , , , , . , . - .
, , , .
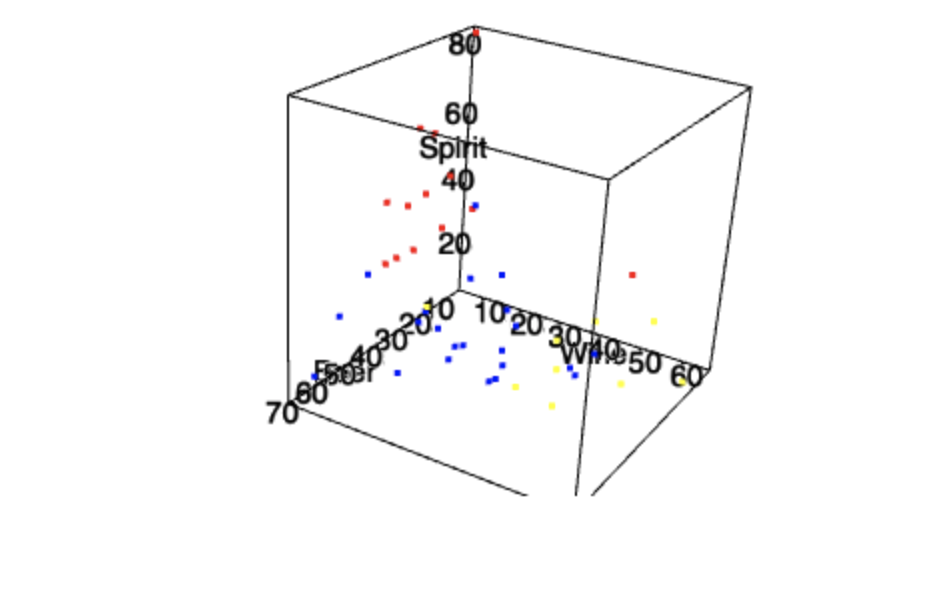
options(rgl.useNULL=TRUE)
open3d()
mfrow3d(2,2)
levelColors <- c('west'='blue', 'east'='red', 'center'='yellow')
plot3d(data$Beer, data$Wine, data$Spirit, xlab="Beer", ylab="Wine", zlab="Spirit", col = levelColors[data$Group], size=3)
widget <- rglwidget()
widget
, . , .
ggpairs(
data,
mapping = ggplot2::aes(color = data$Group),
upper = list(continuous = wrap("cor", alpha = 0.5), combo = "box"),
lower = list(continuous = wrap("points", alpha = 0.3), combo = wrap("dot", alpha = 0.4)),
diag = list(continuous = wrap("densityDiag",alpha = 0.5)),
title = "Alcohol"
)
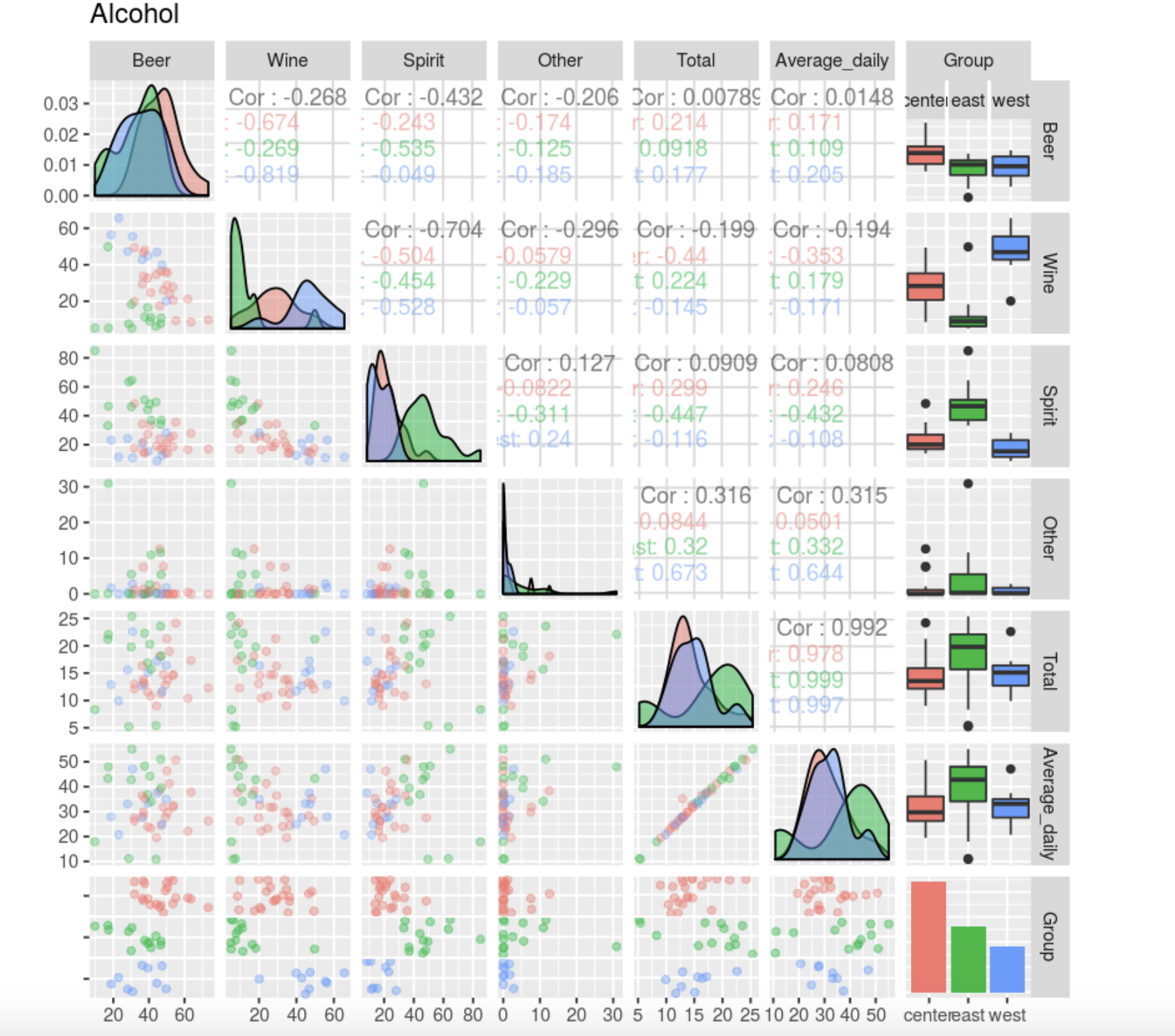
Average Total , Average.
data <- data[, -6]
, , , , . .
data[data$Wine>60,]
, , , , - , , .
data[data$Spirit>70,]
data[data$Spirit<10,]
, , .
,
split(data[,1:5],data$Group)
$center
$east
$west
ggpairs(
data,
mapping = ggplot2::aes(color = data$Group),
diag=list(continuous="bar", alpha=0.4)
)
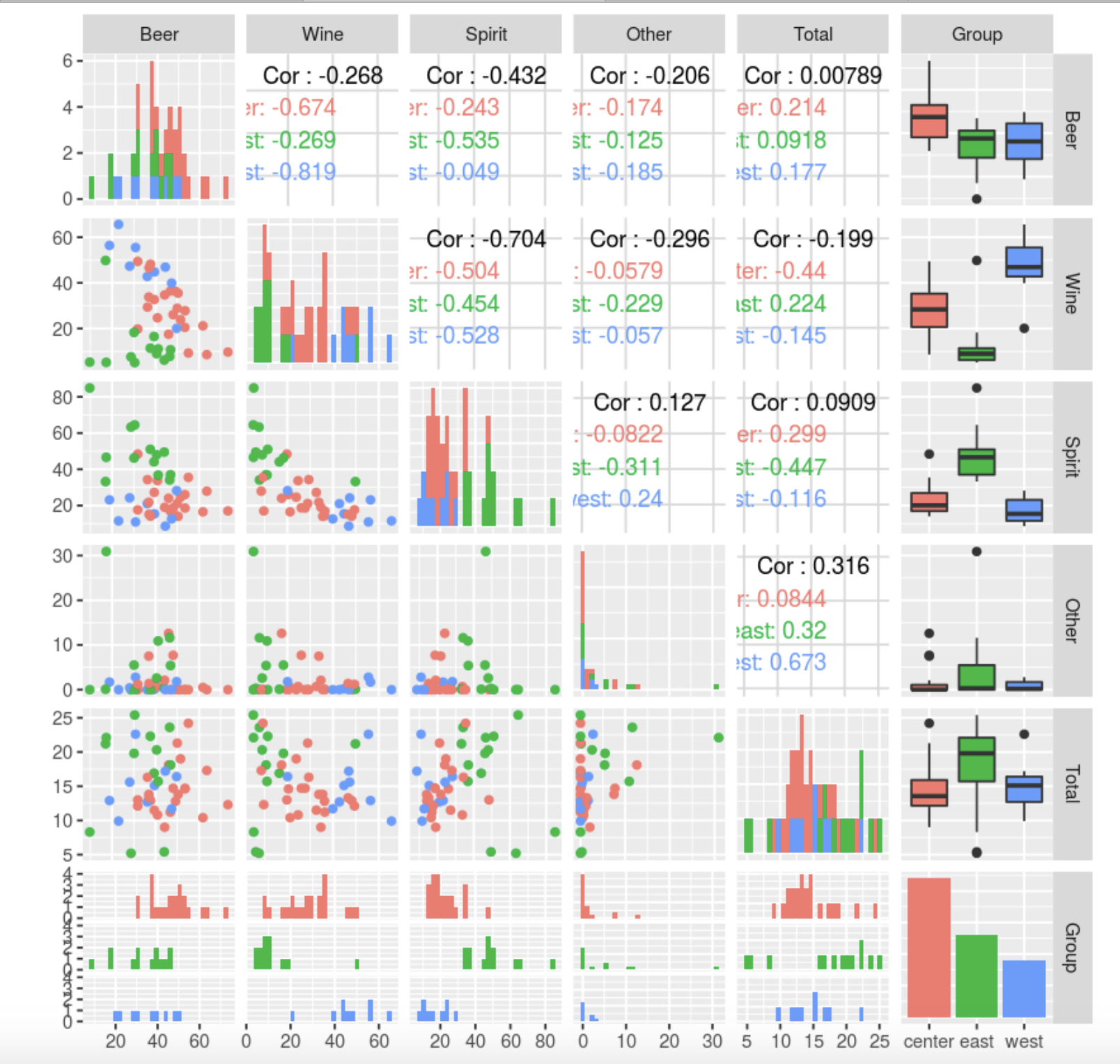
, , . Other, : , , , ( 10-12 , 45, , ). . , , , (). , , . Other .
, , — , — . , — , .
Total Other, . .
, Beer, Spirit Wine . , , , . , , , , , .
Total. , — .
data.group = data[,5]
data <- data[,-5]
data<- data[,-4]
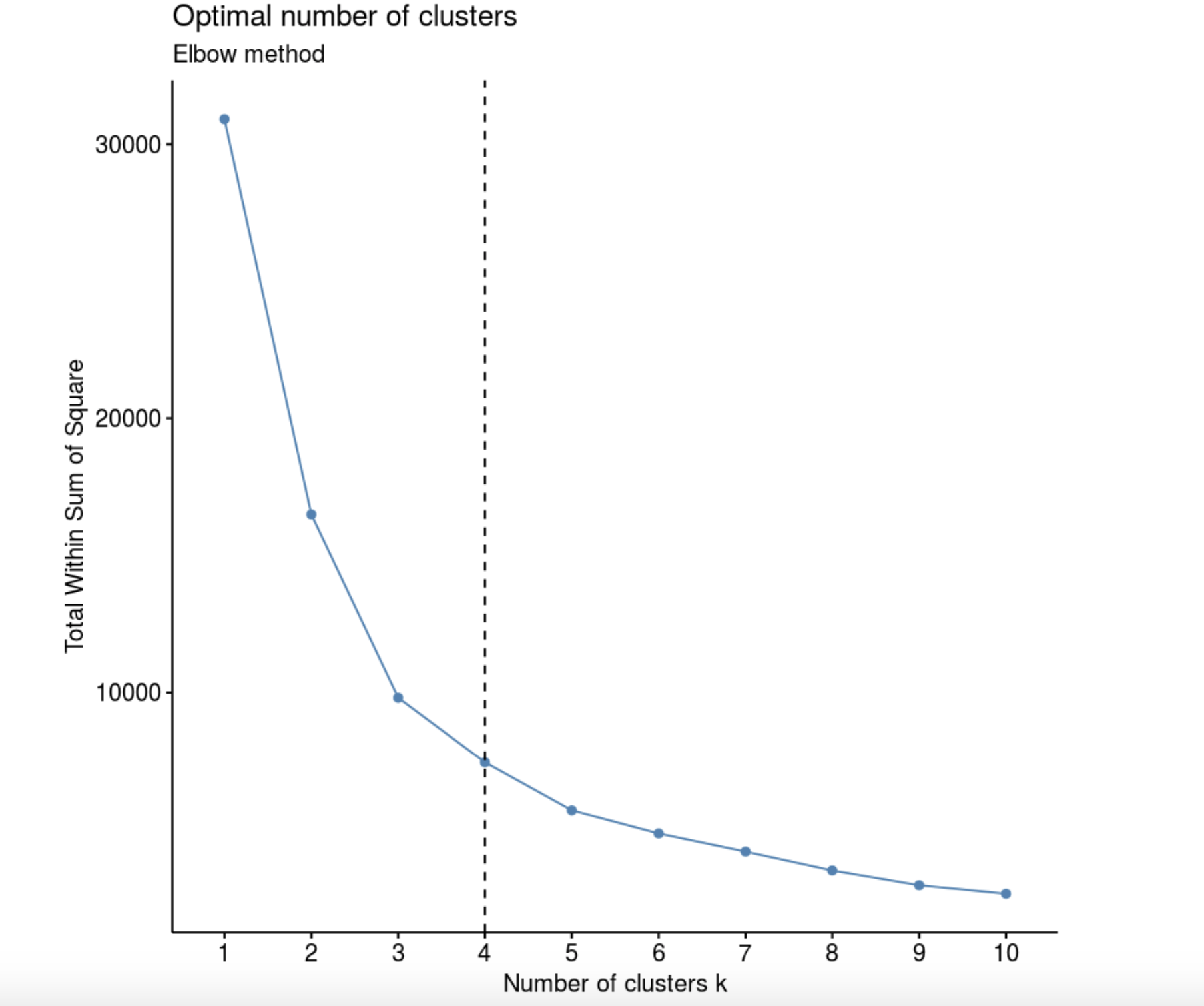
Elbow method (“ ”, “ ”). , k, – W(K), .
library(factoextra)
fviz_nbclust(data, kmeans, method = "wss") +
labs(subtitle = "Elbow method") +
geom_vline(xintercept = 4, linetype = 2)
data.dist <- dist((data))
hc <- hclust(data.dist, method = "ward.D2")
plot(hc, cex = 0.7)
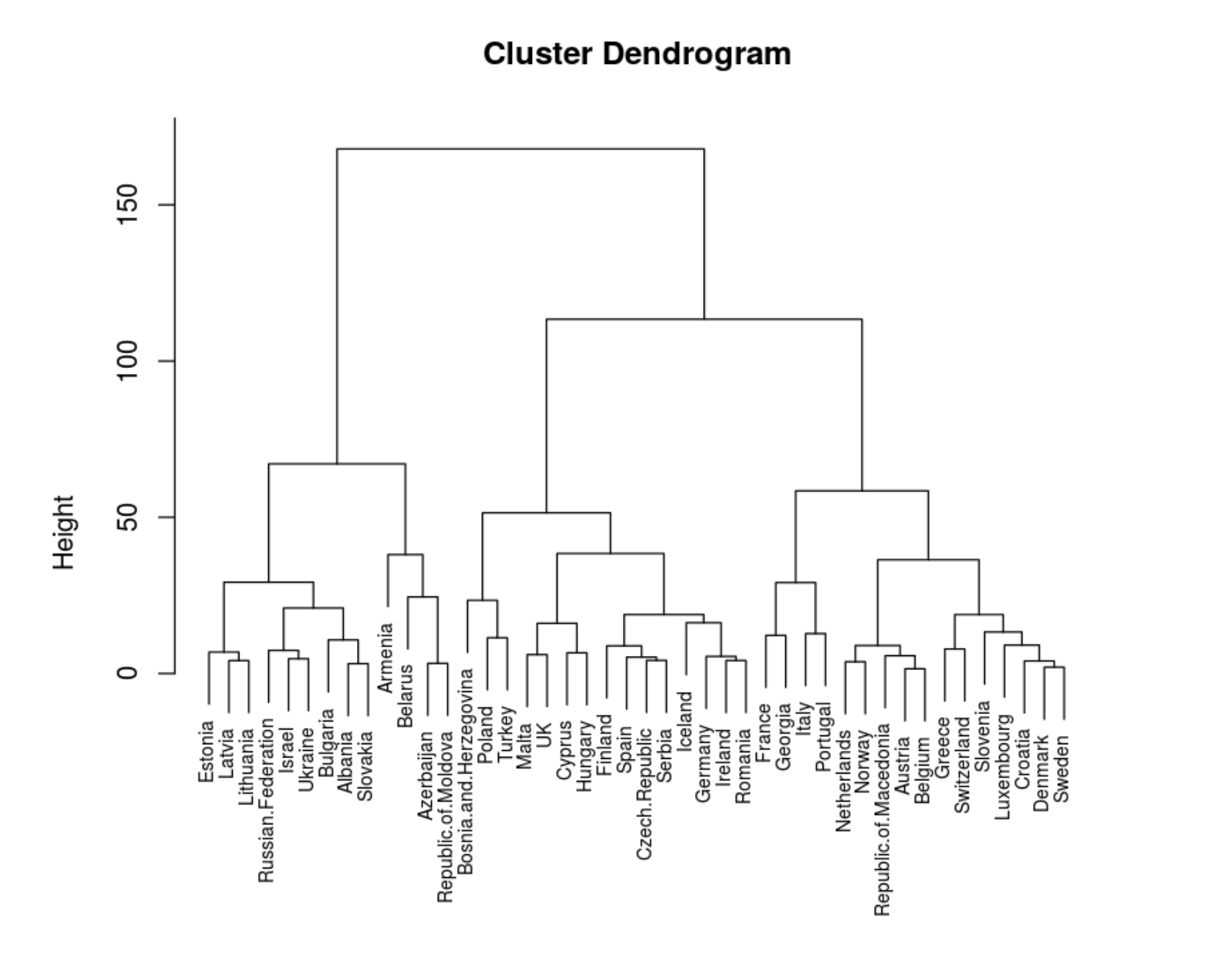
. .
colors=c('green', 'red', 'blue')
hcd = as.dendrogram(hc)
clusMember = cutree(hc, 4)
colLab <- function(n) {
if (is.leaf(n)) {
a <- attributes(n)
labCol <- colors[data.group[n]]
attr(n, "nodePar") <- c(a$nodePar, lab.col = labCol)
}
n
}
clusDendro = dendrapply(hcd, colLab)
plot(clusDendro, main = "Cool Dendrogram", type = "triangle")
rect.hclust(hc, k = 4)
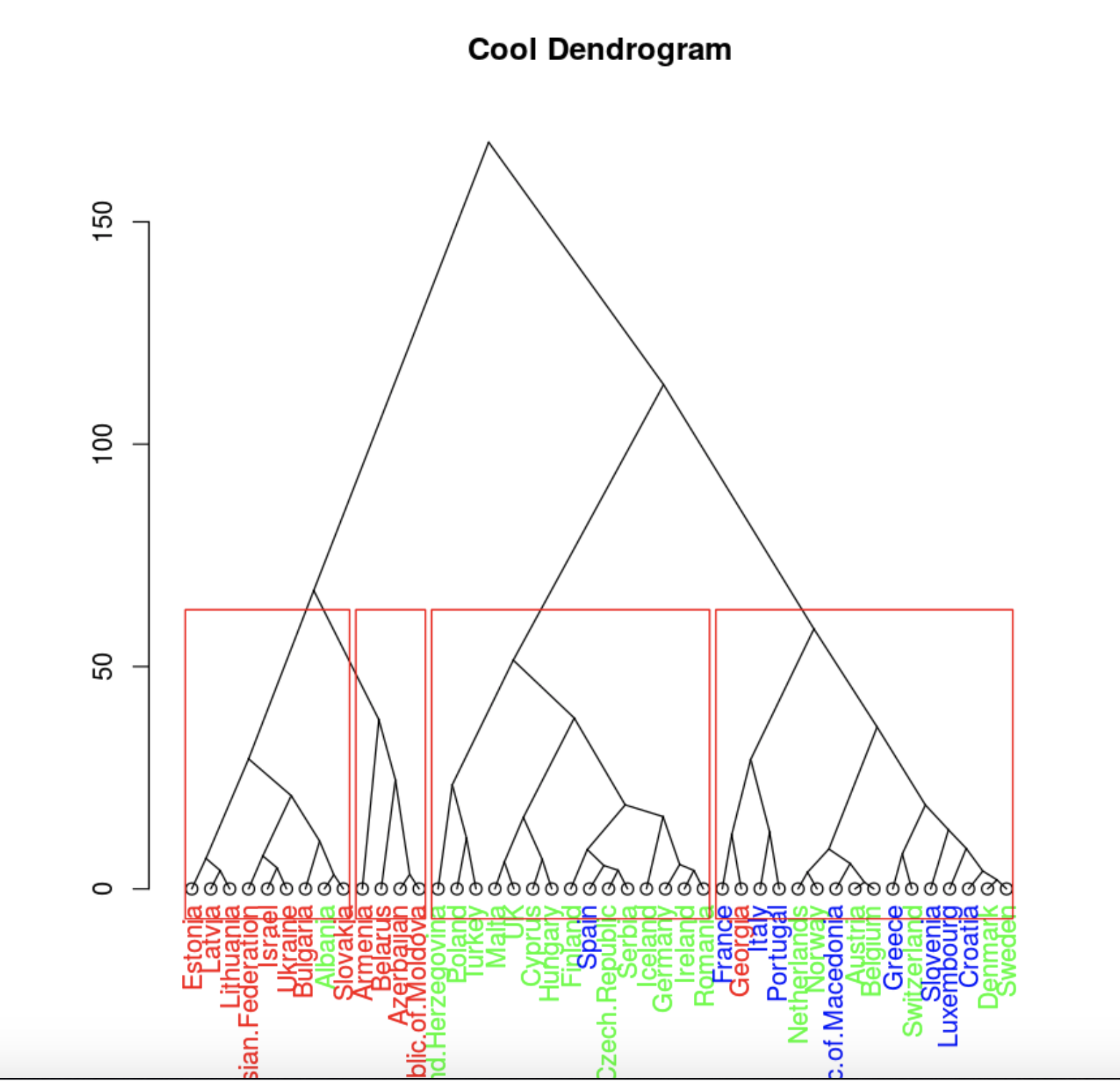
. , .
, , , 4 .
plot(clusDendro, main = "Cool Dendrogram", type = "triangle")
data.hclas_group <- factor(cutree(hc, k = 3))
rect.hclust(hc, k = 3)
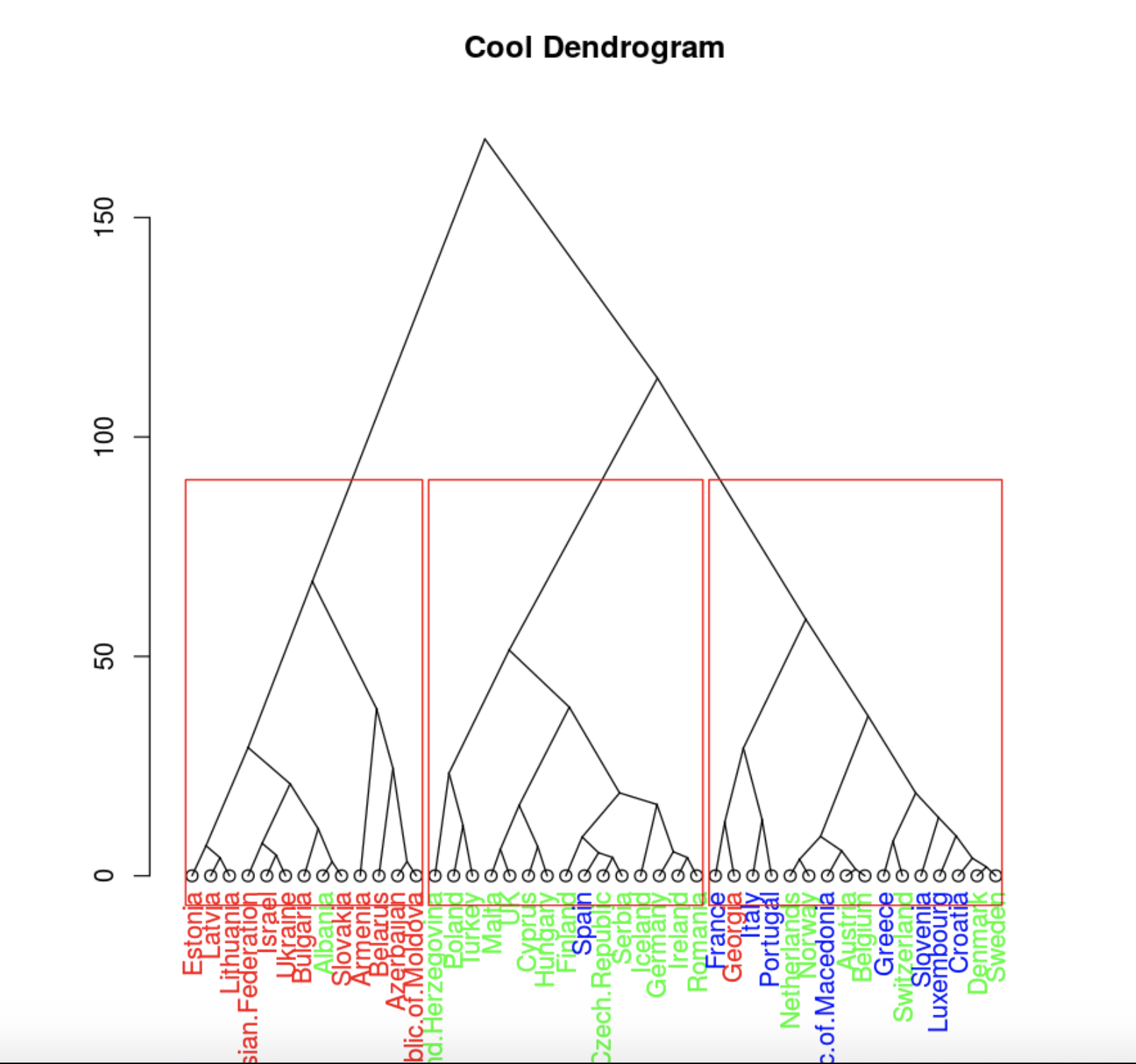
, , .
library(FactoMineR)
res.pca <- PCA(data,scale.unit=T, graph = F)
fviz_pca_biplot(res.pca,
col = colors[data.hclas_group], palette = "jco",
label = "var",
ellipse.level = 0.8,
addEllipses = T,
col.var = "black",
legend.title = "groups4")
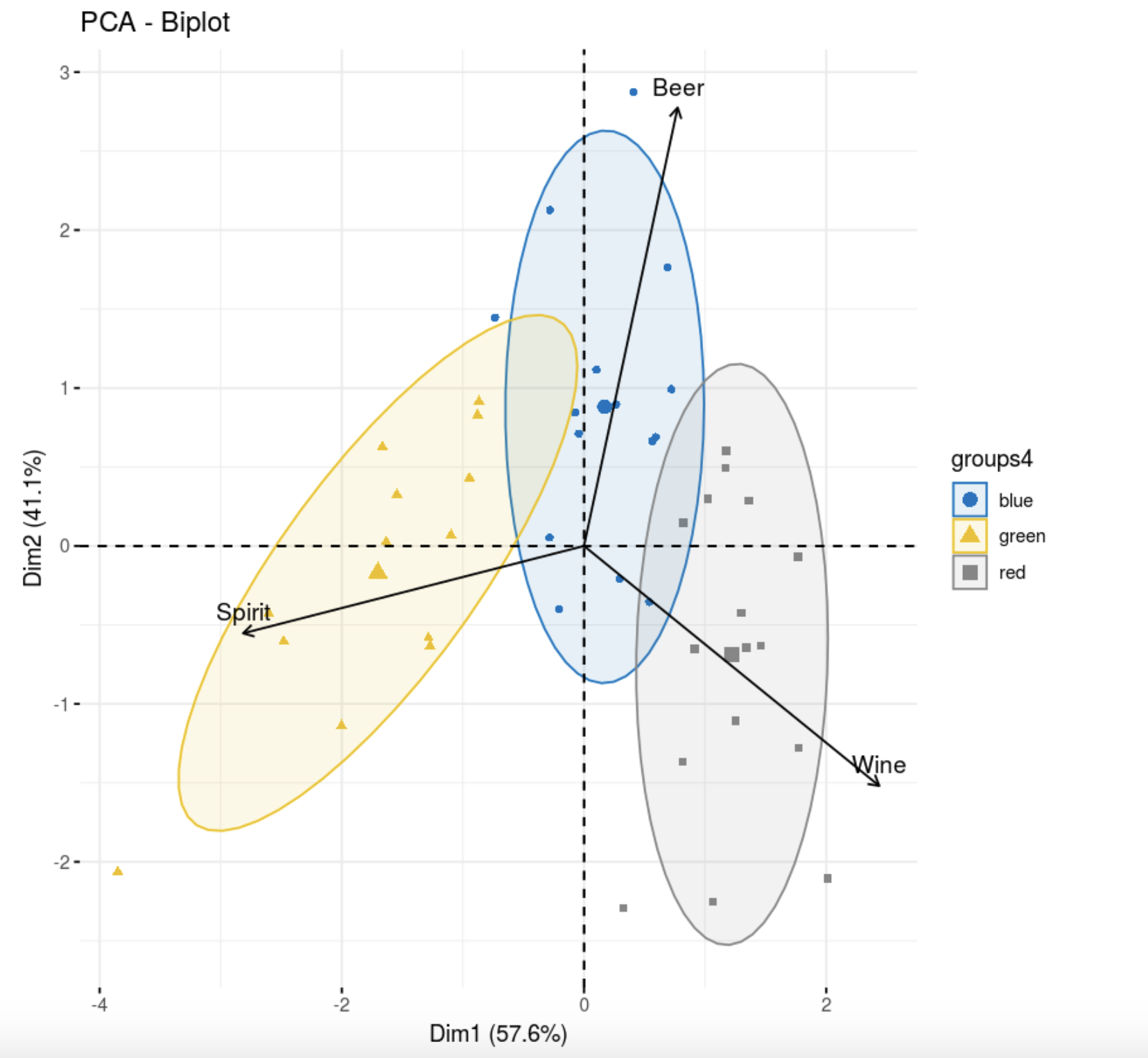
, , . , , , , . , , , k-++.
library(flexclust)
data.kk <- kcca(data, k=3, family=kccaFamily("kmeans"),
control=list(initcent="kmeanspp"))
fviz_pca_biplot(res.pca,
col.ind =as.factor(data.kk@cluster), palette = "jco",
label = "var",
ellipse.level = 0.8,
addEllipses = T,
col.var = "black", repel = TRUE,
legend.title = "clusters")
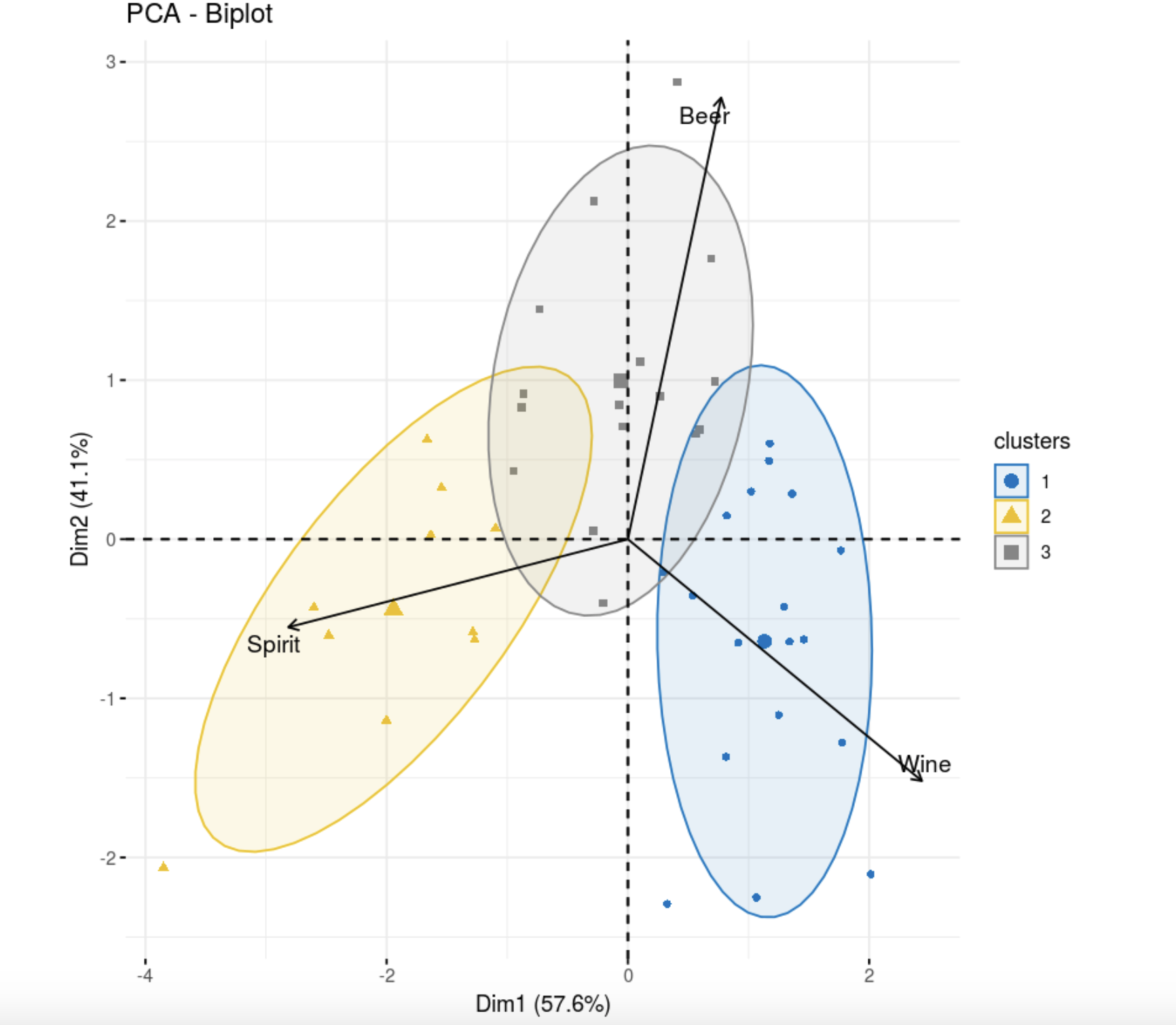
, k- . , , .
, , hclust. .

, , . . , .
. . , , , . , , . , .
Dimungkinkan untuk melakukan pengelompokan berdasarkan asumsi model kluster menggunakan kriteria informasi (di sini adalah deskripsi ), dan juga mencoba analisis diskriminan klasik untuk kumpulan data ini. Jika artikel ini bermanfaat, saya berencana untuk menerbitkan sekuel.