Spesialis pemrosesan dan analisis data memiliki banyak alat untuk membuat model klasifikasi. Salah satu metode yang paling populer dan dapat diandalkan untuk mengembangkan model tersebut adalah dengan menggunakan algoritma Random Forest (RF). Untuk mencoba meningkatkan kinerja model yang dibangun menggunakan algoritma RF , Anda dapat menggunakan optimisasi hyperparameter model ( Hyperparameter Tuning , HT). Selain itu, ada pendekatan luas yang menurutnya data, sebelum dipindahkan ke model, diproses menggunakan Analisis Komponen Utama. , PCA). Tetapi apakah itu layak untuk digunakan? Bukankah tujuan utama dari algoritma RF untuk membantu analis mengartikan pentingnya sifat-sifat tersebut?Ya, penggunaan algoritma PCA dapat menyebabkan sedikit komplikasi dari interpretasi setiap "fitur" dalam analisis "pentingnya fitur" dari model RF. Namun, algoritma PCA mengurangi dimensi ruang fitur, yang dapat menyebabkan penurunan jumlah fitur yang perlu diproses oleh model RF. Harap dicatat bahwa volume perhitungan adalah salah satu kelemahan utama dari algoritma hutan acak (yaitu, ini bisa memakan waktu lama untuk menyelesaikan model). Penerapan algoritma PCA dapat menjadi bagian yang sangat penting dalam pemodelan, terutama dalam kasus di mana mereka bekerja dengan ratusan atau bahkan ribuan fitur. Akibatnya, jika hal yang paling penting adalah menciptakan model yang paling efektif, dan pada saat yang sama Anda dapat mengorbankan keakuratan dalam menentukan pentingnya sifat-sifat tersebut, maka PCA mungkin patut dicoba.Sekarang to the point. Kami akan bekerja dengan dataset kanker payudara - Scikit-belajar "kanker payudara" . Kami akan membuat tiga model dan membandingkan efektivitasnya. Yaitu, kita berbicara tentang model-model berikut:
, PCA). Tetapi apakah itu layak untuk digunakan? Bukankah tujuan utama dari algoritma RF untuk membantu analis mengartikan pentingnya sifat-sifat tersebut?Ya, penggunaan algoritma PCA dapat menyebabkan sedikit komplikasi dari interpretasi setiap "fitur" dalam analisis "pentingnya fitur" dari model RF. Namun, algoritma PCA mengurangi dimensi ruang fitur, yang dapat menyebabkan penurunan jumlah fitur yang perlu diproses oleh model RF. Harap dicatat bahwa volume perhitungan adalah salah satu kelemahan utama dari algoritma hutan acak (yaitu, ini bisa memakan waktu lama untuk menyelesaikan model). Penerapan algoritma PCA dapat menjadi bagian yang sangat penting dalam pemodelan, terutama dalam kasus di mana mereka bekerja dengan ratusan atau bahkan ribuan fitur. Akibatnya, jika hal yang paling penting adalah menciptakan model yang paling efektif, dan pada saat yang sama Anda dapat mengorbankan keakuratan dalam menentukan pentingnya sifat-sifat tersebut, maka PCA mungkin patut dicoba.Sekarang to the point. Kami akan bekerja dengan dataset kanker payudara - Scikit-belajar "kanker payudara" . Kami akan membuat tiga model dan membandingkan efektivitasnya. Yaitu, kita berbicara tentang model-model berikut:- Model dasar berdasarkan pada algoritma RF (kami akan menyingkat model RF ini).
- Model yang sama dengan No. 1, tetapi di mana pengurangan dimensi ruang fitur diterapkan menggunakan metode komponen utama (RF + PCA).
- Model yang sama dengan No. 2, tetapi dibangun menggunakan optimisasi hyperparameter (RF + PCA + HT).
1. Impor data
Untuk memulai, muat data dan buat bingkai data Pandas. Karena kita menggunakan set data "mainan" yang telah dibersihkan dari Scikit-learn, maka setelah itu kita sudah dapat memulai proses pemodelan. Tetapi bahkan ketika menggunakan data tersebut, Anda disarankan untuk selalu mulai bekerja dengan melakukan analisis awal data menggunakan perintah berikut yang diterapkan pada bingkai data ( df):df.head() - untuk melihat bingkai data baru dan melihat apakah itu terlihat seperti yang diharapkan.df.info()- untuk mengetahui fitur tipe data dan konten kolom. Mungkin perlu melakukan konversi tipe data sebelum melanjutkan.df.isna()- untuk memastikan bahwa tidak ada nilai dalam data NaN. Nilai yang sesuai, jika ada, mungkin perlu diproses entah bagaimana, atau, jika perlu, mungkin perlu untuk menghapus seluruh baris dari bingkai data.df.describe() - untuk mengetahui nilai rata-rata minimum, maksimum, rata-rata dari indikator dalam kolom, untuk mengetahui indikator rata-rata kuadrat dan kemungkinan penyimpangan dalam kolom.
Dalam dataset kami, kolom cancer(kanker) adalah variabel target yang nilainya ingin diprediksi menggunakan model. 0berarti "tidak ada penyakit." 1- "Kehadiran penyakit."import pandas as pd
from sklearn.datasets import load_breast_cancer
columns = ['mean radius', 'mean texture', 'mean perimeter', 'mean area', 'mean smoothness', 'mean compactness', 'mean concavity', 'mean concave points', 'mean symmetry', 'mean fractal dimension', 'radius error', 'texture error', 'perimeter error', 'area error', 'smoothness error', 'compactness error', 'concavity error', 'concave points error', 'symmetry error', 'fractal dimension error', 'worst radius', 'worst texture', 'worst perimeter', 'worst area', 'worst smoothness', 'worst compactness', 'worst concavity', 'worst concave points', 'worst symmetry', 'worst fractal dimension']
dataset = load_breast_cancer()
data = pd.DataFrame(dataset['data'], columns=columns)
data['cancer'] = dataset['target']
display(data.head())
display(data.info())
display(data.isna().sum())
display(data.describe())
. . , cancer, , . 0 « ». 1 — « »2.
Sekarang pisahkan data menggunakan fungsi Scikit-learn train_test_split. Kami ingin memberikan model sebanyak mungkin data pelatihan. Namun, kita perlu memiliki data yang cukup untuk menguji model. Secara umum, kita dapat mengatakan bahwa, ketika jumlah baris dalam kumpulan data meningkat, demikian juga jumlah data yang dapat dianggap mendidik.Misalnya, jika ada jutaan baris, Anda dapat membagi set dengan menyoroti 90% dari baris untuk data pelatihan dan 10% untuk data uji. Tetapi kumpulan data uji hanya berisi 569 baris. Dan ini bukan untuk pelatihan dan pengujian model. Sebagai hasilnya, agar adil dalam kaitannya dengan data pelatihan dan verifikasi, kami akan membagi set menjadi dua bagian yang sama - 50% - data pelatihan dan 50% - data verifikasi. Kami memasangstratify=y untuk memastikan bahwa set data pelatihan dan tes memiliki rasio yang sama dengan 0 dan 1 seperti set data asli.from sklearn.model_selection import train_test_split
X = data.drop('cancer', axis=1)
y = data['cancer']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.50, random_state = 2020, stratify=y)
3. Penskalaan data
Sebelum melanjutkan ke pemodelan, Anda perlu "memusat" dan "membakukan" data dengan menskalanya . Penskalaan dilakukan karena fakta bahwa jumlah yang berbeda dinyatakan dalam unit yang berbeda. Prosedur ini memungkinkan Anda untuk mengatur "pertarungan yang adil" antara tanda-tanda dalam menentukan kepentingannya. Selain itu, kami mengonversi y_traindari tipe data Pandas Serieske array NumPy sehingga nantinya model dapat bekerja dengan target yang sesuai.import numpy as np
from sklearn.preprocessing import StandardScaler
ss = StandardScaler()
X_train_scaled = ss.fit_transform(X_train)
X_test_scaled = ss.transform(X_test)
y_train = np.array(y_train)
4. Pelatihan model dasar (model No. 1, RF)
Sekarang buat model nomor 1. Di dalamnya, kita ingat bahwa hanya algoritma Random Forest yang digunakan. Ini menggunakan semua fitur dan dikonfigurasi menggunakan nilai default (detail tentang pengaturan ini dapat ditemukan dalam dokumentasi untuk sklearn.ensemble.RandomForestClassifier ). Inisialisasi model. Setelah itu, kami akan melatihnya tentang data yang diskalakan. Keakuratan model dapat diukur pada data pelatihan:from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import recall_score
rfc = RandomForestClassifier()
rfc.fit(X_train_scaled, y_train)
display(rfc.score(X_train_scaled, y_train))
Jika kita tertarik untuk mengetahui sifat mana yang paling penting untuk model RF dalam memprediksi kanker payudara, kita dapat memvisualisasikan dan mengukur indeks keparahan sifat dengan merujuk pada atribut feature_importances_:feats = {}
for feature, importance in zip(data.columns, rfc_1.feature_importances_):
feats[feature] = importance
importances = pd.DataFrame.from_dict(feats, orient='index').rename(columns={0: 'Gini-Importance'})
importances = importances.sort_values(by='Gini-Importance', ascending=False)
importances = importances.reset_index()
importances = importances.rename(columns={'index': 'Features'})
sns.set(font_scale = 5)
sns.set(style="whitegrid", color_codes=True, font_scale = 1.7)
fig, ax = plt.subplots()
fig.set_size_inches(30,15)
sns.barplot(x=importances['Gini-Importance'], y=importances['Features'], data=importances, color='skyblue')
plt.xlabel('Importance', fontsize=25, weight = 'bold')
plt.ylabel('Features', fontsize=25, weight = 'bold')
plt.title('Feature Importance', fontsize=25, weight = 'bold')
display(plt.show())
display(importances)
Visualisasi "pentingnya" tandaIndikator signifikansi5. Metode komponen utama
Sekarang mari kita bertanya bagaimana kita dapat meningkatkan model RF dasar. Dengan menggunakan teknik mengurangi dimensi ruang fitur, dimungkinkan untuk menyajikan set data awal melalui variabel yang lebih sedikit dan pada saat yang sama mengurangi jumlah sumber daya komputasi yang diperlukan untuk memastikan operasi model. Menggunakan PCA, Anda dapat mempelajari varians sampel kumulatif dari fitur-fitur ini untuk memahami fitur apa yang menjelaskan sebagian besar varian dalam data.Kami menginisialisasi objek PCA ( pca_test), menunjukkan jumlah komponen (fitur) yang perlu dipertimbangkan. Kami menetapkan indikator ini ke 30 untuk melihat perbedaan yang dijelaskan dari semua komponen yang dihasilkan sebelum memutuskan berapa banyak komponen yang kami butuhkan. Lalu kami transfer ke pca_testdata yang diskalakanX_trainmenggunakan metode ini pca_test.fit(). Setelah itu kami memvisualisasikan data.import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.decomposition import PCA
pca_test = PCA(n_components=30)
pca_test.fit(X_train_scaled)
sns.set(style='whitegrid')
plt.plot(np.cumsum(pca_test.explained_variance_ratio_))
plt.xlabel('number of components')
plt.ylabel('cumulative explained variance')
plt.axvline(linewidth=4, color='r', linestyle = '--', x=10, ymin=0, ymax=1)
display(plt.show())
evr = pca_test.explained_variance_ratio_
cvr = np.cumsum(pca_test.explained_variance_ratio_)
pca_df = pd.DataFrame()
pca_df['Cumulative Variance Ratio'] = cvr
pca_df['Explained Variance Ratio'] = evr
display(pca_df.head(10))
Setelah jumlah komponen yang digunakan melebihi 10, peningkatan jumlahnya tidak banyak meningkatkan varian yang dijelaskanKerangka data ini berisi indikator seperti Cumulative Variance Ratio (ukuran kumulatif dari varians yang dijelaskan dari data) dan Explained Variance Ratio (kontribusi masing-masing komponen terhadap total volume dari varian yang dijelaskan)Jika Anda melihat pada kerangka data di atas, ternyata menggunakan PCA untuk berpindah dari 30 variabel menjadi 10 untuk komponen memungkinkan untuk menjelaskan 95% dari dispersi data. 20 komponen lainnya menyumbang kurang dari 5% dari varians, yang berarti bahwa kita dapat menolaknya. Mengikuti logika ini, kami menggunakan PCA untuk mengurangi jumlah komponen dari 30 menjadi 10 untukX_traindanX_test. Kami menulis ini artifisial diciptakan “mengurangi dimensi” Data set diX_train_scaled_pcadan diX_test_scaled_pca.pca = PCA(n_components=10)
pca.fit(X_train_scaled)
X_train_scaled_pca = pca.transform(X_train_scaled)
X_test_scaled_pca = pca.transform(X_test_scaled)
Setiap komponen adalah kombinasi linear dari variabel sumber dengan "bobot" yang sesuai. Kita dapat melihat "bobot" ini untuk setiap komponen dengan membuat bingkai data.pca_dims = []
for x in range(0, len(pca_df)):
pca_dims.append('PCA Component {}'.format(x))
pca_test_df = pd.DataFrame(pca_test.components_, columns=columns, index=pca_dims)
pca_test_df.head(10).T
Dataframe Informasi Komponen6. Pelatihan model RF dasar setelah menerapkan metode komponen utama ke data (model No. 2, RF + PCA)
Sekarang kita dapat meneruskan ke data model RF dasar lainnya X_train_scaled_pcadan y_traindan dapat mencari tahu apakah ada peningkatan dalam akurasi prediksi yang dikeluarkan oleh model.rfc = RandomForestClassifier()
rfc.fit(X_train_scaled_pca, y_train)
display(rfc.score(X_train_scaled_pca, y_train))
Model membandingkan di bawah ini.7. Optimalisasi hiperparameter. Babak 1: RandomizedSearchCV
Setelah memproses data menggunakan metode komponen utama, Anda dapat mencoba menggunakan optimisasi model hiperparameter untuk meningkatkan kualitas prediksi yang dihasilkan oleh model RF. Hyperparameters dapat dianggap sebagai sesuatu seperti "pengaturan" model. Pengaturan yang sempurna untuk satu set data tidak akan berfungsi untuk yang lain - itu sebabnya Anda perlu mengoptimalkannya.Anda bisa mulai dengan algoritma RandomizedSearchCV, yang memungkinkan Anda untuk menjelajahi kira-kira berbagai nilai. Deskripsi semua hiperparameter untuk model RF dapat ditemukan di sini .Dalam perjalanan kerja, kami menghasilkan entitas param_distyang berisi, untuk setiap hyperparameter, rentang nilai yang perlu diuji. Selanjutnya, kita inisialisasi objek.rsmenggunakan fungsi RandomizedSearchCV(), meneruskannya model RF param_dist,, jumlah iterasi dan jumlah validasi silang yang perlu dilakukan.Hyperparameter verbosememungkinkan Anda untuk mengontrol jumlah informasi yang ditampilkan oleh model selama operasinya (seperti output informasi selama pelatihan model). Hyperparameter n_jobsmemungkinkan Anda menentukan berapa banyak inti prosesor yang perlu Anda gunakan untuk memastikan pengoperasian model. Mengaturnya n_jobske nilai -1akan mengarah ke model yang lebih cepat, karena ini akan menggunakan semua inti prosesor.Kami akan terlibat dalam pemilihan hyperparameter berikut:n_estimators - jumlah "pohon" di "hutan acak".max_features - jumlah fitur untuk memilih pemisahan.max_depth - kedalaman maksimum pohon.min_samples_split - jumlah minimum objek yang diperlukan untuk membelah simpul pohon.min_samples_leaf - jumlah minimum objek dalam daun.bootstrap - digunakan untuk membangun pohon subsampel dengan pengembalian.
from sklearn.model_selection import RandomizedSearchCV
n_estimators = [int(x) for x in np.linspace(start = 100, stop = 1000, num = 10)]
max_features = ['log2', 'sqrt']
max_depth = [int(x) for x in np.linspace(start = 1, stop = 15, num = 15)]
min_samples_split = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
min_samples_leaf = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
bootstrap = [True, False]
param_dist = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
rs = RandomizedSearchCV(rfc_2,
param_dist,
n_iter = 100,
cv = 3,
verbose = 1,
n_jobs=-1,
random_state=0)
rs.fit(X_train_scaled_pca, y_train)
rs.best_params_
Dengan nilai-nilai parameter n_iter = 100dan cv = 3, kami menciptakan 300 model RF, secara acak memilih kombinasi dari parameter hiper yang disajikan di atas. Kami dapat merujuk ke atribut best_params_ untuk informasi tentang serangkaian parameter yang memungkinkan Anda untuk membuat model terbaik. Tetapi pada tahap ini, ini mungkin tidak memberi kami data paling menarik tentang rentang parameter yang perlu ditelusuri dalam putaran optimasi berikutnya. Untuk mengetahui rentang nilai mana yang layak untuk terus dicari, kita dapat dengan mudah mendapatkan bingkai data yang berisi hasil dari algoritma RandomizedSearchCV.rs_df = pd.DataFrame(rs.cv_results_).sort_values('rank_test_score').reset_index(drop=True)
rs_df = rs_df.drop([
'mean_fit_time',
'std_fit_time',
'mean_score_time',
'std_score_time',
'params',
'split0_test_score',
'split1_test_score',
'split2_test_score',
'std_test_score'],
axis=1)
rs_df.head(10)
Hasil dari algoritma RandomizedSearchCVSekarang kita akan membuat grafik batang yang, pada sumbu X, adalah nilai hyperparameter, dan pada sumbu Y adalah nilai rata-rata yang ditunjukkan oleh model. Ini akan memungkinkan untuk memahami nilai hiperparameter apa, secara rata-rata, menunjukkan kinerja terbaiknya.fig, axs = plt.subplots(ncols=3, nrows=2)
sns.set(style="whitegrid", color_codes=True, font_scale = 2)
fig.set_size_inches(30,25)
sns.barplot(x='param_n_estimators', y='mean_test_score', data=rs_df, ax=axs[0,0], color='lightgrey')
axs[0,0].set_ylim([.83,.93])axs[0,0].set_title(label = 'n_estimators', size=30, weight='bold')
sns.barplot(x='param_min_samples_split', y='mean_test_score', data=rs_df, ax=axs[0,1], color='coral')
axs[0,1].set_ylim([.85,.93])axs[0,1].set_title(label = 'min_samples_split', size=30, weight='bold')
sns.barplot(x='param_min_samples_leaf', y='mean_test_score', data=rs_df, ax=axs[0,2], color='lightgreen')
axs[0,2].set_ylim([.80,.93])axs[0,2].set_title(label = 'min_samples_leaf', size=30, weight='bold')
sns.barplot(x='param_max_features', y='mean_test_score', data=rs_df, ax=axs[1,0], color='wheat')
axs[1,0].set_ylim([.88,.92])axs[1,0].set_title(label = 'max_features', size=30, weight='bold')
sns.barplot(x='param_max_depth', y='mean_test_score', data=rs_df, ax=axs[1,1], color='lightpink')
axs[1,1].set_ylim([.80,.93])axs[1,1].set_title(label = 'max_depth', size=30, weight='bold')
sns.barplot(x='param_bootstrap',y='mean_test_score', data=rs_df, ax=axs[1,2], color='skyblue')
axs[1,2].set_ylim([.88,.92])
axs[1,2].set_title(label = 'bootstrap', size=30, weight='bold')
plt.show()
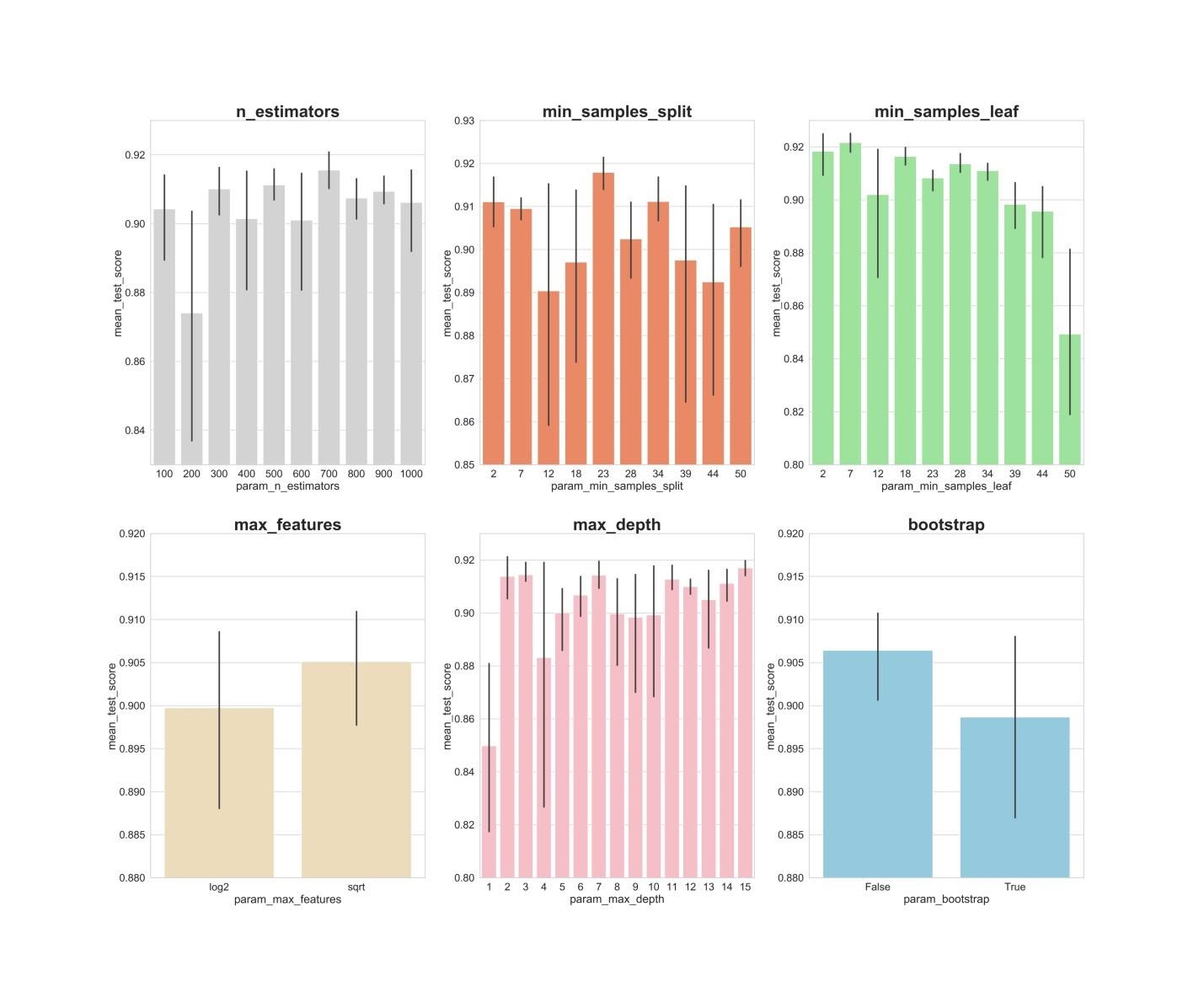
n_estimators: nilai 300, 500, 700, tampaknya, menunjukkan hasil rata-rata terbaik.min_samples_split: Nilai-nilai kecil seperti 2 dan 7 tampaknya menunjukkan hasil terbaik. Nilai 23 juga terlihat bagus. Anda dapat memeriksa beberapa nilai hyperparameter ini lebih dari 2, dan juga beberapa nilai sekitar 23.min_samples_leaf: Ada perasaan bahwa nilai-nilai kecil dari hyperparameter ini memberikan hasil yang lebih baik. Ini berarti bahwa kita dapat mengalami nilai antara 2 dan 7.max_features: opsi sqrtmemberikan hasil rata-rata tertinggi.max_depth: tidak ada hubungan yang jelas antara nilai hyperparameter dan hasil model, tetapi ada perasaan bahwa nilai 2, 3, 7, 11, 15 terlihat bagus.bootstrap: nilai Falsemenunjukkan hasil rata-rata terbaik.
Sekarang, dengan menggunakan temuan ini, kita dapat beralih ke putaran kedua optimasi hyperparameters. Ini akan mempersempit rentang nilai yang kami minati.8. Optimalisasi hiperparameter. Babak 2: GridSearchCV (persiapan akhir parameter untuk model No. 3, RF + PCA + HT)
Setelah menerapkan algoritma RandomizedSearchCV, kami akan menggunakan algoritma GridSearchCV untuk melakukan pencarian yang lebih akurat untuk kombinasi hyperparameter terbaik. Hyperparameter yang sama diselidiki di sini, tetapi sekarang kami menerapkan pencarian yang lebih "menyeluruh" untuk kombinasi terbaiknya. Dengan menggunakan algoritma GridSearchCV, setiap kombinasi hyperparameter diperiksa. Ini membutuhkan lebih banyak sumber daya komputasi daripada menggunakan algoritma RandomizedSearchCV ketika kami secara independen mengatur jumlah iterasi pencarian. Misalnya, meneliti 10 nilai untuk masing-masing 6 hiperparameter dengan validasi silang dalam 3 blok akan memerlukan 10⁶ x 3, atau 3.000.000 sesi pelatihan model. Itu sebabnya kami menggunakan algoritma GridSearchCV setelah, setelah menerapkan RandomizedSearchCV, kami mempersempit rentang nilai dari parameter yang dipelajari.Jadi, menggunakan apa yang kami temukan dengan bantuan RandomizedSearchCV, kami memeriksa nilai-nilai hyperparameters yang telah menunjukkan diri mereka yang terbaik:from sklearn.model_selection import GridSearchCV
n_estimators = [300,500,700]
max_features = ['sqrt']
max_depth = [2,3,7,11,15]
min_samples_split = [2,3,4,22,23,24]
min_samples_leaf = [2,3,4,5,6,7]
bootstrap = [False]
param_grid = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
gs = GridSearchCV(rfc_2, param_grid, cv = 3, verbose = 1, n_jobs=-1)
gs.fit(X_train_scaled_pca, y_train)
rfc_3 = gs.best_estimator_
gs.best_params_
Di sini kami menerapkan validasi silang dalam 3 blok untuk sesi pelatihan model 540 (3 x 1 x 5 x 6 x 6 x 1), yang memberikan 1620 sesi pelatihan model. Dan sekarang, setelah kami menggunakan RandomizedSearchCV dan GridSearchCV, kita dapat beralih ke atribut best_params_untuk mencari tahu nilai hyperparameters mana yang memungkinkan model bekerja paling baik dengan kumpulan data yang diteliti (nilai-nilai ini dapat dilihat di bagian bawah blok kode sebelumnya) . Parameter ini digunakan untuk membuat model nomor 3.9. Evaluasi kualitas model pada data verifikasi
Sekarang Anda dapat mengevaluasi model yang dibuat pada data verifikasi. Yaitu, kita berbicara tentang ketiga model yang dijelaskan di awal materi.Lihat model-model ini:y_pred = rfc.predict(X_test_scaled)
y_pred_pca = rfc.predict(X_test_scaled_pca)
y_pred_gs = gs.best_estimator_.predict(X_test_scaled_pca)
Buat matriks kesalahan untuk model dan cari tahu seberapa baik masing-masing dari mereka dapat memprediksi kanker payudara:from sklearn.metrics import confusion_matrix
conf_matrix_baseline = pd.DataFrame(confusion_matrix(y_test, y_pred), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_baseline_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_pca), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_tuned_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_gs), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
display(conf_matrix_baseline)
display('Baseline Random Forest recall score', recall_score(y_test, y_pred))
display(conf_matrix_baseline_pca)
display('Baseline Random Forest With PCA recall score', recall_score(y_test, y_pred_pca))
display(conf_matrix_tuned_pca)
display('Hyperparameter Tuned Random Forest With PCA Reduced Dimensionality recall score', recall_score(y_test, y_pred_gs))
Hasil karya tiga modelDi sini metrik "kelengkapan" (recall) dievaluasi. Faktanya adalah bahwa kita sedang berhadapan dengan diagnosis kanker. Karena itu, kami sangat tertarik untuk meminimalkan ramalan negatif palsu yang dikeluarkan oleh model.Dengan ini, kita dapat menyimpulkan bahwa model RF dasar memberikan hasil terbaik. Tingkat kelengkapannya adalah 94,97%. Dalam dataset uji, ada catatan 179 pasien yang menderita kanker. Model menemukan 170 di antaranya.Ringkasan
Penelitian ini memberikan pengamatan penting. Terkadang model RF, yang menggunakan metode komponen utama dan optimisasi skala besar dari hiperparameter, mungkin tidak berfungsi sebaik model paling umum dengan pengaturan standar. Tapi ini bukan alasan untuk membatasi diri hanya pada model yang paling sederhana. Tanpa mencoba model yang berbeda, tidak mungkin untuk mengatakan mana yang akan menunjukkan hasil terbaik. Dan dalam kasus model yang digunakan untuk memprediksi keberadaan kanker pada pasien, kita dapat mengatakan bahwa semakin baik model - semakin banyak nyawa dapat diselamatkan.Pembaca yang budiman! Tugas apa yang Anda selesaikan menggunakan metode pembelajaran mesin?