Dans cet article, je vais parler de quelques astuces simples qui sont utiles lorsque vous travaillez avec des données qui ne tiennent pas dans la machine locale, mais qui sont encore trop petites pour être appelées Large. Suivant l'analogie de la langue anglaise (grande mais pas grande), nous appellerons ces données épaisses. Nous parlons de tailles d'unités et de dizaines de gigaoctets.[Clause de non-responsabilité] Si vous aimez SQL, tout ce qui est écrit ci-dessous peut vous faire ressentir des émotions négatives, très probablement, aux Pays-Bas il y a 49262 Tesla, 427 d'entre eux sont des taxis, mieux vaut ne pas lire davantage [/ Disclaimer]. Le point de départ était un article sur le moyeu avec une description d'un ensemble de données intéressant - une liste complète des véhicules immatriculés aux Pays-Bas, 14 millions de lignes, des tracteurs routiers aux vélos électriques à des vitesses supérieures à 25 km / h.L'ensemble est intéressant, prend 7 Go, vous pouvez le télécharger sur le site internet de l'organisation responsable.La tentative de conduire les données telles qu'elles se trouvent dans les pandas pour les filtrer et les nettoyer s'est soldée par un fiasco (messieurs les hussards SQL, j'ai prévenu!). Les pandas sont tombés d'un manque de mémoire sur le bureau avec 8 Go. Avec un peu d'effusion de sang, la question peut être résolue si vous vous souvenez que les pandas peuvent lire les fichiers csv en morceaux de taille modérée. La taille des fragments en lignes est déterminée par le paramètre chunksize.Pour illustrer le travail, nous allons écrire une fonction simple qui fait une demande et détermine combien de voitures Tesla sont au total et quelle proportion d'entre elles travaillent dans des taxis. Sans astuces à lecture fragmentaire, une telle requête mange d'abord toute la mémoire, puis elle souffre longtemps, et à la fin la rampe s'effondre.Avec la lecture de fragments, notre fonction ressemblera à ceci:
Le point de départ était un article sur le moyeu avec une description d'un ensemble de données intéressant - une liste complète des véhicules immatriculés aux Pays-Bas, 14 millions de lignes, des tracteurs routiers aux vélos électriques à des vitesses supérieures à 25 km / h.L'ensemble est intéressant, prend 7 Go, vous pouvez le télécharger sur le site internet de l'organisation responsable.La tentative de conduire les données telles qu'elles se trouvent dans les pandas pour les filtrer et les nettoyer s'est soldée par un fiasco (messieurs les hussards SQL, j'ai prévenu!). Les pandas sont tombés d'un manque de mémoire sur le bureau avec 8 Go. Avec un peu d'effusion de sang, la question peut être résolue si vous vous souvenez que les pandas peuvent lire les fichiers csv en morceaux de taille modérée. La taille des fragments en lignes est déterminée par le paramètre chunksize.Pour illustrer le travail, nous allons écrire une fonction simple qui fait une demande et détermine combien de voitures Tesla sont au total et quelle proportion d'entre elles travaillent dans des taxis. Sans astuces à lecture fragmentaire, une telle requête mange d'abord toute la mémoire, puis elle souffre longtemps, et à la fin la rampe s'effondre.Avec la lecture de fragments, notre fonction ressemblera à ceci:def pandas_chunky_query():
print('reading csv file with pandas in chunks')
filtered_chunk_list=[]
for chunk in pd.read_csv('C:\Open_data\RDW_full.CSV', chunksize=1E+6):
filtered_chunk=chunk[chunk['Merk'].isin(['TESLA MOTORS','TESLA'])]
filtered_chunk_list.append(filtered_chunk)
model_df = pd.concat(filtered_chunk_list)
print(model_df['Taxi indicator'].value_counts())
En spécifiant un million de lignes parfaitement raisonnable, vous pouvez exécuter la requête en 1:46 et utiliser 1965 M de mémoire à son apogée. Tous les chiffres pour un bureau stupide avec quelque chose d'ancien, huit cœurs d'environ 8 Go de mémoire et sous le septième Windows.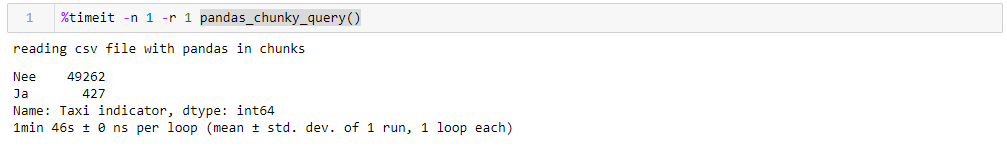
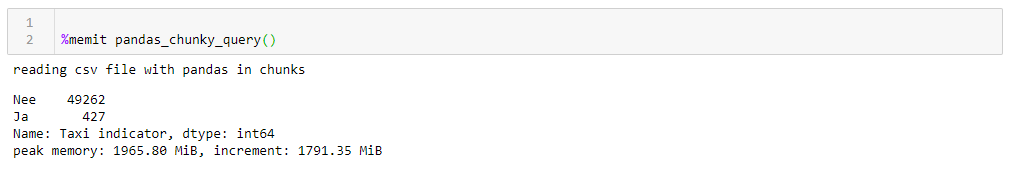 Si vous changez de taille de bloc, la consommation de mémoire maximale la suit littéralement, le temps d'exécution ne change pas beaucoup. Pour les lignes de 0,5 M, la demande prend 1:44 et 1063 Mo, pour 2M 1:53 et 3762 Mo.La vitesse n'est pas très agréable, d'autant plus que la lecture du fichier en fragments vous oblige à écrire adapté à cette fonction, en travaillant avec des listes de fragments qui doivent ensuite être collectées dans un bloc de données. De plus, le format csv lui-même n'est pas très satisfaisant, ce qui prend beaucoup de place et se lit lentement.Comme nous pouvons conduire les données dans une rampe, un format Apachev beaucoup plus compact peut être utilisé pour le stockageparquet où il y a compression, et, grâce au schéma de données, il est beaucoup plus rapide à lire quand il est lu. Et la rampe est tout à fait capable de travailler avec lui. Seulement maintenant, je ne peux pas les lire en fragments. Que faire?- Amusons-nous, prenons l'
Si vous changez de taille de bloc, la consommation de mémoire maximale la suit littéralement, le temps d'exécution ne change pas beaucoup. Pour les lignes de 0,5 M, la demande prend 1:44 et 1063 Mo, pour 2M 1:53 et 3762 Mo.La vitesse n'est pas très agréable, d'autant plus que la lecture du fichier en fragments vous oblige à écrire adapté à cette fonction, en travaillant avec des listes de fragments qui doivent ensuite être collectées dans un bloc de données. De plus, le format csv lui-même n'est pas très satisfaisant, ce qui prend beaucoup de place et se lit lentement.Comme nous pouvons conduire les données dans une rampe, un format Apachev beaucoup plus compact peut être utilisé pour le stockageparquet où il y a compression, et, grâce au schéma de données, il est beaucoup plus rapide à lire quand il est lu. Et la rampe est tout à fait capable de travailler avec lui. Seulement maintenant, je ne peux pas les lire en fragments. Que faire?- Amusons-nous, prenons l' accordéon du bouton Dask et accélérons!Dask! Un substitut à la rampe prête à l'emploi, capable de lire de gros fichiers, capable de travailler en parallèle sur plusieurs cœurs et d'utiliser des calculs paresseux. À ma grande surprise au sujet de Dask sur Habré, il n'y a que 4 publications .Donc, nous prenons le dask, nous y introduisons le csv d'origine et avec une conversion minimale, nous le conduisons au sol. Lors de la lecture, dask jure de l'ambiguïté des types de données dans certaines colonnes, nous les définissons donc explicitement (pour plus de clarté, la même chose a été faite pour la rampe, le temps de fonctionnement est plus élevé en tenant compte de ce facteur, le dictionnaire avec dtypes est coupé pour la clarté de toutes les requêtes), le reste est pour lui-même . De plus, pour vérification, nous apportons de petites améliorations au revêtement de sol, à savoir, nous essayons de réduire les types de données aux plus compacts, de remplacer une paire de colonnes par du texte oui / non par des booléennes et de convertir d'autres données en types les plus économiques (pour le nombre de cylindres du moteur, uint8 est certainement suffisant). Nous enregistrons séparément le revêtement de sol optimisé et voyons ce que nous obtenons.La première chose qui plaît quand on travaille avec Dask, c'est que nous n'avons pas besoin d'écrire quoi que ce soit de superflu simplement parce que nous avons des données épaisses. Si vous ne faites pas attention au fait que le fichier est importé et non à la rampe, tout ressemble au traitement d'un fichier avec cent lignes dans la rampe (plus quelques sifflets décoratifs pour le profilage).def dask_query():
print('reading CSV file with dask')
with ProgressBar(), ResourceProfiler(dt=0.25) as rprof:
raw_data=dd.read_csv('C:\Open_data\RDW_full.CSV')
model_df=raw_data[raw_data['Merk'].isin(['TESLA MOTORS','TESLA'])]
print(model_df['Taxi indicator'].value_counts().compute())
rprof.visualize()
Comparez maintenant l'impact du fichier source sur les performances lorsque vous travaillez avec dasko. Nous lisons d'abord le même fichier csv que lorsque vous travaillez avec la rampe. La même chose environ deux minutes et deux gigaoctets de mémoire (1:38 2096 Mo). Il semblerait, cela valait-il la peine de s'embrasser dans les buissons?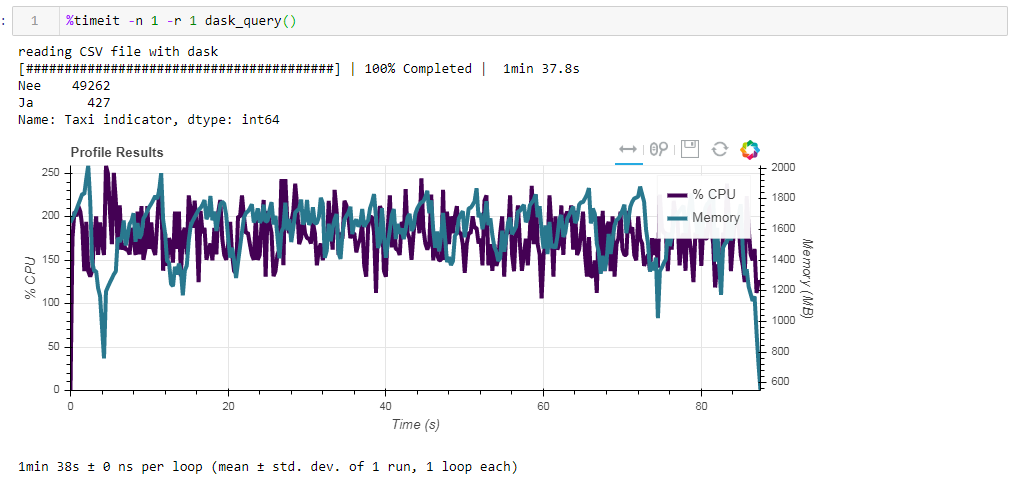 Maintenant, alimentez le dossier de parquet non optimisé. La demande a été traitée en 54 secondes environ, consommant 1 388 Mo de mémoire, et le fichier lui-même pour la demande est maintenant 10 fois plus petit (environ 700 Mo). Ici, les bonus sont déjà visibles de manière convexe. L'utilisation de centaines de pour cent du processeur est une parallélisation entre plusieurs cœurs.
Maintenant, alimentez le dossier de parquet non optimisé. La demande a été traitée en 54 secondes environ, consommant 1 388 Mo de mémoire, et le fichier lui-même pour la demande est maintenant 10 fois plus petit (environ 700 Mo). Ici, les bonus sont déjà visibles de manière convexe. L'utilisation de centaines de pour cent du processeur est une parallélisation entre plusieurs cœurs.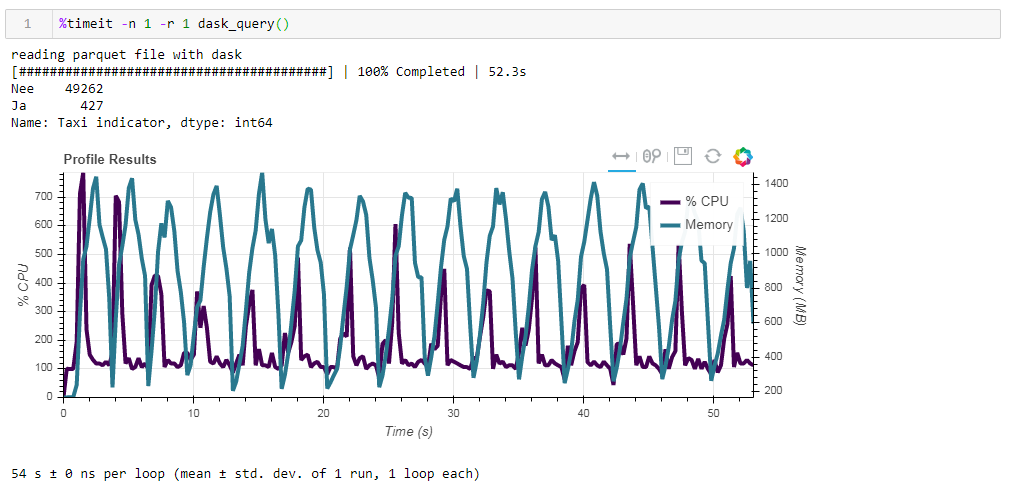 Le parquet précédemment optimisé avec des types de données légèrement modifiés sous forme compressée ne fait que 1 Mo de moins, ce qui signifie que sans indices, tout est compressé assez efficacement. L'augmentation de la productivité n'est pas non plus particulièrement significative. La demande prend les mêmes 53 secondes et consomme un peu moins de mémoire - 1332 Mo.Sur la base des résultats de nos exercices, nous pouvons dire ce qui suit:
Le parquet précédemment optimisé avec des types de données légèrement modifiés sous forme compressée ne fait que 1 Mo de moins, ce qui signifie que sans indices, tout est compressé assez efficacement. L'augmentation de la productivité n'est pas non plus particulièrement significative. La demande prend les mêmes 53 secondes et consomme un peu moins de mémoire - 1332 Mo.Sur la base des résultats de nos exercices, nous pouvons dire ce qui suit:- Si vos données sont «grasses» et que vous êtes habitué à une rampe - la taille de bloc aidera la rampe à digérer ce volume, la vitesse sera supportable.
- Si vous voulez augmenter la vitesse, économiser de l'espace pendant le stockage et que vous ne vous retenez pas en utilisant simplement une rampe, le crépuscule avec du parquet est une bonne combinaison.
Enfin, sur l'informatique paresseuse. L'une des caractéristiques de la tâche est qu'elle utilise des calculs paresseux, c'est-à-dire que les calculs ne sont pas effectués immédiatement car ils se trouvent dans le code, mais lorsqu'ils sont vraiment nécessaires ou lorsque vous l'avez explicitement demandé à l'aide de la méthode de calcul. Par exemple, dans notre fonction, dask ne lit pas toutes les données en mémoire lorsque nous indiquons de lire le fichier. Il les lit plus tard, et uniquement les colonnes qui se rapportent à la demande.Ceci est facilement visible dans l'exemple suivant. Nous prenons un fichier pré-filtré dans lequel nous n'avons laissé que 12 colonnes sur les 64 initiales, le parquet compressé prend 203 Mo. Si vous y exécutez notre requête régulière, elle s'exécutera en 8,8 secondes et l'utilisation maximale de la mémoire sera d'environ 300 Mo, ce qui correspond à un dixième du fichier compressé si vous le dépassez dans un simple csv.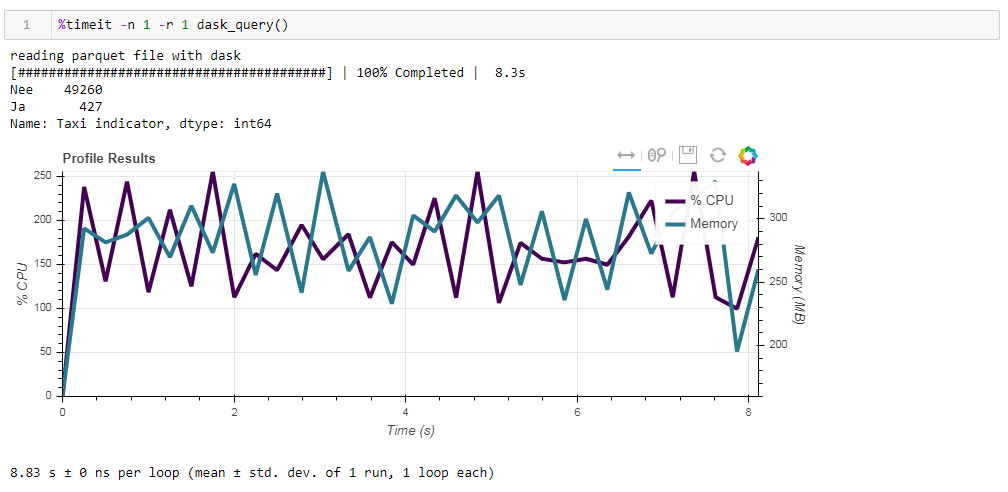 Si nous vous demandons explicitement de lire le fichier, puis d'exécuter la demande, la consommation de mémoire sera presque 10 fois plus élevée. Nous modifions légèrement notre fonction en lisant explicitement le fichier:
Si nous vous demandons explicitement de lire le fichier, puis d'exécuter la demande, la consommation de mémoire sera presque 10 fois plus élevée. Nous modifions légèrement notre fonction en lisant explicitement le fichier:def dask_query():
print('reading parquet file with dask')
with ProgressBar(), ResourceProfiler(dt=0.25) as rprof:
raw_data=dd.read_parquet('C:\Open_data\RDW_filtered.parquet' ).compute()
model_df=raw_data[raw_data['Merk'].isin(['TESLA MOTORS','TESLA'])]
print(model_df['Taxi indicator'].value_counts())
rprof.visualize()
Et voici ce que nous obtenons, 10,5 secondes et 3568 Mo de mémoire (!)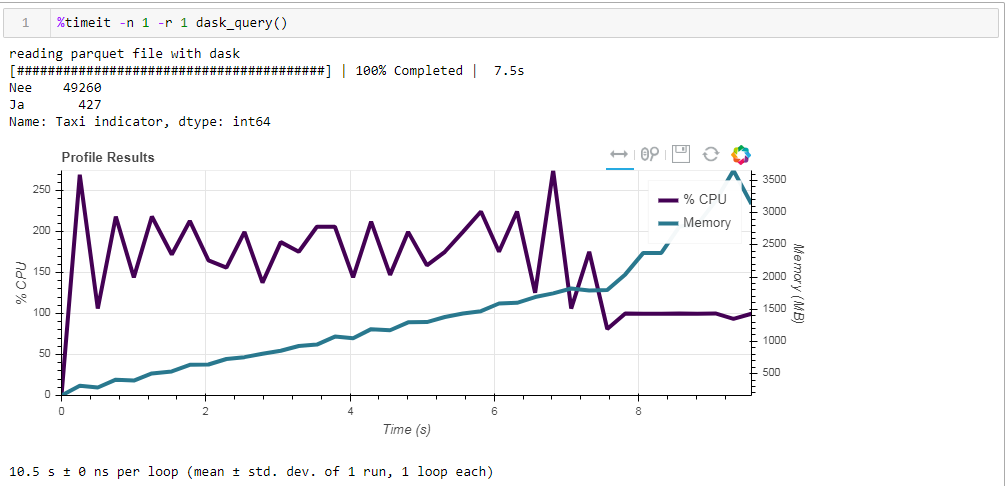 Une fois de plus, nous sommes convaincus que le dask - il est compétent pour faire face à ses tâches lui-même, et encore une fois y grimper avec la micro-gestion n'a pas beaucoup de sens.
Une fois de plus, nous sommes convaincus que le dask - il est compétent pour faire face à ses tâches lui-même, et encore une fois y grimper avec la micro-gestion n'a pas beaucoup de sens.