Les spécialistes du traitement et de l'analyse des données disposent de nombreux outils pour créer des modèles de classification. L'une des méthodes les plus populaires et les plus fiables pour développer de tels modèles est d'utiliser l'algorithme Random Forest (RF). Afin d'essayer d'améliorer les performances d'un modèle construit à l'aide de l'algorithme RF , vous pouvez utiliser l'optimisation de l'hyperparamètre du modèle ( Hyperparameter Tuning , HT). De plus, il existe une approche répandue selon laquelle les données, avant d'être transférées au modèle, sont traitées à l'aide de l' analyse en composantes principales , PCA). Mais vaut-il la peine d'utiliser? Le but principal de l'algorithme RF n'est-il pas d'aider l'analyste à interpréter l'importance des traits?Oui, l'utilisation de l'algorithme PCA peut conduire à une légère complication de l'interprétation de chaque «caractéristique» dans l'analyse de «l'importance des caractéristiques» du modèle RF. Cependant, l'algorithme PCA réduit la dimension de l'espace des fonctionnalités, ce qui peut entraîner une diminution du nombre de fonctionnalités qui doivent être traitées par le modèle RF. Veuillez noter que le volume des calculs est l'un des principaux inconvénients de l'algorithme de forêt aléatoire (c'est-à-dire qu'il peut prendre beaucoup de temps pour terminer le modèle). L'application de l'algorithme PCA peut être une partie très importante de la modélisation, en particulier dans les cas où ils fonctionnent avec des centaines voire des milliers de fonctionnalités. Par conséquent, si la chose la plus importante est de simplement créer le modèle le plus efficace, et en même temps vous pouvez sacrifier la précision de la détermination de l'importance des attributs, alors l'ACP peut valoir la peine d'être essayée.Maintenant au point. Nous travaillerons avec un ensemble de données sur le cancer du sein - Scikit-learn «cancer du sein» . Nous allons créer trois modèles et comparer leur efficacité. À savoir, nous parlons des modèles suivants:
, PCA). Mais vaut-il la peine d'utiliser? Le but principal de l'algorithme RF n'est-il pas d'aider l'analyste à interpréter l'importance des traits?Oui, l'utilisation de l'algorithme PCA peut conduire à une légère complication de l'interprétation de chaque «caractéristique» dans l'analyse de «l'importance des caractéristiques» du modèle RF. Cependant, l'algorithme PCA réduit la dimension de l'espace des fonctionnalités, ce qui peut entraîner une diminution du nombre de fonctionnalités qui doivent être traitées par le modèle RF. Veuillez noter que le volume des calculs est l'un des principaux inconvénients de l'algorithme de forêt aléatoire (c'est-à-dire qu'il peut prendre beaucoup de temps pour terminer le modèle). L'application de l'algorithme PCA peut être une partie très importante de la modélisation, en particulier dans les cas où ils fonctionnent avec des centaines voire des milliers de fonctionnalités. Par conséquent, si la chose la plus importante est de simplement créer le modèle le plus efficace, et en même temps vous pouvez sacrifier la précision de la détermination de l'importance des attributs, alors l'ACP peut valoir la peine d'être essayée.Maintenant au point. Nous travaillerons avec un ensemble de données sur le cancer du sein - Scikit-learn «cancer du sein» . Nous allons créer trois modèles et comparer leur efficacité. À savoir, nous parlons des modèles suivants:- Le modèle de base basé sur l'algorithme RF (nous abrégerons ce modèle RF).
- Le même modèle que le n ° 1, mais dans lequel une réduction de la dimension de l'espace caractéristique est appliquée en utilisant la méthode des composants principaux (RF + PCA).
- Le même modèle que le n ° 2, mais construit en utilisant l'optimisation hyperparamétrique (RF + PCA + HT).
1. Importer des données
Pour commencer, chargez les données et créez une trame de données Pandas. Étant donné que nous utilisons un ensemble de données «jouet» pré-autorisé de Scikit-learn, nous pouvons déjà commencer le processus de modélisation. Mais même lorsque vous utilisez de telles données, il est recommandé de toujours commencer à travailler en effectuant une analyse préliminaire des données à l'aide des commandes suivantes appliquées au bloc de données ( df):df.head() - pour jeter un oeil à la nouvelle trame de données et voir si elle ressemble à celle attendue.df.info()- pour découvrir les caractéristiques des types de données et le contenu des colonnes. Il peut être nécessaire d'effectuer une conversion de type de données avant de continuer.df.isna()- pour vous assurer qu'il n'y a pas de valeurs dans les données NaN. Les valeurs correspondantes, le cas échéant, peuvent devoir être traitées d'une manière ou d'une autre, ou, si nécessaire, il peut être nécessaire de supprimer des lignes entières du bloc de données.df.describe() - connaître les valeurs minimales, maximales et moyennes des indicateurs dans les colonnes, connaître les indicateurs du carré moyen et de l'écart probable dans les colonnes.
Dans notre jeu de données, une colonne cancer(cancer) est la variable cible dont nous voulons prédire la valeur à l'aide du modèle. 0signifie «pas de maladie». 1- "la présence de la maladie".import pandas as pd
from sklearn.datasets import load_breast_cancer
columns = ['mean radius', 'mean texture', 'mean perimeter', 'mean area', 'mean smoothness', 'mean compactness', 'mean concavity', 'mean concave points', 'mean symmetry', 'mean fractal dimension', 'radius error', 'texture error', 'perimeter error', 'area error', 'smoothness error', 'compactness error', 'concavity error', 'concave points error', 'symmetry error', 'fractal dimension error', 'worst radius', 'worst texture', 'worst perimeter', 'worst area', 'worst smoothness', 'worst compactness', 'worst concavity', 'worst concave points', 'worst symmetry', 'worst fractal dimension']
dataset = load_breast_cancer()
data = pd.DataFrame(dataset['data'], columns=columns)
data['cancer'] = dataset['target']
display(data.head())
display(data.info())
display(data.isna().sum())
display(data.describe())
. . , cancer, , . 0 « ». 1 — « »2.
Maintenant, divisez les données à l'aide de la fonction Scikit-learn train_test_split. Nous voulons donner au modèle autant de données d'entraînement que possible. Cependant, nous devons disposer de suffisamment de données pour tester le modèle. En général, nous pouvons dire que, à mesure que le nombre de lignes de l'ensemble de données augmente, la quantité de données pouvant être considérée comme éducative augmente également.Par exemple, s'il y a des millions de lignes, vous pouvez diviser l'ensemble en mettant en surbrillance 90% des lignes pour les données d'apprentissage et 10% pour les données de test. Mais l'ensemble de données de test ne contient que 569 lignes. Et ce n'est pas tant pour la formation et le test du modèle. Par conséquent, afin d'être juste par rapport aux données de formation et de vérification, nous diviserons l'ensemble en deux parties égales - 50% - données de formation et 50% - données de vérification. Nous installonsstratify=y pour garantir que les ensembles de données de formation et de test ont le même rapport de 0 et 1 que l'ensemble de données d'origine.from sklearn.model_selection import train_test_split
X = data.drop('cancer', axis=1)
y = data['cancer']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.50, random_state = 2020, stratify=y)
3. Mise à l'échelle des données
Avant de procéder à la modélisation, vous devez «centrer» et «normaliser» les données en les mettant à l' échelle . La mise à l'échelle est effectuée du fait que différentes quantités sont exprimées dans différentes unités. Cette procédure vous permet d'organiser un «combat loyal» entre les signes pour déterminer leur importance. De plus, nous convertissons y_trainle type de données Pandas Seriesen tableau NumPy afin que plus tard le modèle puisse fonctionner avec les cibles correspondantes.import numpy as np
from sklearn.preprocessing import StandardScaler
ss = StandardScaler()
X_train_scaled = ss.fit_transform(X_train)
X_test_scaled = ss.transform(X_test)
y_train = np.array(y_train)
4. Formation du modèle de base (modèle n ° 1, RF)
Créez maintenant le modèle numéro 1. Dans ce document, nous rappelons que seul l'algorithme Random Forest est utilisé. Il utilise toutes les fonctionnalités et est configuré en utilisant les valeurs par défaut (des détails sur ces paramètres peuvent être trouvés dans la documentation de sklearn.ensemble.RandomForestClassifier ). Initialisez le modèle. Après cela, nous la formerons sur les données à l'échelle. La précision du modèle peut être mesurée sur les données d'entraînement:from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import recall_score
rfc = RandomForestClassifier()
rfc.fit(X_train_scaled, y_train)
display(rfc.score(X_train_scaled, y_train))
Si nous voulons savoir quelles caractéristiques sont les plus importantes pour le modèle RF dans la prédiction du cancer du sein, nous pouvons visualiser et quantifier les indicateurs de l'importance des signes en faisant référence à l'attribut feature_importances_:feats = {}
for feature, importance in zip(data.columns, rfc_1.feature_importances_):
feats[feature] = importance
importances = pd.DataFrame.from_dict(feats, orient='index').rename(columns={0: 'Gini-Importance'})
importances = importances.sort_values(by='Gini-Importance', ascending=False)
importances = importances.reset_index()
importances = importances.rename(columns={'index': 'Features'})
sns.set(font_scale = 5)
sns.set(style="whitegrid", color_codes=True, font_scale = 1.7)
fig, ax = plt.subplots()
fig.set_size_inches(30,15)
sns.barplot(x=importances['Gini-Importance'], y=importances['Features'], data=importances, color='skyblue')
plt.xlabel('Importance', fontsize=25, weight = 'bold')
plt.ylabel('Features', fontsize=25, weight = 'bold')
plt.title('Feature Importance', fontsize=25, weight = 'bold')
display(plt.show())
display(importances)
Visualisation de «l'importance» des signesIndicateurs de signification5. La méthode des principaux composants
Voyons maintenant comment nous pouvons améliorer le modèle RF de base. En utilisant la technique de réduction de la dimension de l'espace d'entité, il est possible de présenter l'ensemble de données initial à travers moins de variables et en même temps de réduire la quantité de ressources informatiques nécessaires pour assurer le fonctionnement du modèle. À l'aide de l'ACP, vous pouvez étudier la variance cumulée de l'échantillon de ces caractéristiques afin de comprendre quelles caractéristiques expliquent la plupart de la variance dans les données.Nous initialisons l'objet PCA ( pca_test), indiquant le nombre de composants (fonctionnalités) à prendre en compte. Nous avons fixé cet indicateur à 30 afin de voir la variance expliquée de tous les composants générés avant de décider du nombre de composants dont nous avons besoin. Ensuite, nous transférons aux pca_testdonnées mises à l' échelleX_trainen utilisant la méthode pca_test.fit(). Après cela, nous visualisons les données.import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.decomposition import PCA
pca_test = PCA(n_components=30)
pca_test.fit(X_train_scaled)
sns.set(style='whitegrid')
plt.plot(np.cumsum(pca_test.explained_variance_ratio_))
plt.xlabel('number of components')
plt.ylabel('cumulative explained variance')
plt.axvline(linewidth=4, color='r', linestyle = '--', x=10, ymin=0, ymax=1)
display(plt.show())
evr = pca_test.explained_variance_ratio_
cvr = np.cumsum(pca_test.explained_variance_ratio_)
pca_df = pd.DataFrame()
pca_df['Cumulative Variance Ratio'] = cvr
pca_df['Explained Variance Ratio'] = evr
display(pca_df.head(10))
Après que le nombre de composants utilisés dépasse 10, l'augmentation de leur nombre n'augmente pas considérablement la variance expliquéeCe bloc de données contient des indicateurs tels que le ratio de variance cumulée (taille cumulée de la variance expliquée des données) et le ratio de variance expliquée (contribution de chaque composant au volume total de la variance expliquée).Si vous regardez le bloc de données ci-dessus, il s'avère que l'utilisation de l'ACP pour passer de 30 variables à 10 aux composants permet d'expliquer 95% de la dispersion des données. Les 20 autres composantes représentent moins de 5% de la variance, ce qui signifie que nous pouvons les refuser. Suivant cette logique, nous utilisons le PCA pour réduire le nombre de composants de 30 à 10 pourX_trainetX_test. Nous écrivons ces ensembles de données de «dimension réduite» créés artificiellement dansX_train_scaled_pcaet endedansX_test_scaled_pca.pca = PCA(n_components=10)
pca.fit(X_train_scaled)
X_train_scaled_pca = pca.transform(X_train_scaled)
X_test_scaled_pca = pca.transform(X_test_scaled)
Chaque composante est une combinaison linéaire de variables sources avec des «pondérations» correspondantes. Nous pouvons voir ces «poids» pour chaque composant en créant un bloc de données.pca_dims = []
for x in range(0, len(pca_df)):
pca_dims.append('PCA Component {}'.format(x))
pca_test_df = pd.DataFrame(pca_test.components_, columns=columns, index=pca_dims)
pca_test_df.head(10).T
Cadre de données d'informations sur les composants6. Formation au modèle RF de base après application de la méthode des composants principaux aux données (modèle n ° 2, RF + PCA)
Maintenant , nous pouvons passer à une autre base de données modèle RF X_train_scaled_pcaet y_trainet peut savoir de savoir s'il y a une amélioration de la précision des prévisions émises par le modèle.rfc = RandomForestClassifier()
rfc.fit(X_train_scaled_pca, y_train)
display(rfc.score(X_train_scaled_pca, y_train))
Les modèles se comparent ci-dessous.7. Optimisation des hyperparamètres. Round 1: RandomizedSearchCV
Après avoir traité les données en utilisant la méthode du composant principal, vous pouvez essayer d'utiliser l' optimisation des hyperparamètres du modèle afin d'améliorer la qualité des prédictions produites par le modèle RF. Les hyperparamètres peuvent être considérés comme quelque chose comme des «paramètres» du modèle. Les paramètres parfaits pour un ensemble de données ne fonctionneront pas pour un autre - c'est pourquoi vous devez les optimiser.Vous pouvez commencer avec l'algorithme RandomizedSearchCV, qui vous permet d'explorer assez approximativement un large éventail de valeurs. Des descriptions de tous les hyperparamètres pour les modèles RF peuvent être trouvées ici .Au cours du travail, nous générons une entité param_distqui contient, pour chaque hyperparamètre, une plage de valeurs à tester. Ensuite, nous initialisons l'objet.rsen utilisant la fonction RandomizedSearchCV(), en lui passant le modèle RF param_dist, le nombre d'itérations et le nombre de validations croisées qui doivent être effectuées.L'hyperparamètre verbosepermet de contrôler la quantité d'informations affichées par le modèle lors de son fonctionnement (comme la sortie d'informations lors de la formation du modèle). L'hyperparamètre n_jobsvous permet de spécifier le nombre de cœurs de processeur que vous devez utiliser pour garantir le fonctionnement du modèle. La définition n_jobsd'une valeur -1entraînera un modèle plus rapide, car cela utilisera tous les cœurs de processeur.Nous serons engagés dans la sélection des hyperparamètres suivants:n_estimators - le nombre d '"arbres" dans la "forêt aléatoire".max_features - le nombre d'entités pour sélectionner le fractionnement.max_depth - profondeur maximale des arbres.min_samples_split - le nombre minimum d'objets nécessaires pour qu'un nœud d'arbre se divise.min_samples_leaf - le nombre minimum d'objets dans les feuilles.bootstrap - utiliser pour construire des arbres de sous-échantillons avec retour.
from sklearn.model_selection import RandomizedSearchCV
n_estimators = [int(x) for x in np.linspace(start = 100, stop = 1000, num = 10)]
max_features = ['log2', 'sqrt']
max_depth = [int(x) for x in np.linspace(start = 1, stop = 15, num = 15)]
min_samples_split = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
min_samples_leaf = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
bootstrap = [True, False]
param_dist = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
rs = RandomizedSearchCV(rfc_2,
param_dist,
n_iter = 100,
cv = 3,
verbose = 1,
n_jobs=-1,
random_state=0)
rs.fit(X_train_scaled_pca, y_train)
rs.best_params_
Avec les valeurs des paramètres n_iter = 100et cv = 3, nous avons créé 300 modèles RF, en choisissant aléatoirement des combinaisons des hyper paramètres présentés ci-dessus. Nous pouvons nous référer à l'attribut best_params_ pour plus d'informations sur un ensemble de paramètres qui vous permet de créer le meilleur modèle. Mais à ce stade, cela ne nous donne peut-être pas les données les plus intéressantes sur les plages de paramètres qui méritent d'être explorées lors de la prochaine phase d'optimisation. Afin de découvrir dans quelle plage de valeurs il vaut la peine de poursuivre la recherche, nous pouvons facilement obtenir une trame de données contenant les résultats de l'algorithme RandomizedSearchCV.rs_df = pd.DataFrame(rs.cv_results_).sort_values('rank_test_score').reset_index(drop=True)
rs_df = rs_df.drop([
'mean_fit_time',
'std_fit_time',
'mean_score_time',
'std_score_time',
'params',
'split0_test_score',
'split1_test_score',
'split2_test_score',
'std_test_score'],
axis=1)
rs_df.head(10)
Résultats de l'algorithme RandomizedSearchCVNous allons maintenant créer des graphiques à barres sur lesquels, sur l'axe X, sont les valeurs d'hyperparamètre, et sur l'axe Y sont les valeurs moyennes affichées par les modèles. Cela permettra de comprendre quelles valeurs des hyperparamètres affichent en moyenne leurs meilleures performances.fig, axs = plt.subplots(ncols=3, nrows=2)
sns.set(style="whitegrid", color_codes=True, font_scale = 2)
fig.set_size_inches(30,25)
sns.barplot(x='param_n_estimators', y='mean_test_score', data=rs_df, ax=axs[0,0], color='lightgrey')
axs[0,0].set_ylim([.83,.93])axs[0,0].set_title(label = 'n_estimators', size=30, weight='bold')
sns.barplot(x='param_min_samples_split', y='mean_test_score', data=rs_df, ax=axs[0,1], color='coral')
axs[0,1].set_ylim([.85,.93])axs[0,1].set_title(label = 'min_samples_split', size=30, weight='bold')
sns.barplot(x='param_min_samples_leaf', y='mean_test_score', data=rs_df, ax=axs[0,2], color='lightgreen')
axs[0,2].set_ylim([.80,.93])axs[0,2].set_title(label = 'min_samples_leaf', size=30, weight='bold')
sns.barplot(x='param_max_features', y='mean_test_score', data=rs_df, ax=axs[1,0], color='wheat')
axs[1,0].set_ylim([.88,.92])axs[1,0].set_title(label = 'max_features', size=30, weight='bold')
sns.barplot(x='param_max_depth', y='mean_test_score', data=rs_df, ax=axs[1,1], color='lightpink')
axs[1,1].set_ylim([.80,.93])axs[1,1].set_title(label = 'max_depth', size=30, weight='bold')
sns.barplot(x='param_bootstrap',y='mean_test_score', data=rs_df, ax=axs[1,2], color='skyblue')
axs[1,2].set_ylim([.88,.92])
axs[1,2].set_title(label = 'bootstrap', size=30, weight='bold')
plt.show()
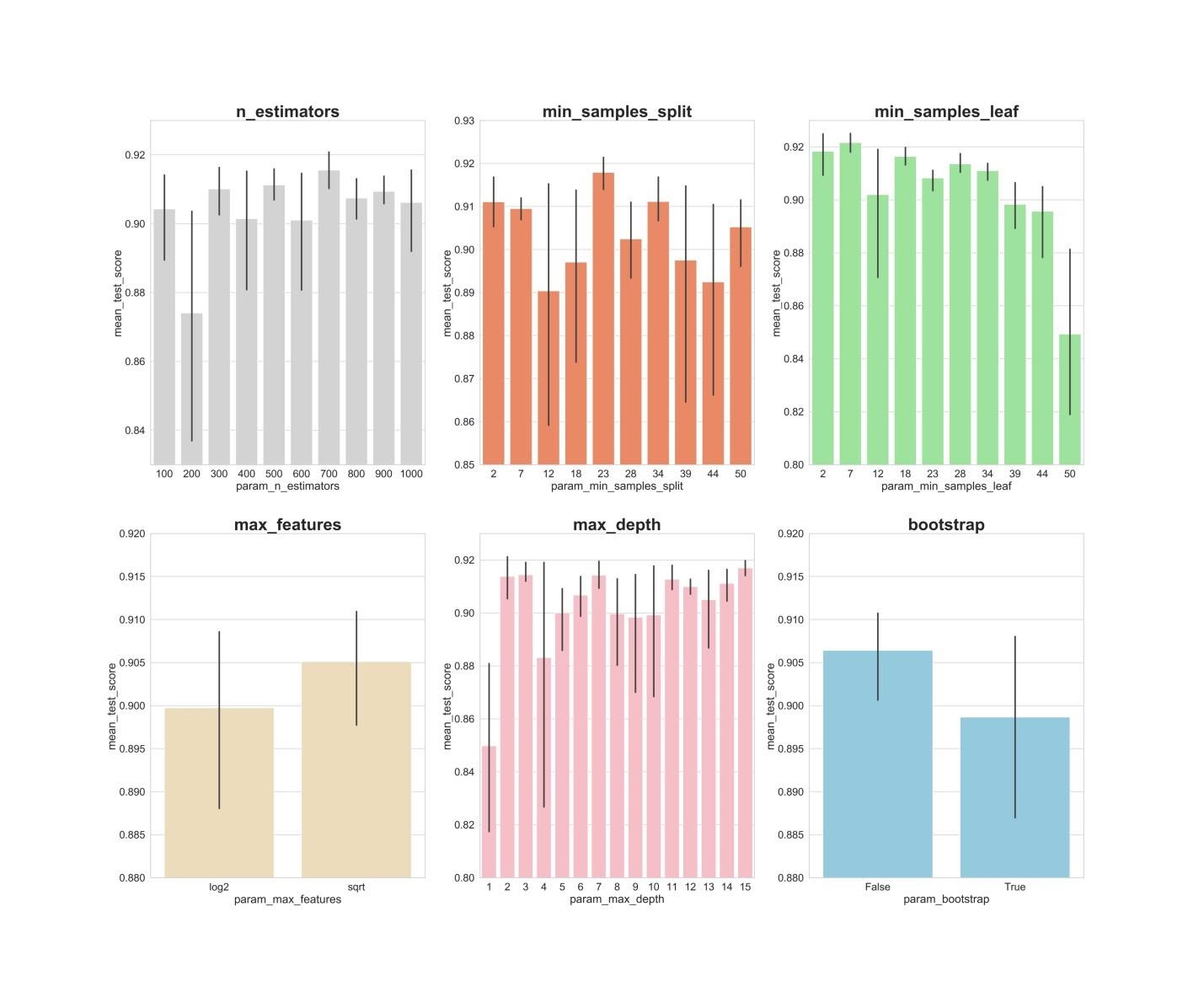
n_estimators: des valeurs de 300, 500, 700, apparemment, montrent les meilleurs résultats moyens.min_samples_split: Les petites valeurs comme 2 et 7 semblent donner les meilleurs résultats. La valeur 23 semble également bonne. Vous pouvez examiner plusieurs valeurs de cet hyperparamètre au-delà de 2, ainsi que plusieurs valeurs d'environ 23.min_samples_leaf: On a le sentiment que les petites valeurs de cet hyperparamètre donnent de meilleurs résultats. Cela signifie que nous pouvons expérimenter des valeurs comprises entre 2 et 7.max_features: l'option sqrtdonne le résultat moyen le plus élevé.max_depth: il n'y a pas de relation claire entre la valeur de l'hyperparamètre et le résultat du modèle, mais on a le sentiment que les valeurs 2, 3, 7, 11, 15 semblent bonnes.bootstrap: la valeur Falseaffiche le meilleur résultat moyen.
Maintenant, en utilisant ces résultats, nous pouvons passer au deuxième cycle d'optimisation des hyperparamètres. Cela réduira la gamme de valeurs qui nous intéresse.8. Optimisation des hyperparamètres. Tour 2: GridSearchCV (préparation finale des paramètres pour le modèle n ° 3, RF + PCA + HT)
Après avoir appliqué l'algorithme RandomizedSearchCV, nous utiliserons l'algorithme GridSearchCV pour effectuer une recherche plus précise de la meilleure combinaison d'hyperparamètres. Les mêmes hyperparamètres sont étudiés ici, mais maintenant nous appliquons une recherche plus «approfondie» pour leur meilleure combinaison. En utilisant l'algorithme GridSearchCV, chaque combinaison d'hyperparamètres est examinée. Cela nécessite beaucoup plus de ressources de calcul que l'utilisation de l'algorithme RandomizedSearchCV lorsque nous définissons indépendamment le nombre d'itérations de recherche. Par exemple, la recherche de 10 valeurs pour chacun des 6 hyperparamètres avec validation croisée en 3 blocs nécessitera 10 × 3 ou 3 000 000 de sessions de formation sur modèle. C'est pourquoi nous utilisons l'algorithme GridSearchCV après, après avoir appliqué RandomizedSearchCV, nous avons rétréci les plages de valeurs des paramètres étudiés.Donc, en utilisant ce que nous avons découvert à l'aide de RandomizedSearchCV, nous examinons les valeurs des hyperparamètres qui se sont le mieux montrées:from sklearn.model_selection import GridSearchCV
n_estimators = [300,500,700]
max_features = ['sqrt']
max_depth = [2,3,7,11,15]
min_samples_split = [2,3,4,22,23,24]
min_samples_leaf = [2,3,4,5,6,7]
bootstrap = [False]
param_grid = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
gs = GridSearchCV(rfc_2, param_grid, cv = 3, verbose = 1, n_jobs=-1)
gs.fit(X_train_scaled_pca, y_train)
rfc_3 = gs.best_estimator_
gs.best_params_
Ici, nous appliquons la validation croisée à 3 blocs pour 540 (3 x 1 x 5 x 6 x 6 x 1) sessions de formation de modèle, ce qui donne 1620 sessions de formation de modèle. Et maintenant, après avoir utilisé RandomizedSearchCV et GridSearchCV, nous pouvons nous tourner vers l'attribut best_params_pour savoir quelles valeurs d'hyperparamètres permettent au modèle de mieux fonctionner avec l'ensemble de données à l'étude (ces valeurs peuvent être vues au bas du bloc de code précédent) . Ces paramètres sont utilisés pour créer le modèle numéro 3.9. Évaluation de la qualité des modèles sur les données de vérification
Vous pouvez maintenant évaluer les modèles créés sur les données de vérification. À savoir, nous parlons de ces trois modèles décrits au tout début du matériau.Découvrez ces modèles:y_pred = rfc.predict(X_test_scaled)
y_pred_pca = rfc.predict(X_test_scaled_pca)
y_pred_gs = gs.best_estimator_.predict(X_test_scaled_pca)
Créez des matrices d'erreurs pour les modèles et découvrez dans quelle mesure chacune d'elles est capable de prédire le cancer du sein:from sklearn.metrics import confusion_matrix
conf_matrix_baseline = pd.DataFrame(confusion_matrix(y_test, y_pred), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_baseline_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_pca), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_tuned_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_gs), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
display(conf_matrix_baseline)
display('Baseline Random Forest recall score', recall_score(y_test, y_pred))
display(conf_matrix_baseline_pca)
display('Baseline Random Forest With PCA recall score', recall_score(y_test, y_pred_pca))
display(conf_matrix_tuned_pca)
display('Hyperparameter Tuned Random Forest With PCA Reduced Dimensionality recall score', recall_score(y_test, y_pred_gs))
Résultats des travaux des trois modèlesIci, la «complétude» métrique (rappel) est évaluée. Le fait est que nous avons affaire à un diagnostic de cancer. Par conséquent, nous sommes extrêmement intéressés à minimiser les prévisions fausses négatives émises par les modèles.Compte tenu de cela, nous pouvons conclure que le modèle RF de base a donné les meilleurs résultats. Son taux d'exhaustivité était de 94,97%. Dans l'ensemble de données de test, il y avait un record de 179 patients atteints de cancer. Le modèle en a trouvé 170.Sommaire
Cette étude fournit une observation importante. Parfois, le modèle RF, qui utilise la méthode des composants principaux et l'optimisation à grande échelle des hyperparamètres, peut ne pas fonctionner aussi bien que le modèle le plus ordinaire avec des paramètres standard. Mais ce n'est pas une raison pour se limiter aux modèles les plus simples. Sans essayer différents modèles, il est impossible de dire lequel affichera le meilleur résultat. Et dans le cas des modèles utilisés pour prédire la présence de cancer chez les patients, nous pouvons dire que meilleur est le modèle - plus de vies peuvent être sauvées.Chers lecteurs! Quelles tâches résolvez-vous en utilisant des méthodes d'apprentissage automatique?