Hola Habr! Hoy quiero compartir un pequeño ejemplo de cómo llevar a cabo el análisis de conglomerados. En este ejemplo, el lector no encontrará redes neuronales y otras direcciones de moda. Este ejemplo puede servir como punto de referencia para realizar un análisis de conglomerados pequeño y completo para otros datos. Cualquier persona interesada, bienvenido a cat.
Inmediatamente haga una reserva, este artículo de ninguna manera pretende ser académico en su totalidad, la unicidad de los resultados obtenidos o la integridad de la cobertura del tema. El artículo pretende demostrar los pasos básicos del análisis de conglomerados clásico, que puede usarse para un estudio simple y significativo (posiblemente antes de un estudio más detallado). Cualquier corrección, comentario y adición sobre el fondo es bienvenida.
Los datos son una muestra del consumo de alcohol por país per cápita por tipo de bebidas alcohólicas (cerveza, vino, licores, etc.) para 2010 como porcentaje del consumo de alcohol per cápita. Los datos también contienen: el consumo diario promedio de alcohol per cápita en gramos de alcohol puro y todo el consumo de alcohol per cápita (registrado + no contabilizado) (solo bebedores en litros de alcohol puro).
Al mismo tiempo, cada país pertenece condicionalmente a uno de los grupos geográficos: este, centro y oeste. La división es muy arbitraria y muy controvertida por varias razones, pero procederemos de lo que tenemos. Fuente de datos - Informe de estado global sobre alcohol y salud 2014, S. 289-364
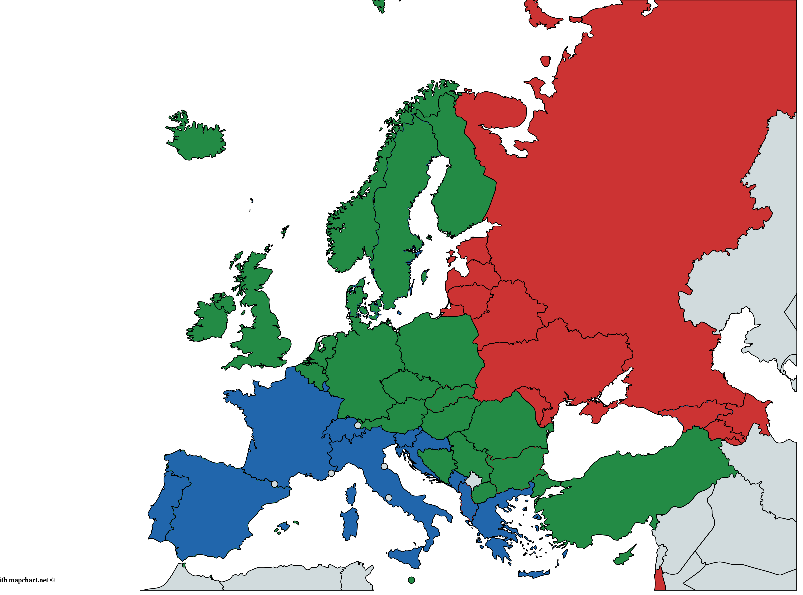
(Pintado a mano, puede haber errores, pero la idea general, creo, es comprensible)
Analisis preliminar
Conecta las bibliotecas utilizadas.
library(rgl)
library(heplots)
library(MVN)
library(klaR)
library('Morpho')
library(caret)
library(mclust)
library(ggplot2)
library(GGally)
library(plyr)
library(psych)
library(GPArotation)
library(ggpubr)
, .
#
data <- read.table("alcohol_data.csv", header=TRUE, sep=",")
#
rownames(data) <- make.names(data[,1], unique = TRUE)
# ,
data <- data[,-1]
data <- na.omit(data)
#
head(data)
summary(data)
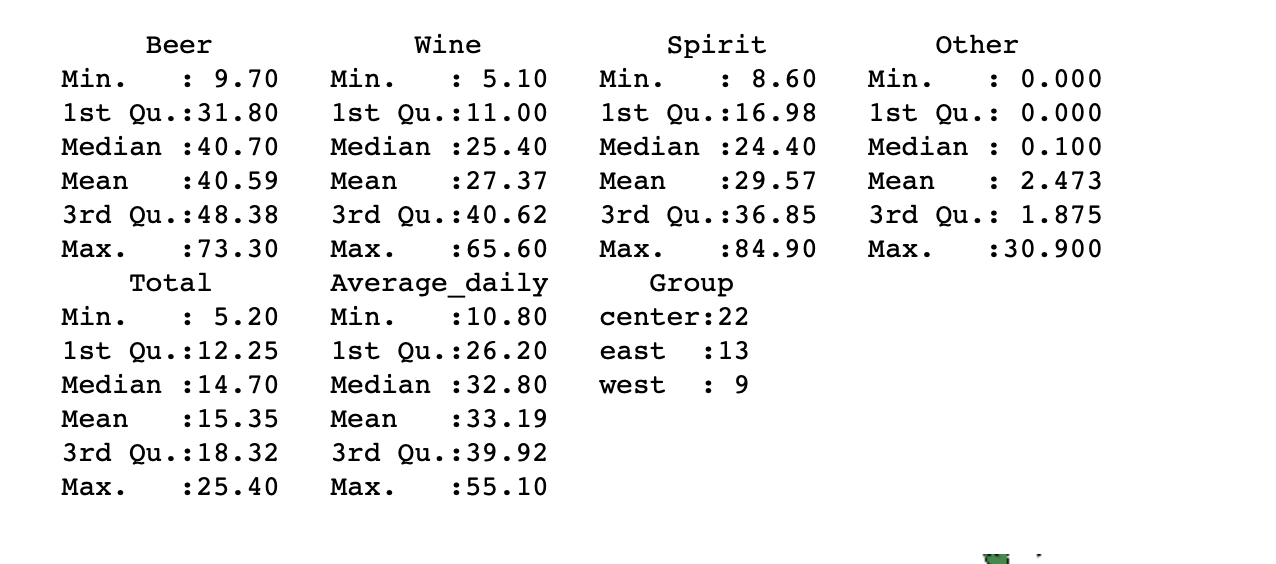
, . , Other , , , , . , , , , . , . - .
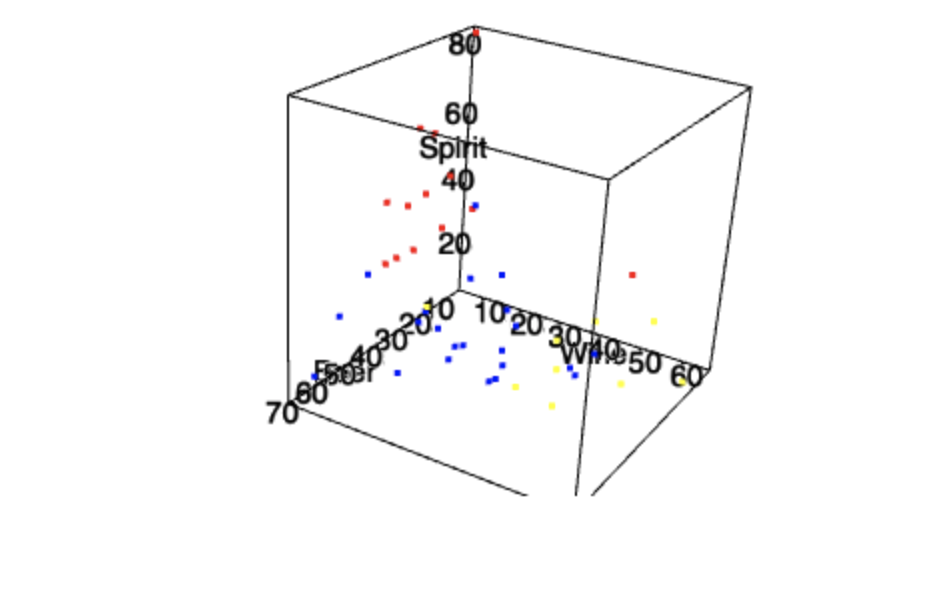
, , , .
options(rgl.useNULL=TRUE)
open3d()
mfrow3d(2,2)
levelColors <- c('west'='blue', 'east'='red', 'center'='yellow')
plot3d(data$Beer, data$Wine, data$Spirit, xlab="Beer", ylab="Wine", zlab="Spirit", col = levelColors[data$Group], size=3)
widget <- rglwidget()
widget
, . , .
ggpairs(
data,
mapping = ggplot2::aes(color = data$Group),
upper = list(continuous = wrap("cor", alpha = 0.5), combo = "box"),
lower = list(continuous = wrap("points", alpha = 0.3), combo = wrap("dot", alpha = 0.4)),
diag = list(continuous = wrap("densityDiag",alpha = 0.5)),
title = "Alcohol"
)
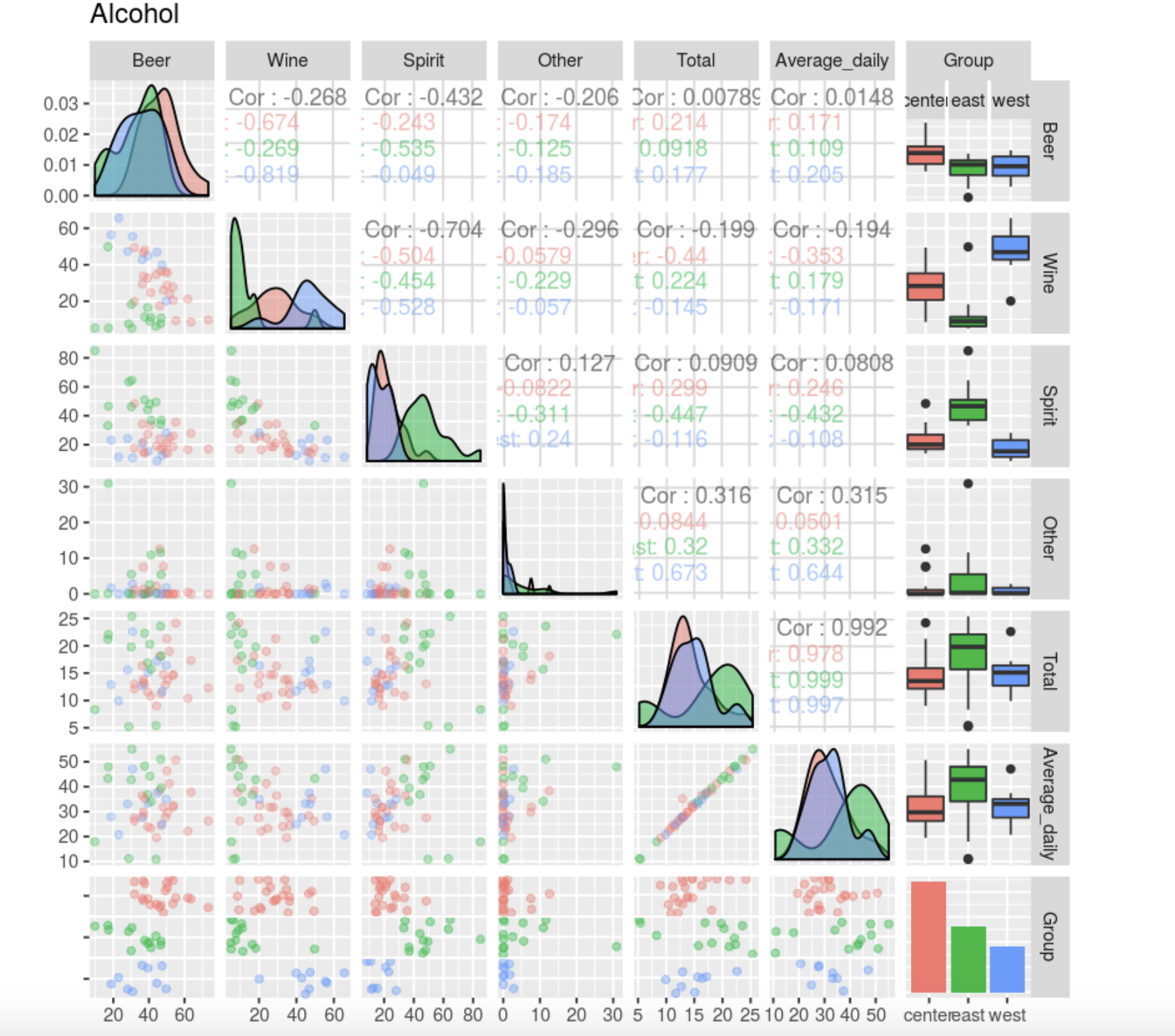
Average Total , Average.
data <- data[, -6]
, , , , . .
data[data$Wine>60,]
, , , , - , , .
data[data$Spirit>70,]
data[data$Spirit<10,]
, , .
,
split(data[,1:5],data$Group)
$center
$east
$west
ggpairs(
data,
mapping = ggplot2::aes(color = data$Group),
diag=list(continuous="bar", alpha=0.4)
)
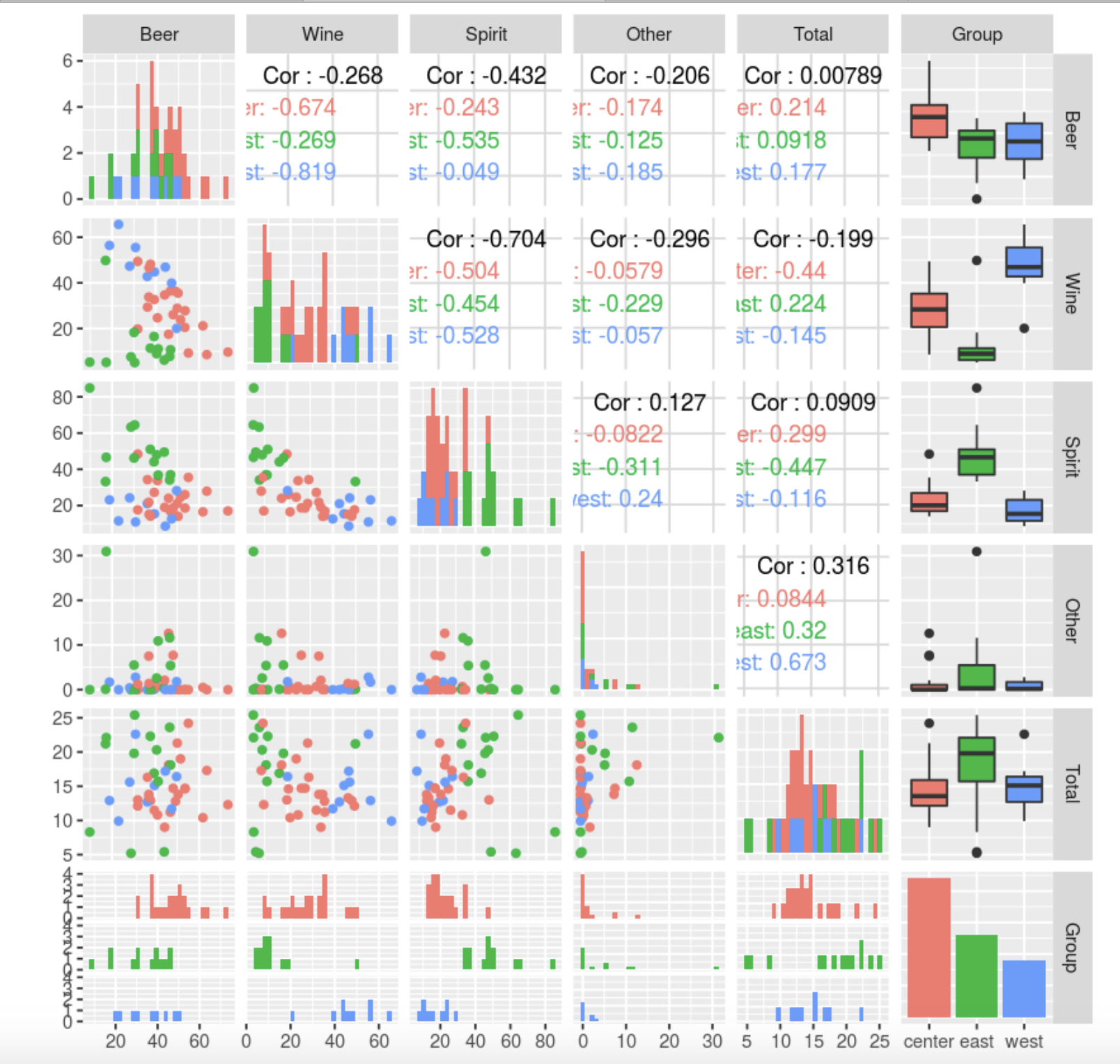
, , . Other, : , , , ( 10-12 , 45, , ). . , , , (). , , . Other .
, , — , — . , — , .
Total Other, . .
, Beer, Spirit Wine . , , , . , , , , , .
Total. , — .
data.group = data[,5]
data <- data[,-5]
data<- data[,-4]
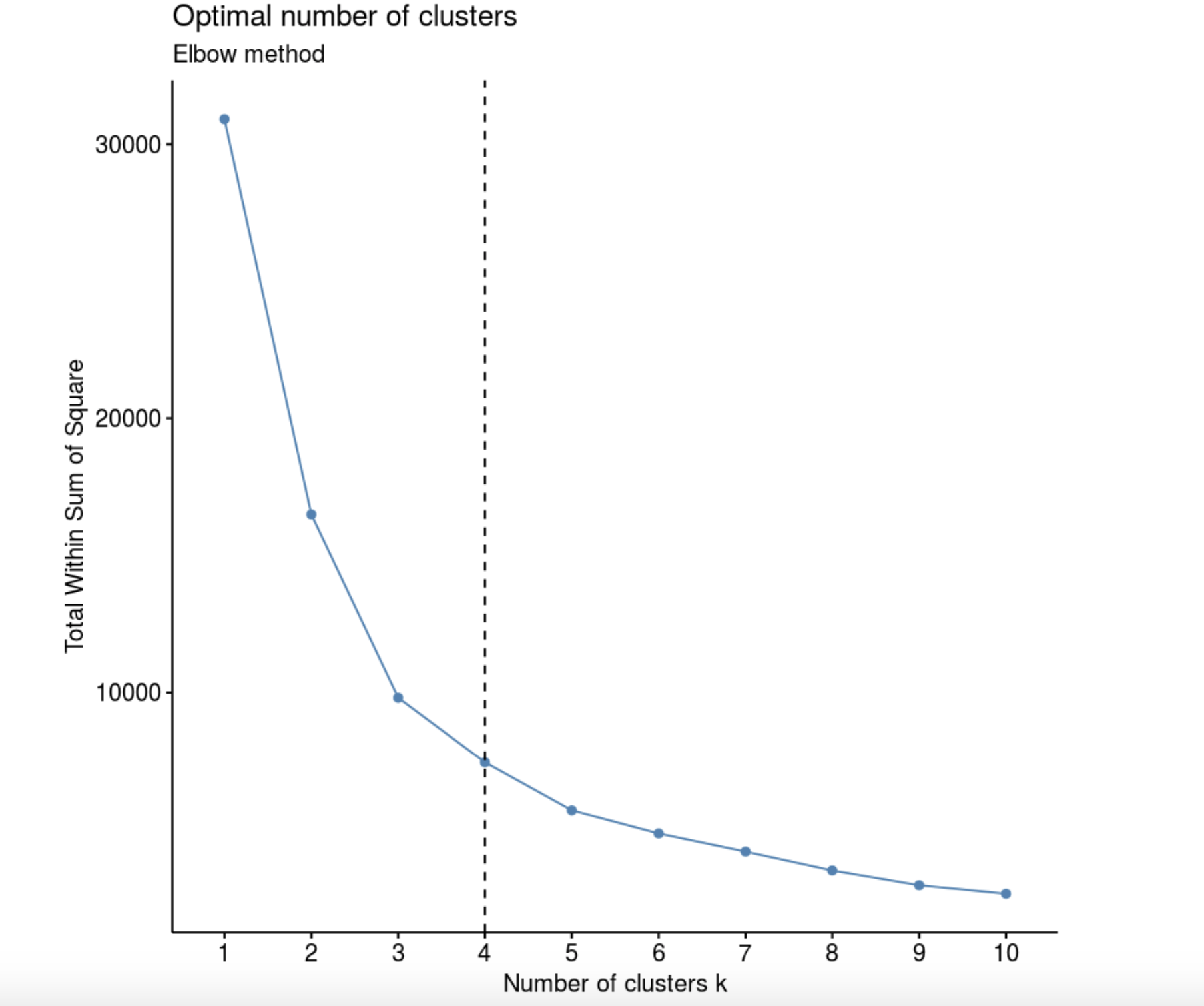
Elbow method (“ ”, “ ”). , k, – W(K), .
library(factoextra)
fviz_nbclust(data, kmeans, method = "wss") +
labs(subtitle = "Elbow method") +
geom_vline(xintercept = 4, linetype = 2)
data.dist <- dist((data))
hc <- hclust(data.dist, method = "ward.D2")
plot(hc, cex = 0.7)
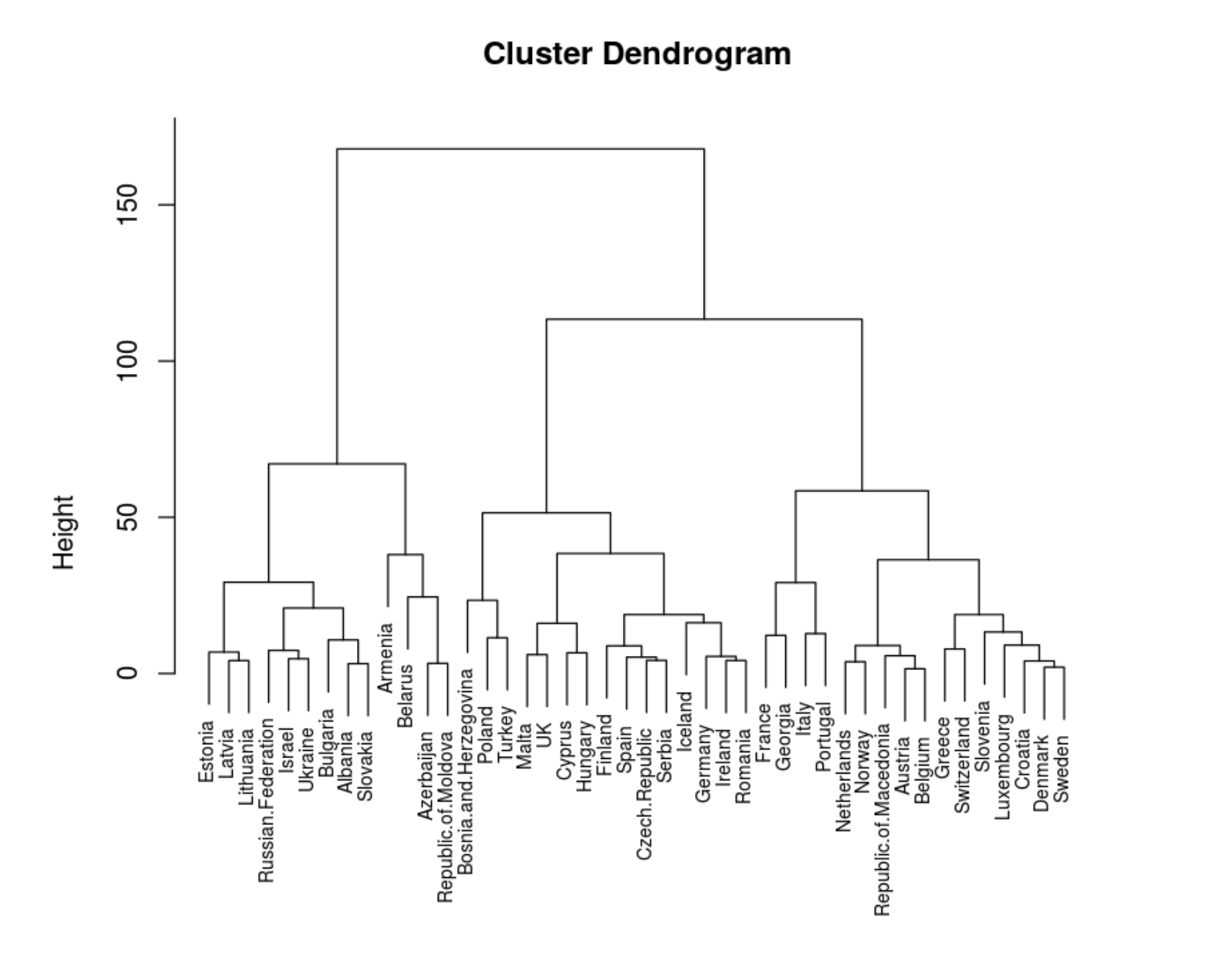
. .
colors=c('green', 'red', 'blue')
hcd = as.dendrogram(hc)
clusMember = cutree(hc, 4)
colLab <- function(n) {
if (is.leaf(n)) {
a <- attributes(n)
labCol <- colors[data.group[n]]
attr(n, "nodePar") <- c(a$nodePar, lab.col = labCol)
}
n
}
clusDendro = dendrapply(hcd, colLab)
plot(clusDendro, main = "Cool Dendrogram", type = "triangle")
rect.hclust(hc, k = 4)
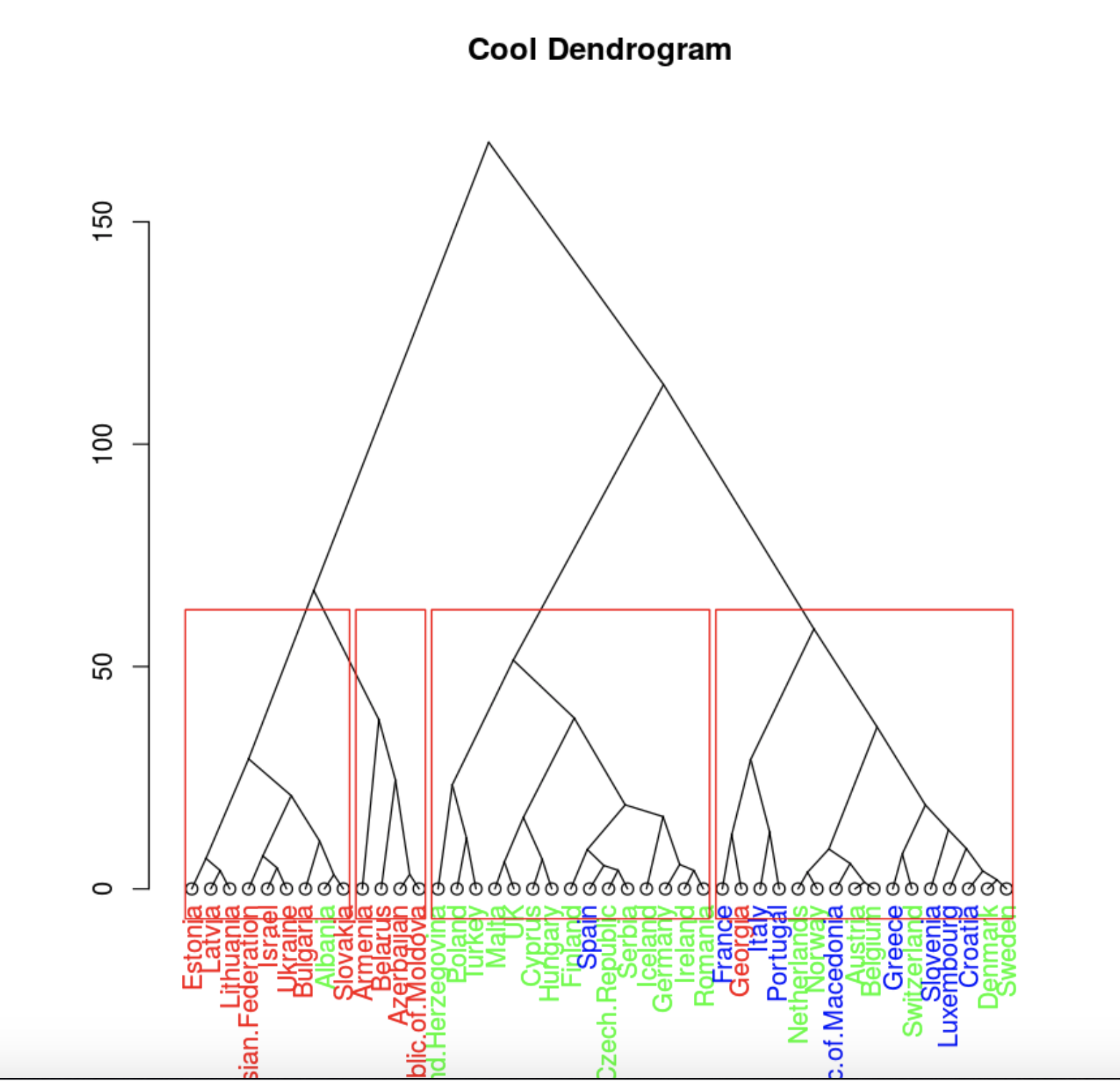
. , .
, , , 4 .
plot(clusDendro, main = "Cool Dendrogram", type = "triangle")
data.hclas_group <- factor(cutree(hc, k = 3))
rect.hclust(hc, k = 3)
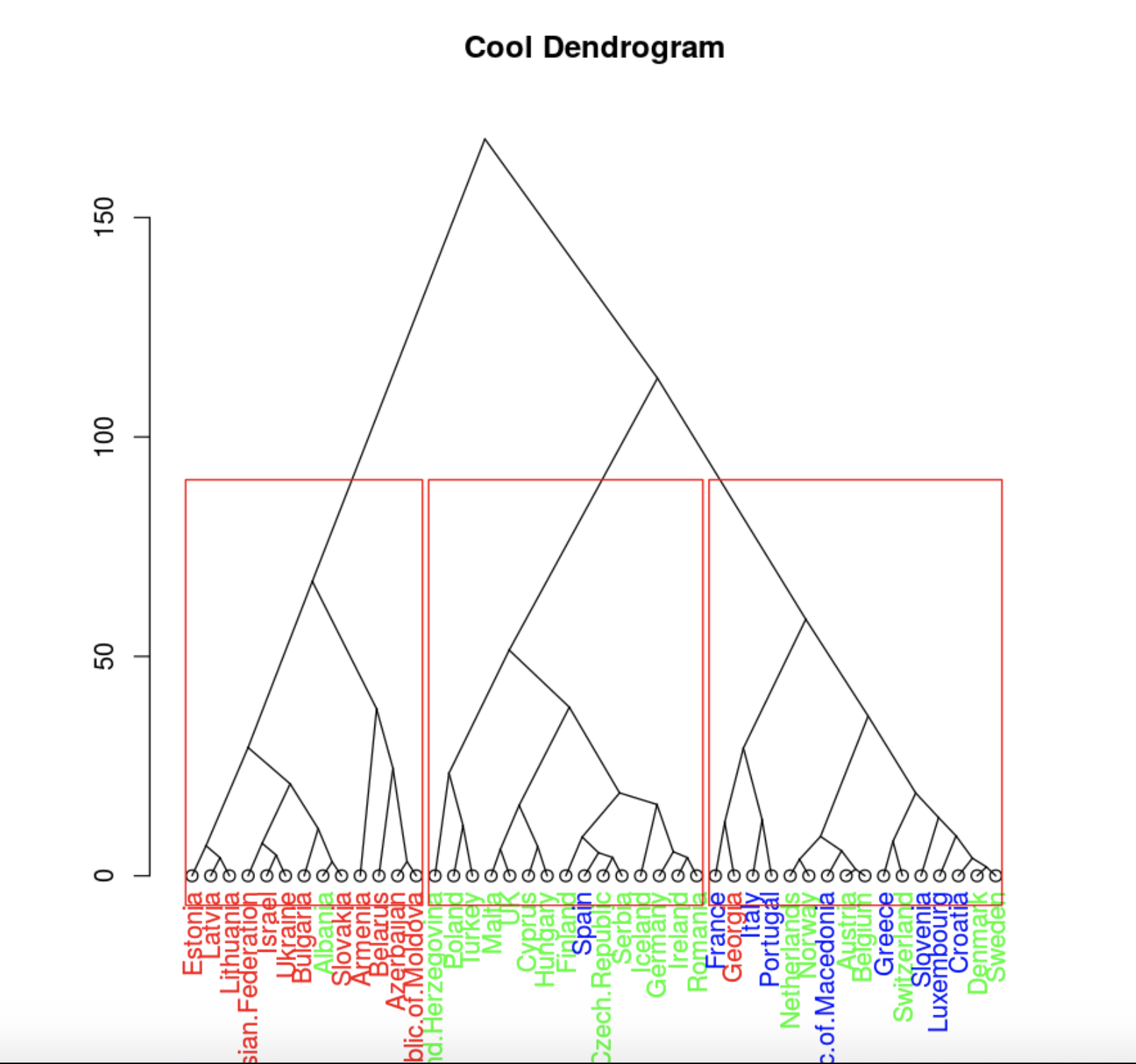
, , .
library(FactoMineR)
res.pca <- PCA(data,scale.unit=T, graph = F)
fviz_pca_biplot(res.pca,
col = colors[data.hclas_group], palette = "jco",
label = "var",
ellipse.level = 0.8,
addEllipses = T,
col.var = "black",
legend.title = "groups4")
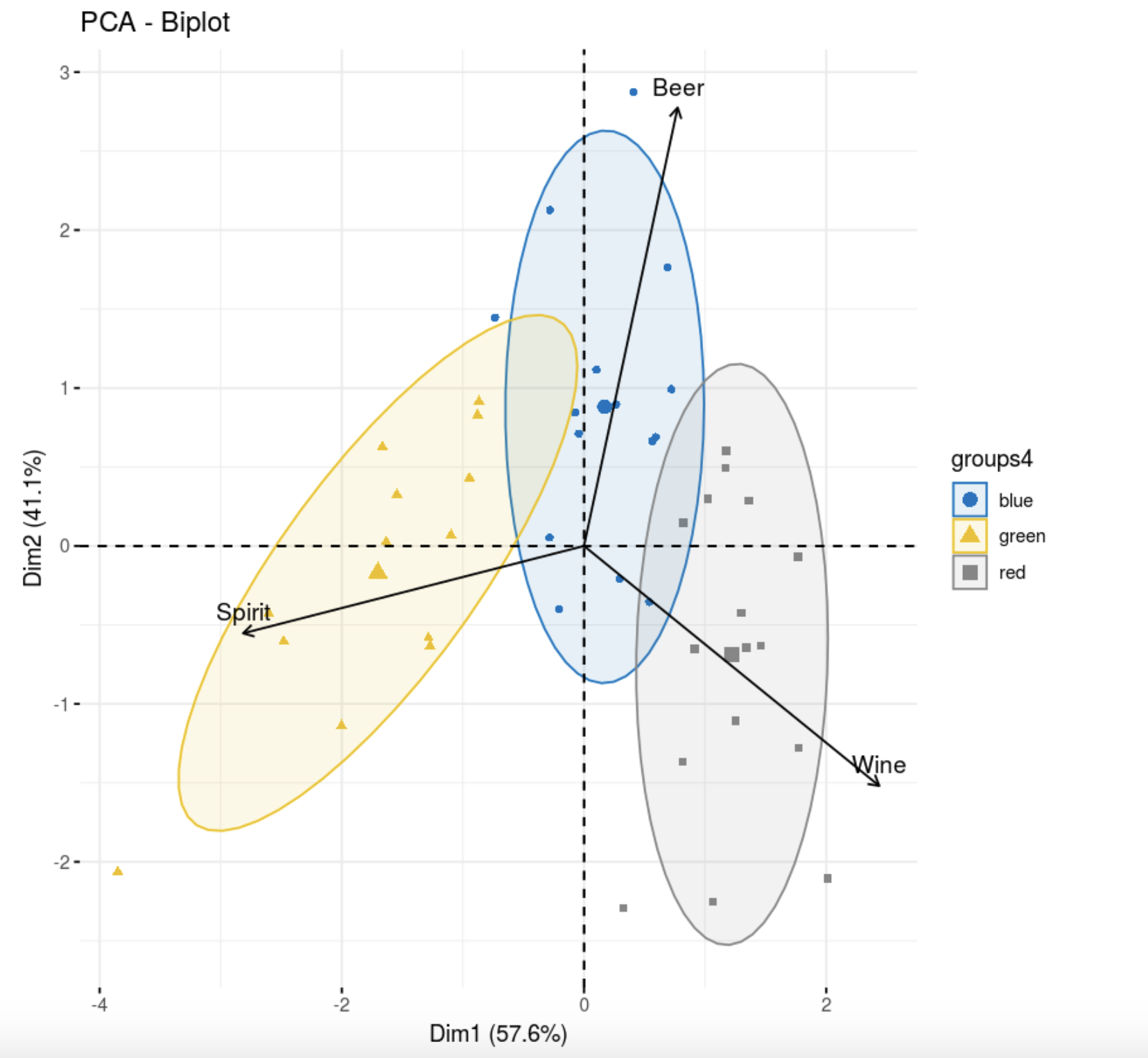
, , . , , , , . , , , k-++.
library(flexclust)
data.kk <- kcca(data, k=3, family=kccaFamily("kmeans"),
control=list(initcent="kmeanspp"))
fviz_pca_biplot(res.pca,
col.ind =as.factor(data.kk@cluster), palette = "jco",
label = "var",
ellipse.level = 0.8,
addEllipses = T,
col.var = "black", repel = TRUE,
legend.title = "clusters")
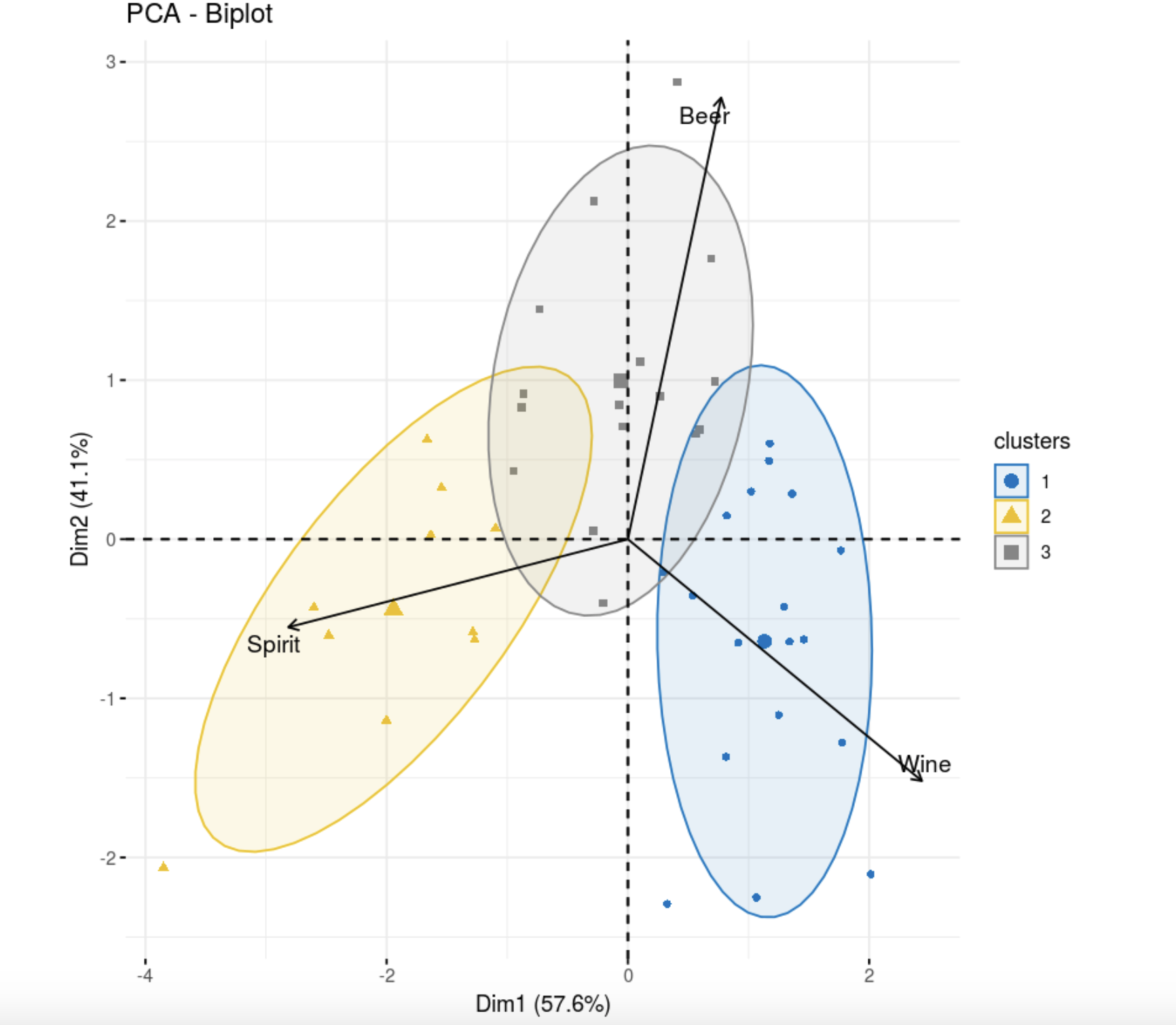
, k- . , , .
, , hclust. .

, , . . , .
. . , , , . , , . , .
Sería posible llevar a cabo la agrupación basada en la suposición de modelos de agrupación utilizando criterios de información ( aquí está la descripción ), y también intentar el análisis discriminante clásico para este conjunto de datos. Si este artículo fue útil, planeo publicar una secuela.