Al estudiar Data Science, decidí compilar para mí un resumen de las técnicas básicas utilizadas en el análisis de datos. Refleja los nombres de los métodos, describe brevemente la esencia y proporciona el código Python para una aplicación rápida. Estaba preparando un compendio para mí, pero pensé que también podría ser útil para alguien, por ejemplo, antes de una entrevista, en una competencia o al comenzar un nuevo proyecto. Diseñado para una audiencia que generalmente está familiarizada con todos estos métodos, pero tiene la necesidad de actualizarlos en la memoria. Artículo debajo del corte.- Clasificador ingenuo de Bayes . La fórmula para calcular la probabilidad de clasificar una observación como una u otra clase:
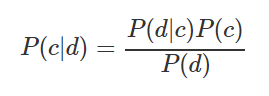
Por ejemplo, debe calcular la probabilidad de que se lleve a cabo un partido deportivo siempre que el clima sea soleado. Los datos de origen y los cálculos se muestran en la tabla a continuación: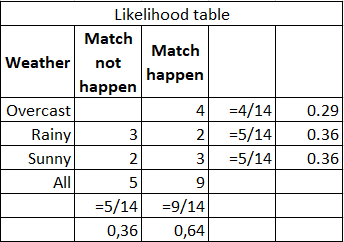
Puede calcular mediante la fórmula (3/9) * (9/14) / (5/14) = 60%, o simplemente desde el sentido común 3 / (2 + 3) = 60%. Fortalezas : fácil de interpretar el resultado, adecuado para muestras grandes y clasificación de varias clases. Debilidades : la suposición de que las características son independientes no siempre se cumple; las características deben formar un grupo completo de eventos.
from sklearn.datasets import load_iris
from sklearn.model_selection import train_test_split
from sklearn.naive_bayes import GaussianNB
X, y = load_iris(return_X_y=True)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.5, random_state=0)
gnb = GaussianNB()
y_pred = gnb.fit(X_train, y_train).predict(X_test)
print("Number of mislabeled points out of a total %d points : %d"
% (X_test.shape[0], (y_test != y_pred).sum()))
- Método de vecinos más cercanos . Clasifica cada observación de acuerdo con el grado de similitud con otras observaciones. El algoritmo no es paramétrico (no hay restricciones en los datos, por ejemplo, la función de distribución) y utiliza entrenamiento diferido (no se utilizan modelos pre-entrenados, todos los datos disponibles se usan durante la clasificación).
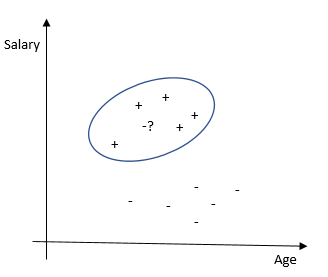
Fortalezas : fácil de interpretar el resultado, muy adecuado para tareas con un pequeño número de variables explicativas. Debilidades : baja precisión en comparación con otros métodos. Requiere una potencia informática considerable con una gran cantidad de variables explicativas y muestras grandes.
from sklearn.neighbors import KNeighborsClassifier
X = [[0], [1], [2], [3]]
y = [0, 0, 1, 1]
neigh = KNeighborsClassifier(n_neighbors=3)
neigh.fit(X, y)
print(neigh.predict([[1.1]]))
print(neigh.predict_proba([[0.9]]))
- Método de vector de soporte (SVM) . Cada objeto de datos se representa como un vector (punto) en el espacio p-dimensional. La tarea es separar los puntos con un hiperplano. Es decir, ¿es posible encontrar dicho hiperplano para que la distancia desde él hasta el punto más cercano sea máxima? Puede haber muchos hiperplanos buscados; por lo tanto, se cree que maximizar la brecha entre clases contribuye a una clasificación más segura.
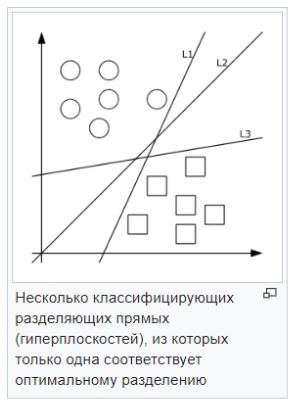
— . , , . . — , , , , . .
from sklearn import svm
X = [[0, 0], [1, 1]]
y = [0, 1]
clf = svm.SVC()
clf.fit(X, y)
clf.predict([[2., 2.]])
- Árboles de decisión . División de datos en submuestras de acuerdo con una determinada condición en forma de estructura de árbol. Matemáticamente, la división en clases ocurre hasta que se encuentran todas las condiciones que determinan la clase con la mayor precisión posible, es decir, cuando no hay representantes de otra clase en cada clase. En la práctica, se utiliza un número limitado de características y capas, y siempre hay dos ramas.
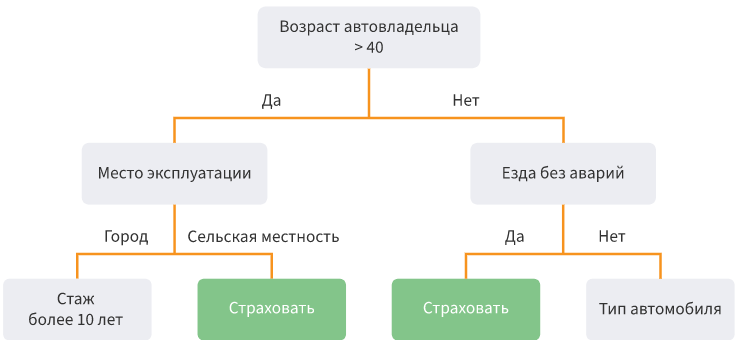
Fortalezas : es posible simular procesos complejos e interpretarlos fácilmente. La clasificación multiclase es posible. Debilidades : es fácil volver a entrenar el modelo si crea muchas capas. Las emisiones pueden afectar la precisión; la solución a estos problemas es recortar los niveles inferiores.
from sklearn.datasets import load_iris
from sklearn import tree
X, y = load_iris(return_X_y=True)
clf = tree.DecisionTreeClassifier()
clf = clf.fit(X, y)
tree.plot_tree(clf.fit(iris.data, iris.target))
- / . . — . (random patching) . oob-.
: , , , , , . , . — , . , ( 100 000), — .
from sklearn.ensemble import RandomForestClassifier
from sklearn.datasets import make_classification
X, y = make_classification(n_samples=1000, n_features=4,
n_informative=2, n_redundant=0,
random_state=0, shuffle=False)
clf = RandomForestClassifier(max_depth=2, random_state=0)
clf.fit(X, y)
print(clf.feature_importances_)
print(clf.predict([[0, 0, 0, 0]]))
- . ( hinge loss function). .
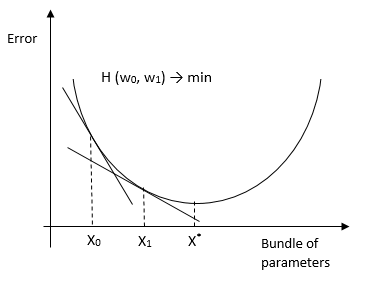
También hay una versión de descenso de gradiente estocástico, que se utiliza para muestras grandes. Su esencia es que considera la derivada no para toda la muestra, sino para cada observación (aprendizaje en línea) (o para el grupo de observación de mini lotes) y cambia los pesos. Como resultado, llega al mismo óptimo que con un HS convencional. Existen métodos para usar HS para OLS, logit, tobit y otros métodos ( evidencia ).Fortalezas : alta precisión de clasificación y pronóstico, adecuado para clasificación de múltiples clases. Debilidades : sensibilidad a los parámetros del modelo.
from sklearn.linear_model import SGDClassifier
X = [[0., 0.], [1., 1.]]
y = [0, 1]
clf = SGDClassifier(loss="hinge", penalty="l2", max_iter=5)
clf.fit(X, y)
clf.predict([[2., 2.]])
clf.coef_
clf.intercept_
- . . , . , , .
: , , , . — .
import numpy as np
import matplotlib.pyplot as plt
from sklearn import ensemble
from sklearn import datasets
from sklearn.utils import shuffle
from sklearn.metrics import mean_squared_error
boston = datasets.load_boston()
X, y = shuffle(boston.data, boston.target, random_state=13)
X = X.astype(np.float32)
offset = int(X.shape[0] * 0.9)
X_train, y_train = X[:offset], y[:offset]
X_test, y_test = X[offset:], y[offset:]
params = {'n_estimators': 500, 'max_depth': 4, 'min_samples_split': 2,
'learning_rate': 0.01, 'loss': 'ls'}
clf = ensemble.GradientBoostingRegressor(**params)
clf.fit(X_train, y_train)
mse = mean_squared_error(y_test, clf.predict(X_test))
print("MSE: %.4f" % mse)
- /logit. 0 1, (log likelihood). — Y w.

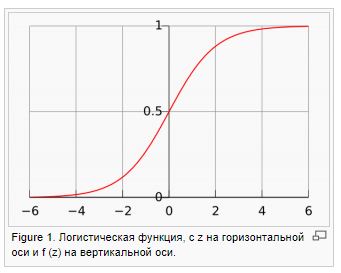
: , . — , .
from sklearn.datasets import load_iris
from sklearn.linear_model import LogisticRegression
X, y = load_iris(return_X_y=True)
clf = LogisticRegression(random_state=0).fit(X, y)
clf.predict(X[:2, :])
clf.predict_proba(X[:2, :])
clf.score(X, y)
-Probit. , , , .
import statsmodels
result_3 = statsmodels.discrete.
discrete_model.Probit(labf_part, ind_var_probit )
print(result_3.summary())
-Tobit. , .
from sklearn.datasets import make_regression
import matplotlib.pyplot as plt
import pandas as pd
from tobit import *
tr = TobitModel()
tr = tr.fit(x, y, cens, verbose=False)
tr.coef_
- , , . , , , . .