Los especialistas en análisis y procesamiento de datos tienen muchas herramientas para crear modelos de clasificación. Uno de los métodos más populares y confiables para desarrollar tales modelos es usar el algoritmo Random Forest (RF). Para intentar mejorar el rendimiento de un modelo construido utilizando el algoritmo de RF , puede utilizar la optimización del hiperparámetro del modelo ( Ajuste de hiperparámetro , HT). Además, existe un enfoque generalizado según el cual los datos, antes de ser transferidos al modelo, se procesan utilizando el Análisis de componentes principales , PCA). ¿Pero vale la pena usarlo? ¿No es el propósito principal del algoritmo de RF ayudar al analista a interpretar la importancia de los rasgos?Sí, el uso del algoritmo PCA puede conducir a una ligera complicación de la interpretación de cada "característica" en el análisis de la "importancia de las características" del modelo de RF. Sin embargo, el algoritmo PCA reduce la dimensión del espacio de características, lo que puede conducir a una disminución en la cantidad de características que el modelo RF debe procesar. Tenga en cuenta que el volumen de cálculos es una de las principales desventajas del algoritmo de bosque aleatorio (es decir, puede llevar mucho tiempo completar el modelo). La aplicación del algoritmo PCA puede ser una parte muy importante del modelado, especialmente en los casos en que funcionan con cientos o incluso miles de características. Como resultado, si lo más importante es simplemente crear el modelo más efectivo, y al mismo tiempo puede sacrificar la precisión de determinar la importancia de los atributos, entonces el PCA puede valer la pena intentarlo.Ahora al grano. Vamos a trabajar con un conjunto de datos sobre el cáncer de mama : Scikit-learn "cáncer de mama" . Crearemos tres modelos y compararemos su efectividad. A saber, estamos hablando de los siguientes modelos:
, PCA). ¿Pero vale la pena usarlo? ¿No es el propósito principal del algoritmo de RF ayudar al analista a interpretar la importancia de los rasgos?Sí, el uso del algoritmo PCA puede conducir a una ligera complicación de la interpretación de cada "característica" en el análisis de la "importancia de las características" del modelo de RF. Sin embargo, el algoritmo PCA reduce la dimensión del espacio de características, lo que puede conducir a una disminución en la cantidad de características que el modelo RF debe procesar. Tenga en cuenta que el volumen de cálculos es una de las principales desventajas del algoritmo de bosque aleatorio (es decir, puede llevar mucho tiempo completar el modelo). La aplicación del algoritmo PCA puede ser una parte muy importante del modelado, especialmente en los casos en que funcionan con cientos o incluso miles de características. Como resultado, si lo más importante es simplemente crear el modelo más efectivo, y al mismo tiempo puede sacrificar la precisión de determinar la importancia de los atributos, entonces el PCA puede valer la pena intentarlo.Ahora al grano. Vamos a trabajar con un conjunto de datos sobre el cáncer de mama : Scikit-learn "cáncer de mama" . Crearemos tres modelos y compararemos su efectividad. A saber, estamos hablando de los siguientes modelos:- El modelo básico basado en el algoritmo de RF (abreviaremos este modelo de RF).
- El mismo modelo que el número 1, pero en el que se aplica una reducción en la dimensión del espacio de características utilizando el método del componente principal (RF + PCA).
- El mismo modelo que el No. 2, pero construido utilizando la optimización de hiperparámetros (RF + PCA + HT).
1. Importar datos
Para comenzar, cargue los datos y cree un marco de datos Pandas. Como utilizamos un conjunto de datos de "juguete" previamente borrado de Scikit-learn, entonces ya podemos comenzar el proceso de modelado. Pero incluso cuando use dichos datos, se recomienda que siempre comience a trabajar realizando un análisis preliminar de los datos utilizando los siguientes comandos aplicados al marco de datos ( df):df.head() - echar un vistazo al nuevo marco de datos y ver si se ve como se esperaba.df.info()- para conocer las características de los tipos de datos y el contenido de las columnas. Puede ser necesario realizar una conversión de tipo de datos antes de continuar.df.isna()- para asegurarse de que no hay valores en los datos NaN. Los valores correspondientes, si los hay, pueden necesitar ser procesados de alguna manera, o, si es necesario, puede ser necesario eliminar filas enteras del marco de datos.df.describe() - para encontrar los valores mínimos, máximos y promedio de los indicadores en las columnas, para encontrar los indicadores del cuadrado medio y la desviación probable en las columnas.
En nuestro conjunto de datos, una columna cancer(cáncer) es la variable objetivo cuyo valor queremos predecir usando el modelo. 0significa "sin enfermedad". 1- "la presencia de la enfermedad".import pandas as pd
from sklearn.datasets import load_breast_cancer
columns = ['mean radius', 'mean texture', 'mean perimeter', 'mean area', 'mean smoothness', 'mean compactness', 'mean concavity', 'mean concave points', 'mean symmetry', 'mean fractal dimension', 'radius error', 'texture error', 'perimeter error', 'area error', 'smoothness error', 'compactness error', 'concavity error', 'concave points error', 'symmetry error', 'fractal dimension error', 'worst radius', 'worst texture', 'worst perimeter', 'worst area', 'worst smoothness', 'worst compactness', 'worst concavity', 'worst concave points', 'worst symmetry', 'worst fractal dimension']
dataset = load_breast_cancer()
data = pd.DataFrame(dataset['data'], columns=columns)
data['cancer'] = dataset['target']
display(data.head())
display(data.info())
display(data.isna().sum())
display(data.describe())
. . , cancer, , . 0 « ». 1 — « »2.
Ahora divida los datos usando la función Scikit-learn train_test_split. Queremos darle al modelo tantos datos de entrenamiento como sea posible. Sin embargo, necesitamos tener suficientes datos a nuestra disposición para probar el modelo. En general, podemos decir que, a medida que aumenta el número de filas en el conjunto de datos, también lo hace la cantidad de datos que se pueden considerar educativos.Por ejemplo, si hay millones de líneas, puede dividir el conjunto resaltando el 90% de las líneas para datos de entrenamiento y el 10% para datos de prueba. Pero el conjunto de datos de prueba contiene solo 569 filas. Y esto no es tanto para entrenar y probar el modelo. Como resultado, para ser justos en relación con los datos de capacitación y verificación, dividiremos el conjunto en dos partes iguales: 50% de datos de capacitación y 50% de datos de verificación. Instalamosstratify=y para garantizar que tanto el conjunto de datos de entrenamiento como el de prueba tengan la misma proporción de 0 y 1 que el conjunto de datos original.from sklearn.model_selection import train_test_split
X = data.drop('cancer', axis=1)
y = data['cancer']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.50, random_state = 2020, stratify=y)
3. Escalado de datos
Antes de proceder al modelado, debe "centrar" y "estandarizar" los datos escalando . El escalado se realiza debido al hecho de que diferentes cantidades se expresan en diferentes unidades. Este procedimiento le permite organizar una "lucha justa" entre los signos para determinar su importancia. Además, convertimos y_traindel tipo de datos Pandas Seriesa la matriz NumPy para que luego el modelo pueda trabajar con los objetivos correspondientes.import numpy as np
from sklearn.preprocessing import StandardScaler
ss = StandardScaler()
X_train_scaled = ss.fit_transform(X_train)
X_test_scaled = ss.transform(X_test)
y_train = np.array(y_train)
4. Entrenamiento del modelo básico (modelo No. 1, RF)
Ahora cree el modelo número 1. En él, recordamos que solo se utiliza el algoritmo Random Forest. Utiliza todas las funciones y se configura con los valores predeterminados (los detalles sobre estas configuraciones se pueden encontrar en la documentación de sklearn.ensemble.RandomForestClassifier ). Inicializa el modelo. Después de eso, la entrenaremos en datos escalados. La precisión del modelo se puede medir en los datos de entrenamiento:from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import recall_score
rfc = RandomForestClassifier()
rfc.fit(X_train_scaled, y_train)
display(rfc.score(X_train_scaled, y_train))
Si estamos interesados en saber qué rasgos son los más importantes para el modelo de RF en la predicción del cáncer de mama, podemos visualizar y cuantificar los índices de gravedad de los rasgos haciendo referencia al atributo feature_importances_:feats = {}
for feature, importance in zip(data.columns, rfc_1.feature_importances_):
feats[feature] = importance
importances = pd.DataFrame.from_dict(feats, orient='index').rename(columns={0: 'Gini-Importance'})
importances = importances.sort_values(by='Gini-Importance', ascending=False)
importances = importances.reset_index()
importances = importances.rename(columns={'index': 'Features'})
sns.set(font_scale = 5)
sns.set(style="whitegrid", color_codes=True, font_scale = 1.7)
fig, ax = plt.subplots()
fig.set_size_inches(30,15)
sns.barplot(x=importances['Gini-Importance'], y=importances['Features'], data=importances, color='skyblue')
plt.xlabel('Importance', fontsize=25, weight = 'bold')
plt.ylabel('Features', fontsize=25, weight = 'bold')
plt.title('Feature Importance', fontsize=25, weight = 'bold')
display(plt.show())
display(importances)
Visualización de la "importancia" de los signos.Indicadores de significancia5. El método de los componentes principales.
Ahora preguntemos cómo podemos mejorar el modelo básico de RF. Utilizando la técnica de reducir la dimensión del espacio de características, es posible presentar el conjunto de datos inicial a través de menos variables y, al mismo tiempo, reducir la cantidad de recursos computacionales necesarios para garantizar el funcionamiento del modelo. Con el PCA, puede estudiar la varianza de muestra acumulativa de estas características para comprender qué características explican la mayor parte de la varianza en los datos.Inicializamos el objeto PCA ( pca_test), indicando el número de componentes (características) que deben considerarse. Establecemos este indicador en 30 para ver la varianza explicada de todos los componentes generados antes de decidir cuántos componentes necesitamos. Luego transferimos a los pca_testdatos escaladosX_trainutilizando el método pca_test.fit(). Después de eso visualizamos los datos.import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.decomposition import PCA
pca_test = PCA(n_components=30)
pca_test.fit(X_train_scaled)
sns.set(style='whitegrid')
plt.plot(np.cumsum(pca_test.explained_variance_ratio_))
plt.xlabel('number of components')
plt.ylabel('cumulative explained variance')
plt.axvline(linewidth=4, color='r', linestyle = '--', x=10, ymin=0, ymax=1)
display(plt.show())
evr = pca_test.explained_variance_ratio_
cvr = np.cumsum(pca_test.explained_variance_ratio_)
pca_df = pd.DataFrame()
pca_df['Cumulative Variance Ratio'] = cvr
pca_df['Explained Variance Ratio'] = evr
display(pca_df.head(10))
Después de que el número de componentes utilizados excede de 10, el aumento en su número no aumenta en gran medida la varianza explicadaEste marco de datos contiene indicadores tales como la relación de varianza acumulada (tamaño acumulado de la varianza explicada de los datos) y la razón de varianza explicada (contribución de cada componente al volumen total de la varianza explicada).Si observa el marco de datos anterior, resulta que usar el PCA para pasar de 30 variables a 10 a componentes permite explicar el 95% de la dispersión de datos. Los otros 20 componentes representan menos del 5% de la variación, lo que significa que podemos rechazarlos. Siguiendo esta lógica, utilizamos el PCA para reducir el número de componentes de 30 a 10 paraX_trainyX_test. Escribimos estos conjuntos de datos de "dimensión reducida" creados artificialmente enX_train_scaled_pcay dentroX_test_scaled_pca.pca = PCA(n_components=10)
pca.fit(X_train_scaled)
X_train_scaled_pca = pca.transform(X_train_scaled)
X_test_scaled_pca = pca.transform(X_test_scaled)
Cada componente es una combinación lineal de variables de origen con los correspondientes "pesos". Podemos ver estos "pesos" para cada componente creando un marco de datos.pca_dims = []
for x in range(0, len(pca_df)):
pca_dims.append('PCA Component {}'.format(x))
pca_test_df = pd.DataFrame(pca_test.components_, columns=columns, index=pca_dims)
pca_test_df.head(10).T
Marco de datos de información de componentes6. Capacitación del modelo básico de RF después de aplicar el método de componentes principales a los datos (modelo No. 2, RF + PCA)
Ahora podemos pasar a otro modelo de datos básicos de RF X_train_scaled_pcay y_trainy podemos averiguar acerca de si hay una mejora en la precisión de las predicciones emitidas por el modelo.rfc = RandomForestClassifier()
rfc.fit(X_train_scaled_pca, y_train)
display(rfc.score(X_train_scaled_pca, y_train))
Los modelos se comparan a continuación.7. Optimización de hiperparámetros. Ronda 1: RandomizedSearchCV
Después de procesar los datos utilizando el método del componente principal, puede intentar utilizar la optimización de los hiperparámetros del modelo para mejorar la calidad de las predicciones producidas por el modelo de RF. Los hiperparámetros se pueden considerar como algo así como "configuraciones" del modelo. Las configuraciones que son perfectas para un conjunto de datos no funcionarán para otro, es por eso que necesita optimizarlas.Puede comenzar con el algoritmo RandomizedSearchCV, que le permite explorar más o menos una amplia gama de valores. Las descripciones de todos los hiperparámetros para los modelos de RF se pueden encontrar aquí .En el curso del trabajo, generamos una entidad param_distque contiene, para cada hiperparámetro, un rango de valores que necesitan ser probados. A continuación, inicializamos el objeto.rsusando la función RandomizedSearchCV(), pasándole el modelo RF param_dist, el número de iteraciones y el número de validaciones cruzadas que deben realizarse.El hiperparámetro le verbosepermite controlar la cantidad de información que muestra el modelo durante su funcionamiento (como la salida de información durante el entrenamiento del modelo). El hiperparámetro le n_jobspermite especificar cuántos núcleos de procesador necesita usar para garantizar el funcionamiento del modelo. Establecerlo n_jobsen un valor -1conducirá a un modelo más rápido, ya que usará todos los núcleos de procesador.Nos dedicaremos a la selección de los siguientes hiperparámetros:n_estimators - el número de "árboles" en el "bosque aleatorio".max_features - la cantidad de funciones para seleccionar la división.max_depth - Profundidad máxima de los árboles.min_samples_split - el número mínimo de objetos necesarios para que un nodo de árbol se divida.min_samples_leaf - El número mínimo de objetos en las hojas.bootstrap - Se utiliza para construir árboles de submuestras con retorno.
from sklearn.model_selection import RandomizedSearchCV
n_estimators = [int(x) for x in np.linspace(start = 100, stop = 1000, num = 10)]
max_features = ['log2', 'sqrt']
max_depth = [int(x) for x in np.linspace(start = 1, stop = 15, num = 15)]
min_samples_split = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
min_samples_leaf = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
bootstrap = [True, False]
param_dist = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
rs = RandomizedSearchCV(rfc_2,
param_dist,
n_iter = 100,
cv = 3,
verbose = 1,
n_jobs=-1,
random_state=0)
rs.fit(X_train_scaled_pca, y_train)
rs.best_params_
Con los valores de los parámetros n_iter = 100y cv = 3, creamos 300 modelos de RF, eligiendo aleatoriamente combinaciones de los hiperparámetros presentados anteriormente. Podemos consultar el atributo best_params_ para obtener información sobre un conjunto de parámetros que le permite crear el mejor modelo. Pero en esta etapa, esto puede no darnos los datos más interesantes sobre los rangos de parámetros que vale la pena explorar en la próxima ronda de optimización. Para descubrir en qué rango de valores vale la pena continuar buscando, podemos obtener fácilmente un marco de datos que contiene los resultados del algoritmo RandomizedSearchCV.rs_df = pd.DataFrame(rs.cv_results_).sort_values('rank_test_score').reset_index(drop=True)
rs_df = rs_df.drop([
'mean_fit_time',
'std_fit_time',
'mean_score_time',
'std_score_time',
'params',
'split0_test_score',
'split1_test_score',
'split2_test_score',
'std_test_score'],
axis=1)
rs_df.head(10)
Resultados del algoritmo RandomizedSearchCVAhora crearemos gráficos de barras en los que, en el eje X, se encuentran los valores de hiperparámetro, y en el eje Y son los valores promedio que muestran los modelos. Esto permitirá comprender qué valores de hiperparámetros, en promedio, muestran su mejor rendimiento.fig, axs = plt.subplots(ncols=3, nrows=2)
sns.set(style="whitegrid", color_codes=True, font_scale = 2)
fig.set_size_inches(30,25)
sns.barplot(x='param_n_estimators', y='mean_test_score', data=rs_df, ax=axs[0,0], color='lightgrey')
axs[0,0].set_ylim([.83,.93])axs[0,0].set_title(label = 'n_estimators', size=30, weight='bold')
sns.barplot(x='param_min_samples_split', y='mean_test_score', data=rs_df, ax=axs[0,1], color='coral')
axs[0,1].set_ylim([.85,.93])axs[0,1].set_title(label = 'min_samples_split', size=30, weight='bold')
sns.barplot(x='param_min_samples_leaf', y='mean_test_score', data=rs_df, ax=axs[0,2], color='lightgreen')
axs[0,2].set_ylim([.80,.93])axs[0,2].set_title(label = 'min_samples_leaf', size=30, weight='bold')
sns.barplot(x='param_max_features', y='mean_test_score', data=rs_df, ax=axs[1,0], color='wheat')
axs[1,0].set_ylim([.88,.92])axs[1,0].set_title(label = 'max_features', size=30, weight='bold')
sns.barplot(x='param_max_depth', y='mean_test_score', data=rs_df, ax=axs[1,1], color='lightpink')
axs[1,1].set_ylim([.80,.93])axs[1,1].set_title(label = 'max_depth', size=30, weight='bold')
sns.barplot(x='param_bootstrap',y='mean_test_score', data=rs_df, ax=axs[1,2], color='skyblue')
axs[1,2].set_ylim([.88,.92])
axs[1,2].set_title(label = 'bootstrap', size=30, weight='bold')
plt.show()
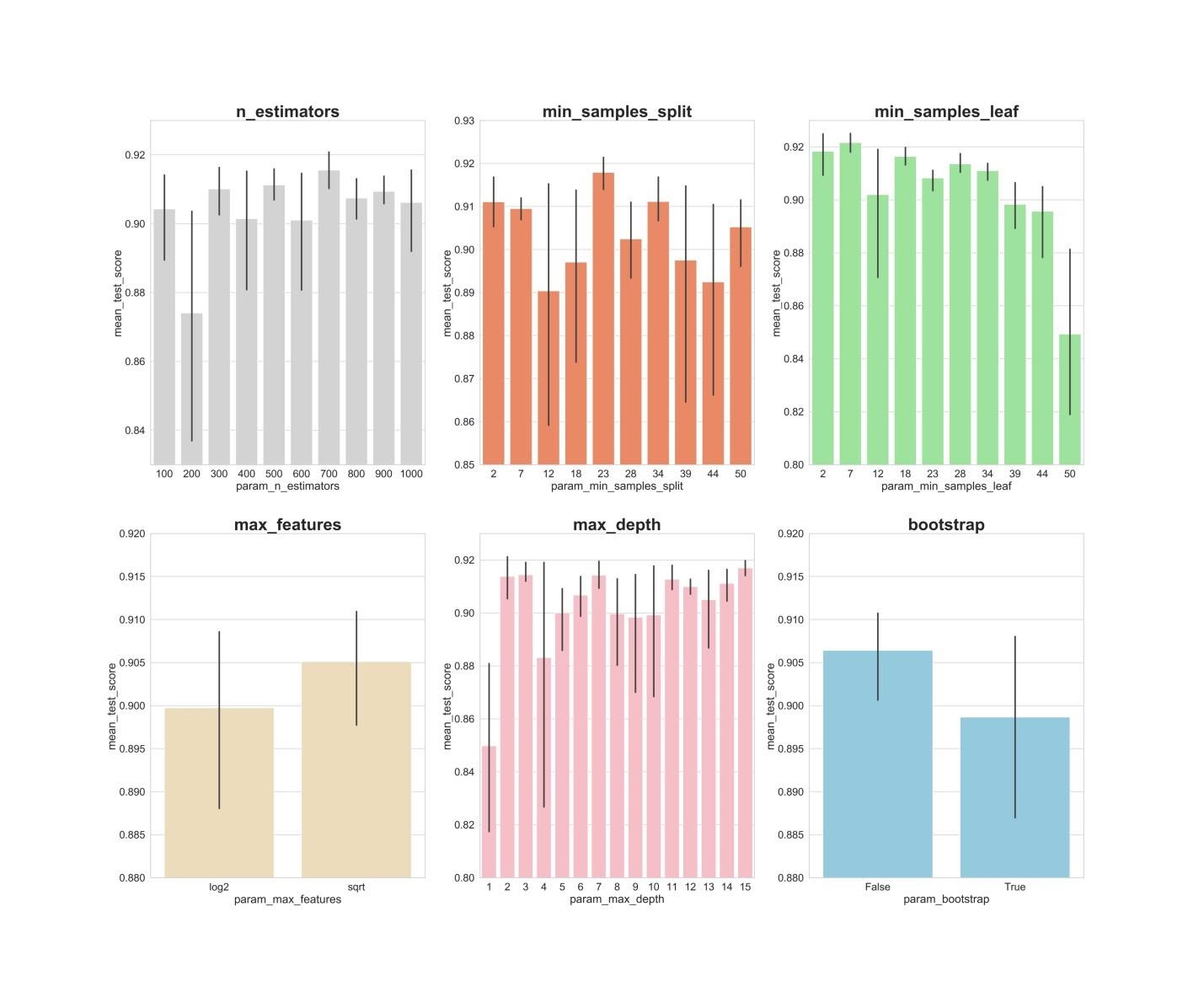
n_estimators: los valores de 300, 500, 700, aparentemente, muestran los mejores resultados promedio.min_samples_split: Los valores pequeños como 2 y 7 parecen mostrar los mejores resultados. El valor 23 también se ve bien. Puede examinar varios valores de este hiperparámetro en exceso de 2, así como varios valores de aproximadamente 23.min_samples_leaf: Existe la sensación de que los valores pequeños de este hiperparámetro dan mejores resultados. Esto significa que podemos experimentar valores entre 2 y 7.max_features: la opción sqrtda el resultado promedio más alto.max_depth: no existe una relación clara entre el valor del hiperparámetro y el resultado del modelo, pero existe la sensación de que los valores 2, 3, 7, 11, 15 se ven bien.bootstrap: el valor Falsemuestra el mejor resultado promedio.
Ahora, utilizando estos hallazgos, podemos pasar a la segunda ronda de optimización de hiperparámetros. Esto reducirá el rango de valores que nos interesan.8. Optimización de hiperparámetros. Ronda 2: GridSearchCV (preparación final de parámetros para el modelo No. 3, RF + PCA + HT)
Después de aplicar el algoritmo RandomizedSearchCV, utilizaremos el algoritmo GridSearchCV para realizar una búsqueda más precisa de la mejor combinación de hiperparámetros. Aquí se investigan los mismos hiperparámetros, pero ahora estamos aplicando una búsqueda más "exhaustiva" de su mejor combinación. Usando el algoritmo GridSearchCV, se examina cada combinación de hiperparámetros. Esto requiere muchos más recursos computacionales que usar el algoritmo RandomizedSearchCV cuando establecemos independientemente el número de iteraciones de búsqueda. Por ejemplo, investigar 10 valores para cada uno de los 6 hiperparámetros con validación cruzada en 3 bloques requerirá 10⁶ x 3, o 3,000,000 de sesiones de entrenamiento modelo. Es por eso que usamos el algoritmo GridSearchCV después de, después de aplicar RandomizedSearchCV, redujimos los rangos de los valores de los parámetros estudiados.Entonces, usando lo que descubrimos con la ayuda de RandomizedSearchCV, examinamos los valores de los hiperparámetros que se han mostrado mejor:from sklearn.model_selection import GridSearchCV
n_estimators = [300,500,700]
max_features = ['sqrt']
max_depth = [2,3,7,11,15]
min_samples_split = [2,3,4,22,23,24]
min_samples_leaf = [2,3,4,5,6,7]
bootstrap = [False]
param_grid = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
gs = GridSearchCV(rfc_2, param_grid, cv = 3, verbose = 1, n_jobs=-1)
gs.fit(X_train_scaled_pca, y_train)
rfc_3 = gs.best_estimator_
gs.best_params_
Aquí aplicamos la validación cruzada en 3 bloques para 540 (3 x 1 x 5 x 6 x 6 x 1) sesiones de entrenamiento modelo, lo que da 1620 sesiones de entrenamiento modelo. Y ahora, después de usar RandomizedSearchCV y GridSearchCV, podemos recurrir al atributo best_params_para averiguar qué valores de hiperparámetros permiten que el modelo funcione mejor con el conjunto de datos en estudio (estos valores se pueden ver en la parte inferior del bloque de código anterior) . Estos parámetros se utilizan para crear el modelo número 3.9. Evaluación de la calidad de los modelos en los datos de verificación.
Ahora puede evaluar los modelos creados en los datos de verificación. Es decir, estamos hablando de esos tres modelos descritos al comienzo del material.Mira estos modelos:y_pred = rfc.predict(X_test_scaled)
y_pred_pca = rfc.predict(X_test_scaled_pca)
y_pred_gs = gs.best_estimator_.predict(X_test_scaled_pca)
Cree matrices de error para los modelos y descubra qué tan bien cada uno de ellos puede predecir el cáncer de seno:from sklearn.metrics import confusion_matrix
conf_matrix_baseline = pd.DataFrame(confusion_matrix(y_test, y_pred), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_baseline_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_pca), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_tuned_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_gs), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
display(conf_matrix_baseline)
display('Baseline Random Forest recall score', recall_score(y_test, y_pred))
display(conf_matrix_baseline_pca)
display('Baseline Random Forest With PCA recall score', recall_score(y_test, y_pred_pca))
display(conf_matrix_tuned_pca)
display('Hyperparameter Tuned Random Forest With PCA Reduced Dimensionality recall score', recall_score(y_test, y_pred_gs))
Resultados del trabajo de los tres modelosAquí se evalúa la métrica "integridad" (recuerdo). El hecho es que estamos lidiando con un diagnóstico de cáncer. Por lo tanto, estamos extremadamente interesados en minimizar los pronósticos falsos negativos emitidos por los modelos.Ante esto, podemos concluir que el modelo básico de RF dio los mejores resultados. Su tasa de completitud fue del 94,97%. En el conjunto de datos de la prueba, hubo un registro de 179 pacientes con cáncer. El modelo encontró 170 de ellos.Resumen
Este estudio proporciona una observación importante. A veces, el modelo RF, que utiliza el método del componente principal y la optimización a gran escala de hiperparámetros, puede no funcionar tan bien como el modelo más común con configuraciones estándar. Pero esta no es una razón para limitarse solo a los modelos más simples. Sin probar diferentes modelos, es imposible decir cuál mostrará el mejor resultado. Y en el caso de los modelos que se utilizan para predecir la presencia de cáncer en los pacientes, podemos decir que cuanto mejor sea el modelo, más vidas se pueden salvar.¡Queridos lectores! ¿Qué tareas resuelves usando métodos de aprendizaje automático?