Datenverarbeitungs- und Analysespezialisten verfügen über zahlreiche Tools zum Erstellen von Klassifizierungsmodellen. Eine der beliebtesten und zuverlässigsten Methoden zur Entwicklung solcher Modelle ist die Verwendung des Random Forest (RF) -Algorithmus. Um zu versuchen, die Leistung eines mit dem RF- Algorithmus erstellten Modells zu verbessern , können Sie die Optimierung des Hyperparameters des Modells ( Hyperparameter Tuning , HT) verwenden. Darüber hinaus gibt es einen weit verbreiteten Ansatz, nach dem die Daten vor der Übertragung in das Modell mithilfe der Hauptkomponentenanalyse verarbeitet werden , PCA). Aber lohnt es sich zu benutzen? Ist es nicht der Hauptzweck des RF-Algorithmus, dem Analytiker zu helfen, die Bedeutung der Merkmale zu interpretieren?Ja, die Verwendung des PCA-Algorithmus kann zu einer geringfügigen Komplikation der Interpretation jedes „Merkmals“ bei der Analyse der „Bedeutung von Merkmalen“ des RF-Modells führen. Der PCA-Algorithmus reduziert jedoch die Dimension des Merkmalsraums, was zu einer Verringerung der Anzahl von Merkmalen führen kann, die vom RF-Modell verarbeitet werden müssen. Bitte beachten Sie, dass das Rechenvolumen einer der Hauptnachteile des Random Forest-Algorithmus ist (dh die Fertigstellung des Modells kann lange dauern). Die Anwendung des PCA-Algorithmus kann ein sehr wichtiger Bestandteil der Modellierung sein, insbesondere in Fällen, in denen sie mit Hunderten oder sogar Tausenden von Funktionen arbeiten. Wenn das Wichtigste darin besteht, einfach das effektivste Modell zu erstellen und gleichzeitig die Genauigkeit der Bestimmung der Wichtigkeit der Attribute zu opfern, ist die PCA möglicherweise einen Versuch wert.Nun zum Punkt. Wir werden mit einem Brustkrebs-Datensatz arbeiten - Scikit-Learn "Brustkrebs" . Wir werden drei Modelle erstellen und deren Wirksamkeit vergleichen. Wir sprechen nämlich über folgende Modelle:
, PCA). Aber lohnt es sich zu benutzen? Ist es nicht der Hauptzweck des RF-Algorithmus, dem Analytiker zu helfen, die Bedeutung der Merkmale zu interpretieren?Ja, die Verwendung des PCA-Algorithmus kann zu einer geringfügigen Komplikation der Interpretation jedes „Merkmals“ bei der Analyse der „Bedeutung von Merkmalen“ des RF-Modells führen. Der PCA-Algorithmus reduziert jedoch die Dimension des Merkmalsraums, was zu einer Verringerung der Anzahl von Merkmalen führen kann, die vom RF-Modell verarbeitet werden müssen. Bitte beachten Sie, dass das Rechenvolumen einer der Hauptnachteile des Random Forest-Algorithmus ist (dh die Fertigstellung des Modells kann lange dauern). Die Anwendung des PCA-Algorithmus kann ein sehr wichtiger Bestandteil der Modellierung sein, insbesondere in Fällen, in denen sie mit Hunderten oder sogar Tausenden von Funktionen arbeiten. Wenn das Wichtigste darin besteht, einfach das effektivste Modell zu erstellen und gleichzeitig die Genauigkeit der Bestimmung der Wichtigkeit der Attribute zu opfern, ist die PCA möglicherweise einen Versuch wert.Nun zum Punkt. Wir werden mit einem Brustkrebs-Datensatz arbeiten - Scikit-Learn "Brustkrebs" . Wir werden drei Modelle erstellen und deren Wirksamkeit vergleichen. Wir sprechen nämlich über folgende Modelle:- Das Basismodell basiert auf dem RF-Algorithmus (wir werden dieses RF-Modell abkürzen).
- Das gleiche Modell wie Nr. 1, jedoch eines, bei dem eine Verringerung der Dimension des Merkmalsraums unter Verwendung der Hauptkomponentenmethode (RF + PCA) angewendet wird.
- Das gleiche Modell wie Nr. 2, jedoch mit Hyperparameteroptimierung (RF + PCA + HT) erstellt.
1. Daten importieren
Laden Sie zunächst die Daten und erstellen Sie einen Pandas-Datenrahmen. Da wir einen vorab gelöschten „Spielzeug“ -Datensatz von Scikit-learn verwenden, können wir danach bereits mit dem Modellierungsprozess beginnen. Aber selbst wenn Sie solche Daten verwenden, wird empfohlen, dass Sie immer mit der Arbeit beginnen, indem Sie eine vorläufige Analyse der Daten mit den folgenden Befehlen durchführen, die auf den Datenrahmen ( df) angewendet werden :df.head() - um einen Blick auf den neuen Datenrahmen zu werfen und zu sehen, ob er wie erwartet aussieht.df.info()- um die Merkmale von Datentypen und Spalteninhalten herauszufinden. Möglicherweise muss die Datentypkonvertierung durchgeführt werden, bevor Sie fortfahren können.df.isna()- um sicherzustellen, dass die Daten keine Werte enthalten NaN. Die entsprechenden Werte müssen gegebenenfalls irgendwie verarbeitet werden, oder es kann erforderlich sein, ganze Zeilen aus dem Datenrahmen zu entfernen.df.describe() - Ermittlung der minimalen, maximalen und durchschnittlichen Werte der Indikatoren in den Spalten, Ermittlung der Indikatoren für das mittlere Quadrat und die wahrscheinliche Abweichung in den Spalten.
In unserem Datensatz ist eine Spalte cancer(Krebs) die Zielvariable, deren Wert wir mithilfe des Modells vorhersagen möchten. 0bedeutet "keine Krankheit". 1- "das Vorhandensein der Krankheit."import pandas as pd
from sklearn.datasets import load_breast_cancer
columns = ['mean radius', 'mean texture', 'mean perimeter', 'mean area', 'mean smoothness', 'mean compactness', 'mean concavity', 'mean concave points', 'mean symmetry', 'mean fractal dimension', 'radius error', 'texture error', 'perimeter error', 'area error', 'smoothness error', 'compactness error', 'concavity error', 'concave points error', 'symmetry error', 'fractal dimension error', 'worst radius', 'worst texture', 'worst perimeter', 'worst area', 'worst smoothness', 'worst compactness', 'worst concavity', 'worst concave points', 'worst symmetry', 'worst fractal dimension']
dataset = load_breast_cancer()
data = pd.DataFrame(dataset['data'], columns=columns)
data['cancer'] = dataset['target']
display(data.head())
display(data.info())
display(data.isna().sum())
display(data.describe())
. . , cancer, , . 0 « ». 1 — « »2.
Teilen Sie nun die Daten mit der Scikit-Lernfunktion auf train_test_split. Wir möchten dem Modell so viele Trainingsdaten wie möglich geben. Wir müssen jedoch über genügend Daten verfügen, um das Modell zu testen. Im Allgemeinen können wir sagen, dass mit zunehmender Anzahl von Zeilen im Datensatz auch die Datenmenge zunimmt, die als lehrreich angesehen werden kann.Wenn beispielsweise Millionen von Zeilen vorhanden sind, können Sie den Satz aufteilen, indem Sie 90% der Zeilen für Trainingsdaten und 10% für Testdaten markieren. Der Testdatensatz enthält jedoch nur 569 Zeilen. Und das ist nicht so sehr für das Training und Testen des Modells. Um in Bezug auf die Trainings- und Verifizierungsdaten fair zu sein, werden wir den Satz in zwei gleiche Teile aufteilen - 50% - Trainingsdaten und 50% - Verifizierungsdaten. Wir installierenstratify=y um sicherzustellen, dass sowohl der Trainings- als auch der Testdatensatz das gleiche Verhältnis von 0 und 1 wie der Originaldatensatz haben.from sklearn.model_selection import train_test_split
X = data.drop('cancer', axis=1)
y = data['cancer']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.50, random_state = 2020, stratify=y)
3. Datenskalierung
Bevor Sie mit der Modellierung fortfahren, müssen Sie die Daten durch Skalieren „zentrieren“ und „standardisieren“ . Die Skalierung erfolgt aufgrund der Tatsache, dass unterschiedliche Größen in unterschiedlichen Einheiten ausgedrückt werden. Mit diesem Verfahren können Sie einen „fairen Kampf“ zwischen den Zeichen organisieren, um deren Bedeutung zu bestimmen. Außerdem konvertieren wir y_trainvom Pandas-Datentyp Seriesin das NumPy-Array, damit das Modell später mit den entsprechenden Zielen arbeiten kann.import numpy as np
from sklearn.preprocessing import StandardScaler
ss = StandardScaler()
X_train_scaled = ss.fit_transform(X_train)
X_test_scaled = ss.transform(X_test)
y_train = np.array(y_train)
4. Training des Grundmodells (Modell Nr. 1, RF)
Erstellen Sie nun Modellnummer 1. Darin erinnern wir uns, dass nur der Random Forest-Algorithmus verwendet wird. Es verwendet alle Funktionen und wird mit den Standardwerten konfiguriert (Details zu diesen Einstellungen finden Sie in der Dokumentation zu sklearn.ensemble.RandomForestClassifier ). Initialisieren Sie das Modell. Danach werden wir sie in skalierten Daten schulen. Die Genauigkeit des Modells kann an den Trainingsdaten gemessen werden:from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import recall_score
rfc = RandomForestClassifier()
rfc.fit(X_train_scaled, y_train)
display(rfc.score(X_train_scaled, y_train))
Wenn wir wissen möchten, welche Merkmale für das RF-Modell für die Vorhersage von Brustkrebs am wichtigsten sind, können wir Indikatoren für den Schweregrad von Merkmalen anhand des Attributs visualisieren und quantifizieren feature_importances_:feats = {}
for feature, importance in zip(data.columns, rfc_1.feature_importances_):
feats[feature] = importance
importances = pd.DataFrame.from_dict(feats, orient='index').rename(columns={0: 'Gini-Importance'})
importances = importances.sort_values(by='Gini-Importance', ascending=False)
importances = importances.reset_index()
importances = importances.rename(columns={'index': 'Features'})
sns.set(font_scale = 5)
sns.set(style="whitegrid", color_codes=True, font_scale = 1.7)
fig, ax = plt.subplots()
fig.set_size_inches(30,15)
sns.barplot(x=importances['Gini-Importance'], y=importances['Features'], data=importances, color='skyblue')
plt.xlabel('Importance', fontsize=25, weight = 'bold')
plt.ylabel('Features', fontsize=25, weight = 'bold')
plt.title('Feature Importance', fontsize=25, weight = 'bold')
display(plt.show())
display(importances)
Visualisierung der „Wichtigkeit“ von ZeichenSignifikanzindikatoren5. Die Methode der Hauptkomponenten
Fragen wir nun, wie wir das grundlegende HF-Modell verbessern können. Mithilfe der Technik zum Reduzieren der Dimension des Merkmalsraums ist es möglich, den anfänglichen Datensatz durch weniger Variablen darzustellen und gleichzeitig die Menge an Rechenressourcen zu reduzieren, die erforderlich sind, um den Betrieb des Modells sicherzustellen. Mithilfe der PCA können Sie die kumulative Stichprobenvarianz dieser Merkmale untersuchen, um zu verstehen, welche Merkmale den größten Teil der Varianz in den Daten erklären.Wir initialisieren das PCA ( pca_test) - Objekt und geben die Anzahl der Komponenten (Features) an, die berücksichtigt werden müssen. Wir setzen diesen Indikator auf 30, um die erklärte Varianz aller generierten Komponenten zu sehen, bevor wir entscheiden, wie viele Komponenten wir benötigen. Dann übertragen wir auf die pca_testskalierten DatenX_trainmit der Methode pca_test.fit(). Danach visualisieren wir die Daten.import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.decomposition import PCA
pca_test = PCA(n_components=30)
pca_test.fit(X_train_scaled)
sns.set(style='whitegrid')
plt.plot(np.cumsum(pca_test.explained_variance_ratio_))
plt.xlabel('number of components')
plt.ylabel('cumulative explained variance')
plt.axvline(linewidth=4, color='r', linestyle = '--', x=10, ymin=0, ymax=1)
display(plt.show())
evr = pca_test.explained_variance_ratio_
cvr = np.cumsum(pca_test.explained_variance_ratio_)
pca_df = pd.DataFrame()
pca_df['Cumulative Variance Ratio'] = cvr
pca_df['Explained Variance Ratio'] = evr
display(pca_df.head(10))
Nachdem die Anzahl der verwendeten Komponenten 10 überschreitet, erhöht die Erhöhung ihrer Anzahl die erklärte Varianz nicht wesentlichDieser Datenrahmen enthält Indikatoren wie Kumulative Variance Ratio (kumulative Größe der Varianz der Daten erklärten) und erklärte Varianz Ratio (Anteil der einzelnen Komponenten auf das Gesamtvolumen der erklärten Varianz)Wenn man sich den oben Datenrahmen aussehen, es stellt sich herausdass der PCA unter Verwendung von 30 Variablen 10 bewegen zu Komponenten ermöglicht es, 95% der Datenstreuung zu erklären. Die anderen 20 Komponenten machen weniger als 5% der Varianz aus, was bedeutet, dass wir sie ablehnen können. Nach dieser Logik verwenden wir die PCA, um die Anzahl der Komponenten fürX_trainundvon 30 auf 10 zu reduzierenX_test. Wir schreiben diese künstlich erstellten Datensätze mit reduzierter Dimension inX_train_scaled_pcaund inX_test_scaled_pca.pca = PCA(n_components=10)
pca.fit(X_train_scaled)
X_train_scaled_pca = pca.transform(X_train_scaled)
X_test_scaled_pca = pca.transform(X_test_scaled)
Jede Komponente ist eine lineare Kombination von Quellvariablen mit entsprechenden „Gewichten“. Wir können diese „Gewichte“ für jede Komponente sehen, indem wir einen Datenrahmen erstellen.pca_dims = []
for x in range(0, len(pca_df)):
pca_dims.append('PCA Component {}'.format(x))
pca_test_df = pd.DataFrame(pca_test.components_, columns=columns, index=pca_dims)
pca_test_df.head(10).T
Komponenteninformationsdatenrahmen6. Training des grundlegenden RF-Modells nach Anwendung der Hauptkomponentenmethode auf die Daten (Modell Nr. 2, RF + PCA)
Jetzt können wir zu einem anderen Grund RF-Modelldaten übergeben X_train_scaled_pcaund y_trainund können herausfinden, ob es eine Verbesserung der Genauigkeit der Vorhersagen durch das Modell ausgestellt ist.rfc = RandomForestClassifier()
rfc.fit(X_train_scaled_pca, y_train)
display(rfc.score(X_train_scaled_pca, y_train))
Modelle vergleichen unten.7. Optimierung von Hyperparametern. Runde 1: RandomizedSearchCV
Nach der Verarbeitung der Daten mit der Hauptkomponentenmethode können Sie versuchen, die Optimierung von Modellhyperparametern zu verwenden, um die Qualität der vom RF-Modell erzeugten Vorhersagen zu verbessern. Hyperparameter können als „Einstellungen“ des Modells betrachtet werden. Einstellungen, die für einen Datensatz perfekt sind, funktionieren für einen anderen nicht - deshalb müssen Sie sie optimieren.Sie können mit dem RandomizedSearchCV-Algorithmus beginnen, mit dem Sie einen weiten Wertebereich ziemlich grob untersuchen können. Beschreibungen aller Hyperparameter für RF-Modelle finden Sie hier .Im Laufe der Arbeit generieren wir eine Entität param_dist, die für jeden Hyperparameter einen Wertebereich enthält, der getestet werden muss. Als nächstes initialisieren wir das Objekt.rsRandomizedSearchCV()Übergeben der Funktion , Übergeben des RF-Modells param_dist, der Anzahl der Iterationen und der Anzahl der Kreuzvalidierungen , die durchgeführt werden müssen.Mit dem Hyperparameter verbosekönnen Sie die Informationsmenge steuern, die das Modell während seines Betriebs anzeigt (wie die Ausgabe von Informationen während des Trainings des Modells). Mit dem Hyperparameter n_jobskönnen Sie angeben, wie viele Prozessorkerne Sie verwenden müssen, um den Betrieb des Modells sicherzustellen. Das Festlegen n_jobseines Werts -1führt zu einem schnelleren Modell, da hierfür alle Prozessorkerne verwendet werden.Wir werden uns mit der Auswahl der folgenden Hyperparameter befassen:n_estimators - die Anzahl der "Bäume" im "zufälligen Wald".max_features - Die Anzahl der Features, für die die Aufteilung ausgewählt werden soll.max_depth - maximale Tiefe der Bäume.min_samples_split - Die Mindestanzahl von Objekten, die zum Teilen eines Baumknotens erforderlich sind.min_samples_leaf - die Mindestanzahl von Objekten in den Blättern.bootstrap - Verwendung zum Erstellen von Teilmusterbäumen mit Rückgabe.
from sklearn.model_selection import RandomizedSearchCV
n_estimators = [int(x) for x in np.linspace(start = 100, stop = 1000, num = 10)]
max_features = ['log2', 'sqrt']
max_depth = [int(x) for x in np.linspace(start = 1, stop = 15, num = 15)]
min_samples_split = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
min_samples_leaf = [int(x) for x in np.linspace(start = 2, stop = 50, num = 10)]
bootstrap = [True, False]
param_dist = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
rs = RandomizedSearchCV(rfc_2,
param_dist,
n_iter = 100,
cv = 3,
verbose = 1,
n_jobs=-1,
random_state=0)
rs.fit(X_train_scaled_pca, y_train)
rs.best_params_
Mit den Werten der Parameter n_iter = 100und cv = 3haben wir 300 RF-Modelle erstellt und zufällig Kombinationen der oben dargestellten Hyperparameter ausgewählt. best_params_ Informationen zu einer Reihe von Parametern, mit denen Sie das beste Modell erstellen können, finden Sie im Attribut . Zum jetzigen Zeitpunkt liefert dies jedoch möglicherweise nicht die interessantesten Daten zu den Parameterbereichen, die es wert sind, in der nächsten Optimierungsrunde untersucht zu werden. Um herauszufinden, in welchem Wertebereich es sich lohnt, weiter zu suchen, können wir leicht einen Datenrahmen erhalten, der die Ergebnisse des RandomizedSearchCV-Algorithmus enthält.rs_df = pd.DataFrame(rs.cv_results_).sort_values('rank_test_score').reset_index(drop=True)
rs_df = rs_df.drop([
'mean_fit_time',
'std_fit_time',
'mean_score_time',
'std_score_time',
'params',
'split0_test_score',
'split1_test_score',
'split2_test_score',
'std_test_score'],
axis=1)
rs_df.head(10)
Ergebnisse des RandomizedSearchCV-AlgorithmusNun erstellen wir Balkendiagramme, auf denen auf der X-Achse die Hyperparameterwerte und auf der Y-Achse die von den Modellen angezeigten Durchschnittswerte angegeben sind. Auf diese Weise können Sie nachvollziehen, welche Werte von Hyperparametern im Durchschnitt ihre beste Leistung zeigen.fig, axs = plt.subplots(ncols=3, nrows=2)
sns.set(style="whitegrid", color_codes=True, font_scale = 2)
fig.set_size_inches(30,25)
sns.barplot(x='param_n_estimators', y='mean_test_score', data=rs_df, ax=axs[0,0], color='lightgrey')
axs[0,0].set_ylim([.83,.93])axs[0,0].set_title(label = 'n_estimators', size=30, weight='bold')
sns.barplot(x='param_min_samples_split', y='mean_test_score', data=rs_df, ax=axs[0,1], color='coral')
axs[0,1].set_ylim([.85,.93])axs[0,1].set_title(label = 'min_samples_split', size=30, weight='bold')
sns.barplot(x='param_min_samples_leaf', y='mean_test_score', data=rs_df, ax=axs[0,2], color='lightgreen')
axs[0,2].set_ylim([.80,.93])axs[0,2].set_title(label = 'min_samples_leaf', size=30, weight='bold')
sns.barplot(x='param_max_features', y='mean_test_score', data=rs_df, ax=axs[1,0], color='wheat')
axs[1,0].set_ylim([.88,.92])axs[1,0].set_title(label = 'max_features', size=30, weight='bold')
sns.barplot(x='param_max_depth', y='mean_test_score', data=rs_df, ax=axs[1,1], color='lightpink')
axs[1,1].set_ylim([.80,.93])axs[1,1].set_title(label = 'max_depth', size=30, weight='bold')
sns.barplot(x='param_bootstrap',y='mean_test_score', data=rs_df, ax=axs[1,2], color='skyblue')
axs[1,2].set_ylim([.88,.92])
axs[1,2].set_title(label = 'bootstrap', size=30, weight='bold')
plt.show()
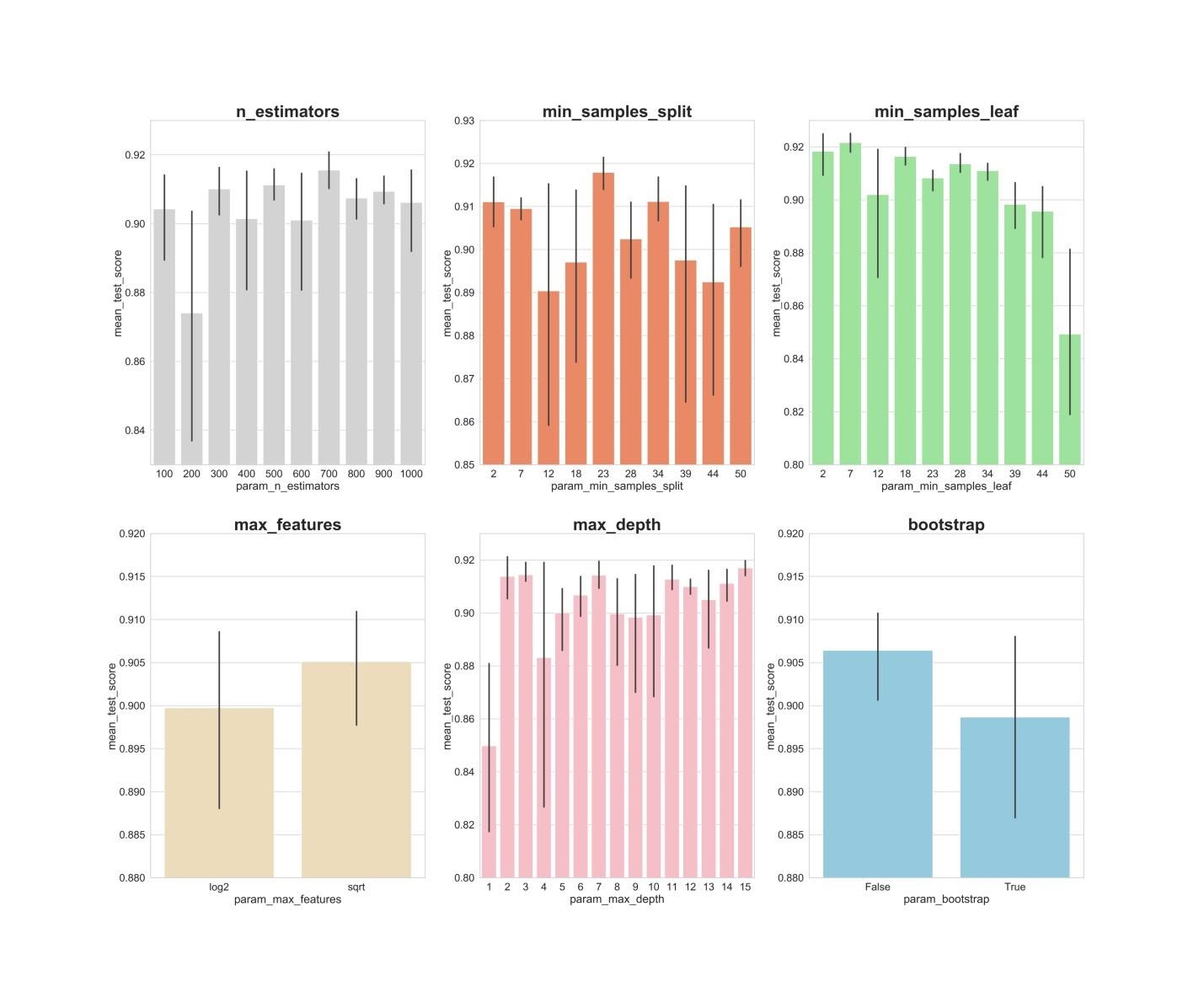
n_estimators: Werte von 300, 500, 700 zeigen anscheinend die besten durchschnittlichen Ergebnisse.min_samples_split: Kleine Werte wie 2 und 7 scheinen die besten Ergebnisse zu zeigen. Der Wert 23 sieht auch gut aus. Sie können mehrere Werte dieses Hyperparameters über 2 sowie mehrere Werte von etwa 23 untersuchen.min_samples_leaf: Es besteht das Gefühl, dass kleine Werte dieses Hyperparameters bessere Ergebnisse liefern. Dies bedeutet, dass wir Werte zwischen 2 und 7 erfahren können.max_features: Option sqrtgibt das höchste durchschnittliche Ergebnis.max_depth: Es gibt keine klare Beziehung zwischen dem Wert des Hyperparameters und dem Ergebnis des Modells, aber es besteht das Gefühl, dass die Werte 2, 3, 7, 11, 15 gut aussehen.bootstrap: Der Wert Falsezeigt das beste Durchschnittsergebnis.
Mit diesen Erkenntnissen können wir nun zur zweiten Runde der Optimierung von Hyperparametern übergehen. Dies wird den Wertebereich einschränken, an dem wir interessiert sind.8. Optimierung von Hyperparametern. Runde 2: GridSearchCV (endgültige Vorbereitung der Parameter für Modell Nr. 3, RF + PCA + HT)
Nach der Anwendung des RandomizedSearchCV-Algorithmus verwenden wir den GridSearchCV-Algorithmus, um eine genauere Suche nach der besten Kombination von Hyperparametern durchzuführen. Die gleichen Hyperparameter werden hier untersucht, aber jetzt wenden wir eine "gründlichere" Suche nach ihrer besten Kombination an. Mit dem GridSearchCV-Algorithmus wird jede Kombination von Hyperparametern untersucht. Dies erfordert viel mehr Rechenressourcen als die Verwendung des RandomizedSearchCV-Algorithmus, wenn wir die Anzahl der Suchiterationen unabhängig voneinander festlegen. Zum Beispiel erfordert die Untersuchung von 10 Werten für jeden der 6 Hyperparameter mit Kreuzvalidierung in 3 Blöcken 10⁶ x 3 oder 3.000.000 Modellschulungen. Aus diesem Grund verwenden wir den GridSearchCV-Algorithmus, nachdem wir nach der Anwendung von RandomizedSearchCV die Wertebereiche der untersuchten Parameter eingegrenzt haben.Mit dem, was wir mithilfe von RandomizedSearchCV herausgefunden haben, untersuchen wir die Werte der Hyperparameter, die sich am besten gezeigt haben:from sklearn.model_selection import GridSearchCV
n_estimators = [300,500,700]
max_features = ['sqrt']
max_depth = [2,3,7,11,15]
min_samples_split = [2,3,4,22,23,24]
min_samples_leaf = [2,3,4,5,6,7]
bootstrap = [False]
param_grid = {'n_estimators': n_estimators,
'max_features': max_features,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'bootstrap': bootstrap}
gs = GridSearchCV(rfc_2, param_grid, cv = 3, verbose = 1, n_jobs=-1)
gs.fit(X_train_scaled_pca, y_train)
rfc_3 = gs.best_estimator_
gs.best_params_
Hier wenden wir die Kreuzvalidierung in 3 Blöcken für 540 (3 x 1 x 5 x 6 x 6 x 1) Modellschulungen an, was 1620 Modellschulungen ergibt. Nachdem wir RandomizedSearchCV und GridSearchCV verwendet haben, können wir uns nun dem Attribut zuwenden, best_params_um herauszufinden, welche Werte von Hyperparametern es dem Modell ermöglichen, am besten mit dem untersuchten Datensatz zu arbeiten (diese Werte sind am Ende des vorherigen Codeblocks zu sehen). . Diese Parameter werden verwendet, um Modellnummer 3 zu erstellen.9. Bewertung der Qualität der Modelle anhand der Verifizierungsdaten
Jetzt können Sie die erstellten Modelle anhand der Verifizierungsdaten auswerten. Wir sprechen nämlich über diese drei Modelle, die ganz am Anfang des Materials beschrieben wurden.Schauen Sie sich diese Modelle an:y_pred = rfc.predict(X_test_scaled)
y_pred_pca = rfc.predict(X_test_scaled_pca)
y_pred_gs = gs.best_estimator_.predict(X_test_scaled_pca)
Erstellen Sie Fehlermatrizen für die Modelle und finden Sie heraus, wie gut jeder von ihnen Brustkrebs vorhersagen kann:from sklearn.metrics import confusion_matrix
conf_matrix_baseline = pd.DataFrame(confusion_matrix(y_test, y_pred), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_baseline_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_pca), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
conf_matrix_tuned_pca = pd.DataFrame(confusion_matrix(y_test, y_pred_gs), index = ['actual 0', 'actual 1'], columns = ['predicted 0', 'predicted 1'])
display(conf_matrix_baseline)
display('Baseline Random Forest recall score', recall_score(y_test, y_pred))
display(conf_matrix_baseline_pca)
display('Baseline Random Forest With PCA recall score', recall_score(y_test, y_pred_pca))
display(conf_matrix_tuned_pca)
display('Hyperparameter Tuned Random Forest With PCA Reduced Dimensionality recall score', recall_score(y_test, y_pred_gs))
Ergebnisse der Arbeit der drei Modelle.Hier wird die Metrik als "Rückruf" bezeichnet. Tatsache ist, dass es sich um eine Krebsdiagnose handelt. Daher sind wir äußerst daran interessiert, falsch negative Prognosen von Modellen zu minimieren.In Anbetracht dessen können wir den Schluss ziehen, dass das grundlegende HF-Modell die besten Ergebnisse lieferte. Die Vollständigkeitsrate betrug 94,97%. Im Testdatensatz wurden 179 krebskranke Patienten erfasst. Das Modell fand 170 von ihnen.Zusammenfassung
Diese Studie liefert eine wichtige Beobachtung. Manchmal funktioniert das RF-Modell, das die Hauptkomponentenmethode und die Optimierung von Hyperparametern in großem Maßstab verwendet, möglicherweise nicht so gut wie das üblichste Modell mit Standardeinstellungen. Dies ist jedoch kein Grund, sich nur auf die einfachsten Modelle zu beschränken. Ohne verschiedene Modelle auszuprobieren, ist es unmöglich zu sagen, welches das beste Ergebnis zeigt. Und bei Modellen, mit denen das Vorhandensein von Krebs bei Patienten vorhergesagt wird, können wir sagen, dass je besser das Modell ist, desto mehr Leben können gerettet werden.Liebe Leser! Welche Aufgaben lösen Sie mit maschinellen Lernmethoden?