مرحبا يا هابر! أريد اليوم أن أشارك مثالاً صغيراً عن كيفية إجراء التحليل العنقودي. في هذا المثال ، لن يجد القارئ الشبكات العصبية واتجاهات الموضة الأخرى. يمكن أن يعمل هذا المثال كنقطة مرجعية لإجراء تحليل عنقودي صغير وكامل للبيانات الأخرى. أي شخص مهتم - مرحبا بك في القط.
إجراء حجز على الفور ، لا تدعي هذه المقالة بأي حال من الأحوال أنها أكاديمية بالكامل ، أو تفرد النتائج التي تم الحصول عليها ، أو اكتمال تغطية المشكلة. تهدف المقالة إلى إظهار الخطوات الأساسية لتحليل الكتلة الكلاسيكي ، والتي يمكن استخدامها في دراسة بسيطة وذات مغزى (ربما قبل إجراء دراسة أكثر تفصيلاً). نرحب بأي تصحيحات وتعليقات وإضافات على الأسس الموضوعية.
البيانات عبارة عن عينة من استهلاك الكحول حسب البلد للفرد حسب نوع المشروبات الكحولية (البيرة والنبيذ والمشروبات الروحية ، وما إلى ذلك) لعام 2010 كنسبة مئوية من استهلاك الفرد من الكحول. تحتوي البيانات أيضًا على: متوسط استهلاك الفرد اليومي من الكحول بالجرام من الكحول النقي وجميع استهلاك الكحول (المسجل + غير المحسوب) للفرد (يشربون فقط لترًا من الكحول النقي).
وفي الوقت نفسه ، تنتمي كل دولة بشكل مشروط إلى إحدى المجموعات الجغرافية: الشرق والوسط والغرب. التقسيم تعسفي للغاية ومثير للجدل لأسباب مختلفة ، لكننا سننطلق مما لدينا. مصدر البيانات - تقرير الحالة العالمي للكحول والصحة 2014 ، ص 289-364
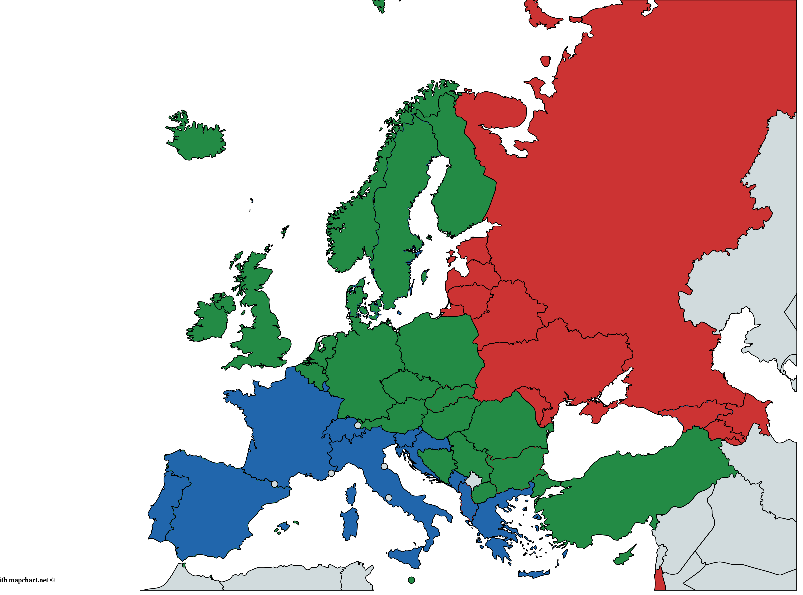
(مرسومة باليد ، قد تكون هناك أخطاء ، ولكن أعتقد أن الفكرة العامة مفهومة)
تحليل أولي
ربط المكتبات المستخدمة.
library(rgl)
library(heplots)
library(MVN)
library(klaR)
library('Morpho')
library(caret)
library(mclust)
library(ggplot2)
library(GGally)
library(plyr)
library(psych)
library(GPArotation)
library(ggpubr)
, .
#
data <- read.table("alcohol_data.csv", header=TRUE, sep=",")
#
rownames(data) <- make.names(data[,1], unique = TRUE)
# ,
data <- data[,-1]
data <- na.omit(data)
#
head(data)
summary(data)
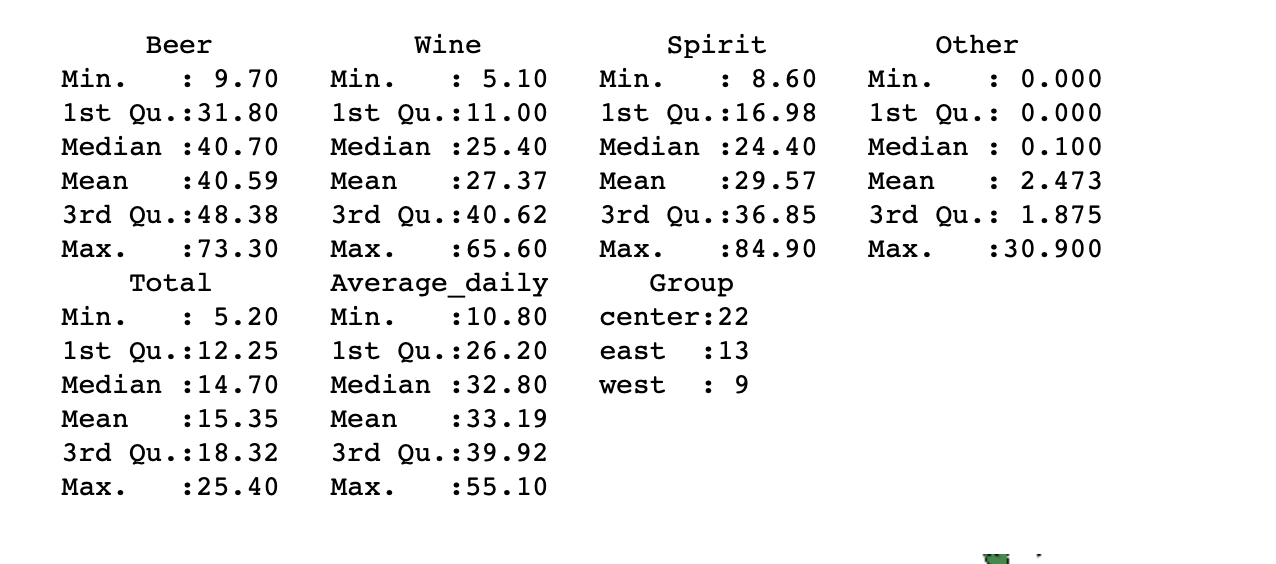
, . , Other , , , , . , , , , . , . - .
, , , .
options(rgl.useNULL=TRUE)
open3d()
mfrow3d(2,2)
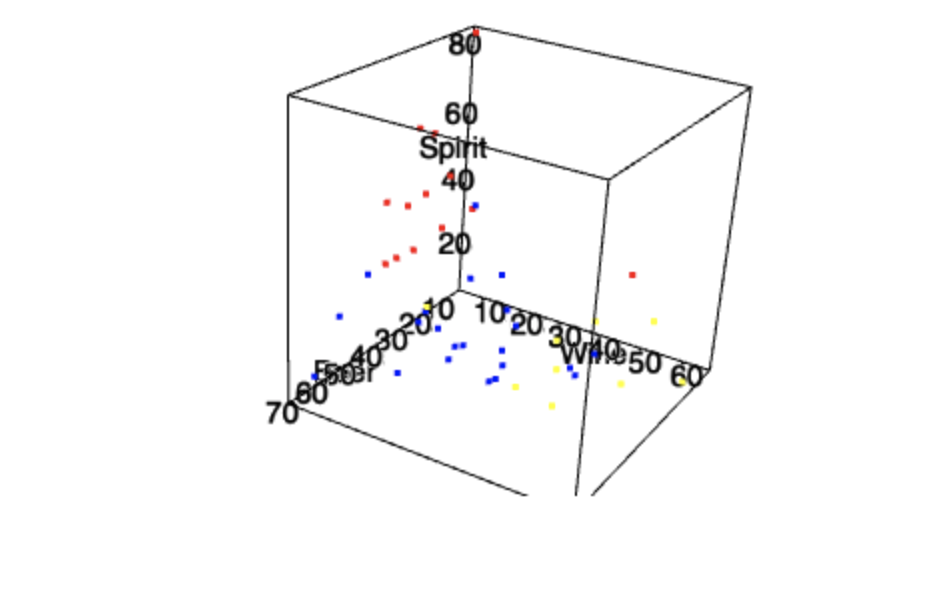
levelColors <- c('west'='blue', 'east'='red', 'center'='yellow')
plot3d(data$Beer, data$Wine, data$Spirit, xlab="Beer", ylab="Wine", zlab="Spirit", col = levelColors[data$Group], size=3)
widget <- rglwidget()
widget
, . , .
ggpairs(
data,
mapping = ggplot2::aes(color = data$Group),
upper = list(continuous = wrap("cor", alpha = 0.5), combo = "box"),
lower = list(continuous = wrap("points", alpha = 0.3), combo = wrap("dot", alpha = 0.4)),
diag = list(continuous = wrap("densityDiag",alpha = 0.5)),
title = "Alcohol"
)
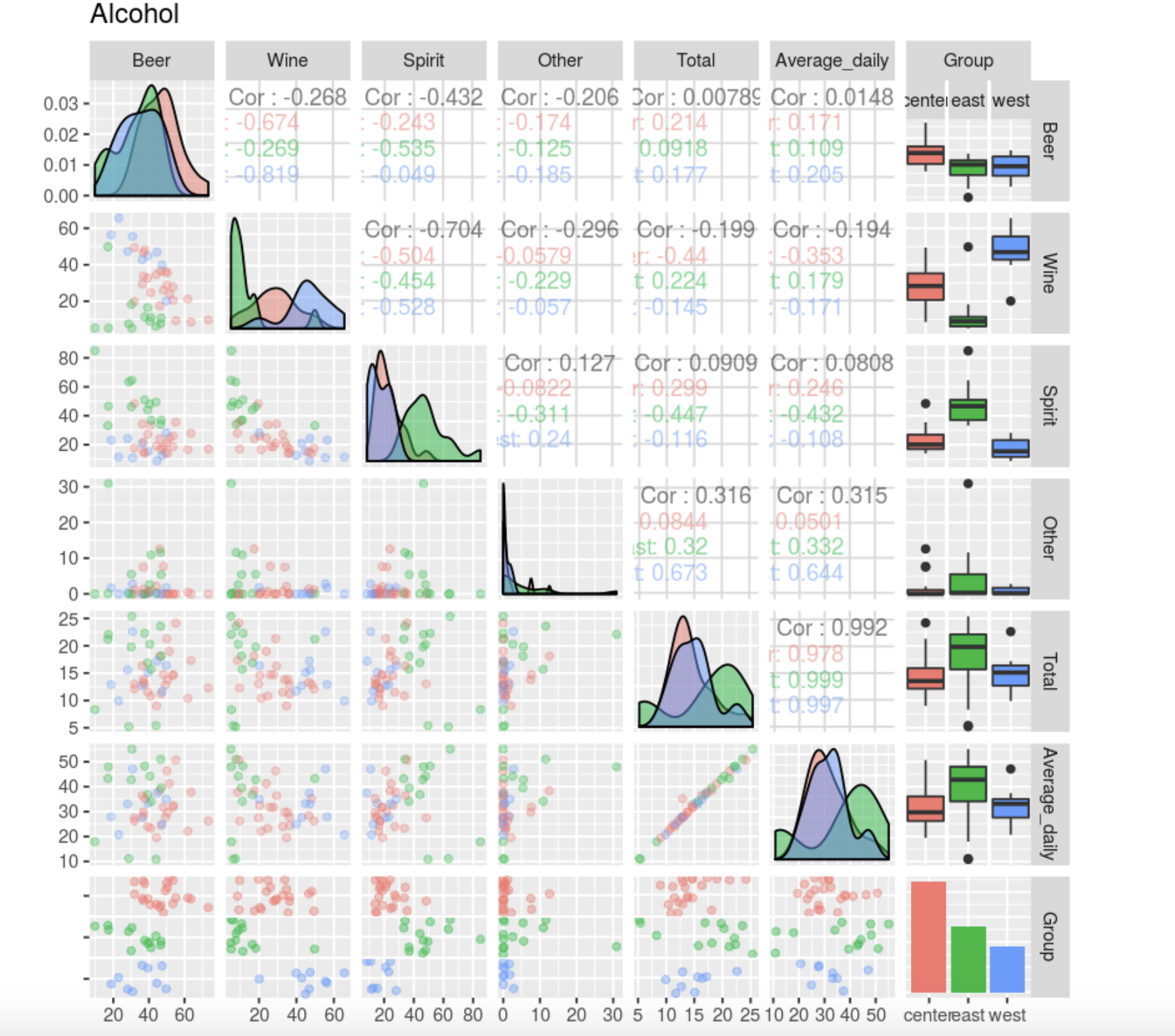
Average Total , Average.
data <- data[, -6]
, , , , . .
data[data$Wine>60,]
, , , , - , , .
data[data$Spirit>70,]
data[data$Spirit<10,]
, , .
,
split(data[,1:5],data$Group)
$center
$east
$west
ggpairs(
data,
mapping = ggplot2::aes(color = data$Group),
diag=list(continuous="bar", alpha=0.4)
)
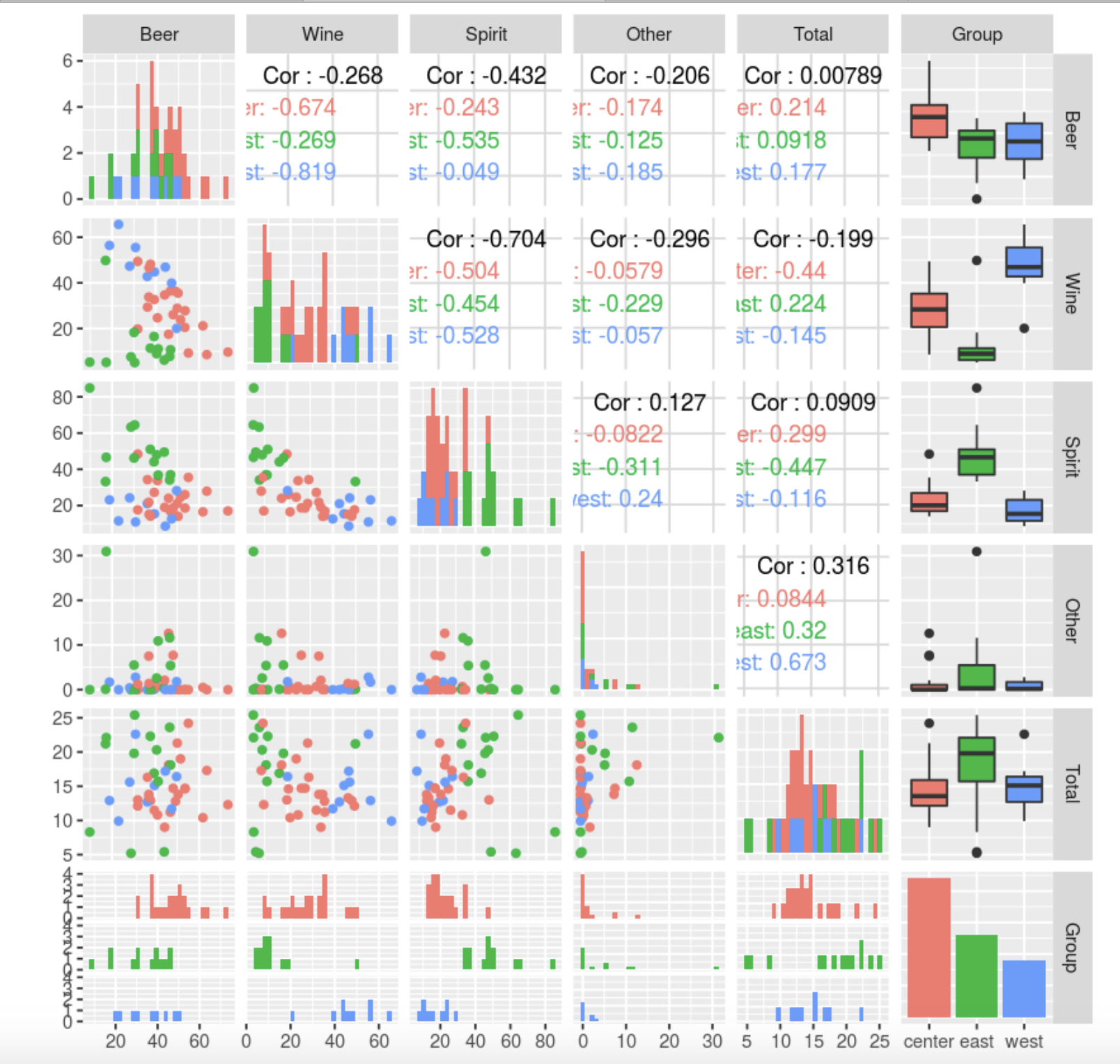
, , . Other, : , , , ( 10-12 , 45, , ). . , , , (). , , . Other .
, , — , — . , — , .
Total Other, . .
, Beer, Spirit Wine . , , , . , , , , , .
Total. , — .
data.group = data[,5]
data <- data[,-5]
data<- data[,-4]
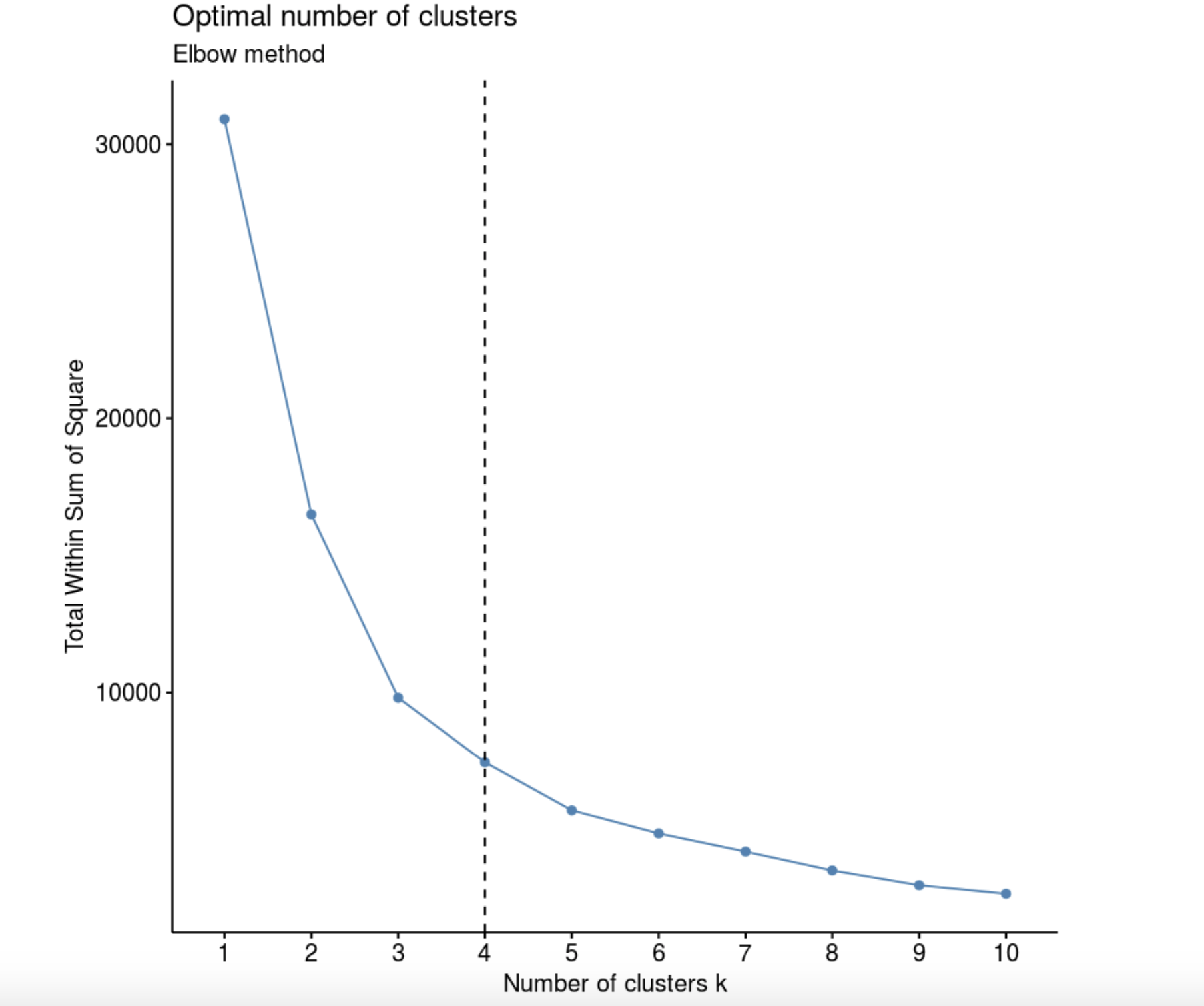
Elbow method (“ ”, “ ”). , k, – W(K), .
library(factoextra)
fviz_nbclust(data, kmeans, method = "wss") +
labs(subtitle = "Elbow method") +
geom_vline(xintercept = 4, linetype = 2)
data.dist <- dist((data))
hc <- hclust(data.dist, method = "ward.D2")
plot(hc, cex = 0.7)
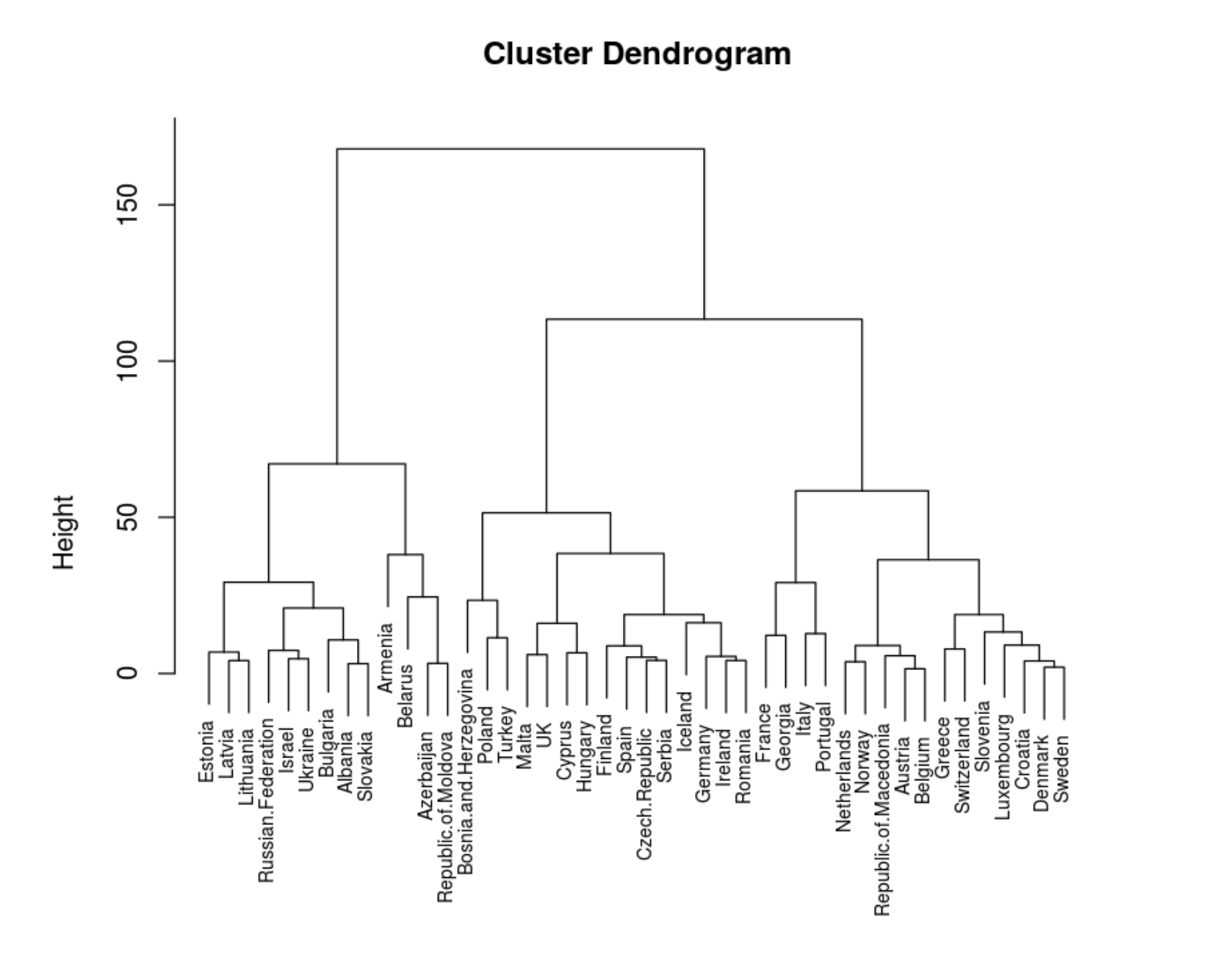
. .
colors=c('green', 'red', 'blue')
hcd = as.dendrogram(hc)
clusMember = cutree(hc, 4)
colLab <- function(n) {
if (is.leaf(n)) {
a <- attributes(n)
labCol <- colors[data.group[n]]
attr(n, "nodePar") <- c(a$nodePar, lab.col = labCol)
}
n
}
clusDendro = dendrapply(hcd, colLab)
plot(clusDendro, main = "Cool Dendrogram", type = "triangle")
rect.hclust(hc, k = 4)
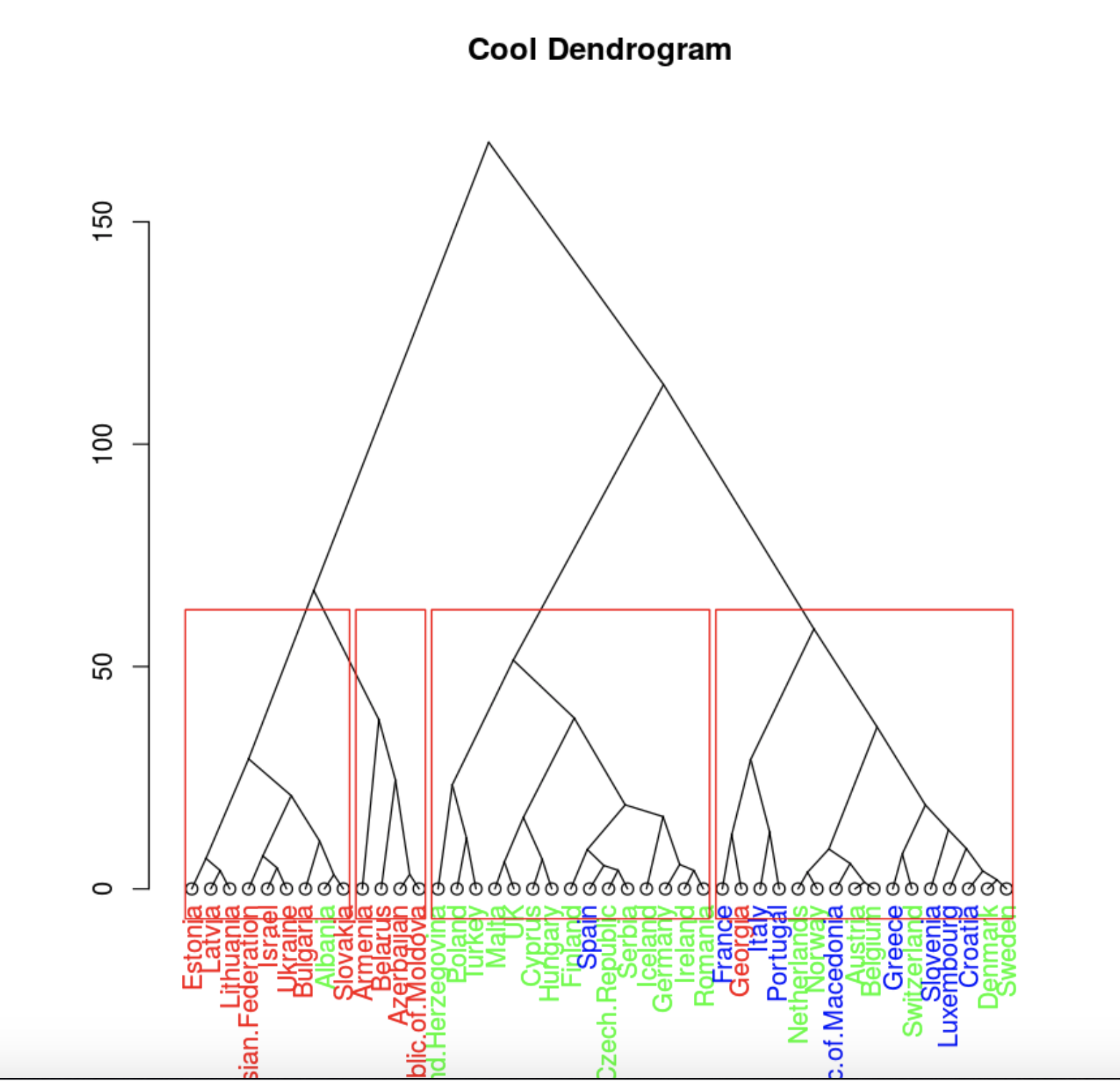
. , .
, , , 4 .
plot(clusDendro, main = "Cool Dendrogram", type = "triangle")
data.hclas_group <- factor(cutree(hc, k = 3))
rect.hclust(hc, k = 3)
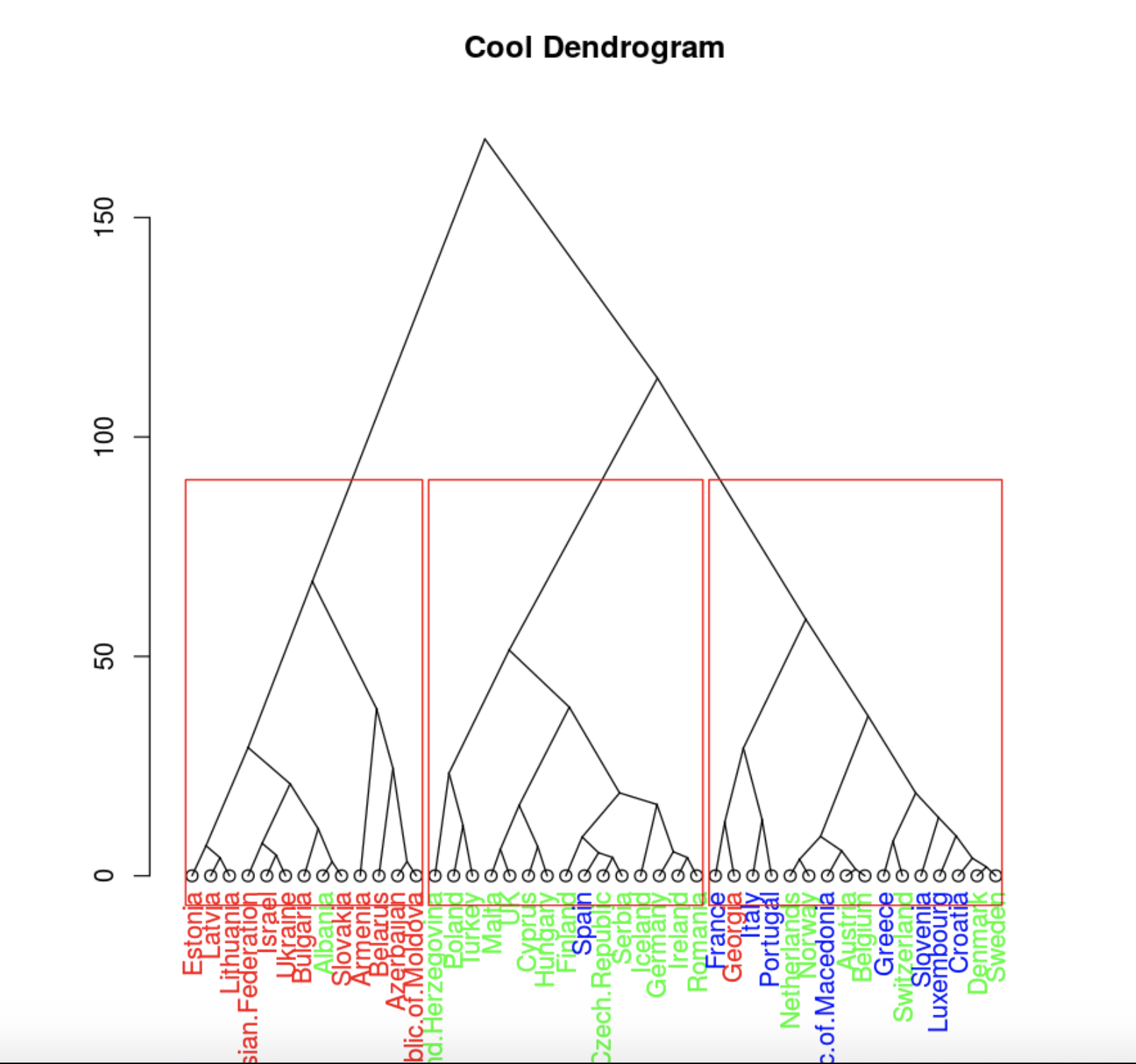
, , .
library(FactoMineR)
res.pca <- PCA(data,scale.unit=T, graph = F)
fviz_pca_biplot(res.pca,
col = colors[data.hclas_group], palette = "jco",
label = "var",
ellipse.level = 0.8,
addEllipses = T,
col.var = "black",
legend.title = "groups4")
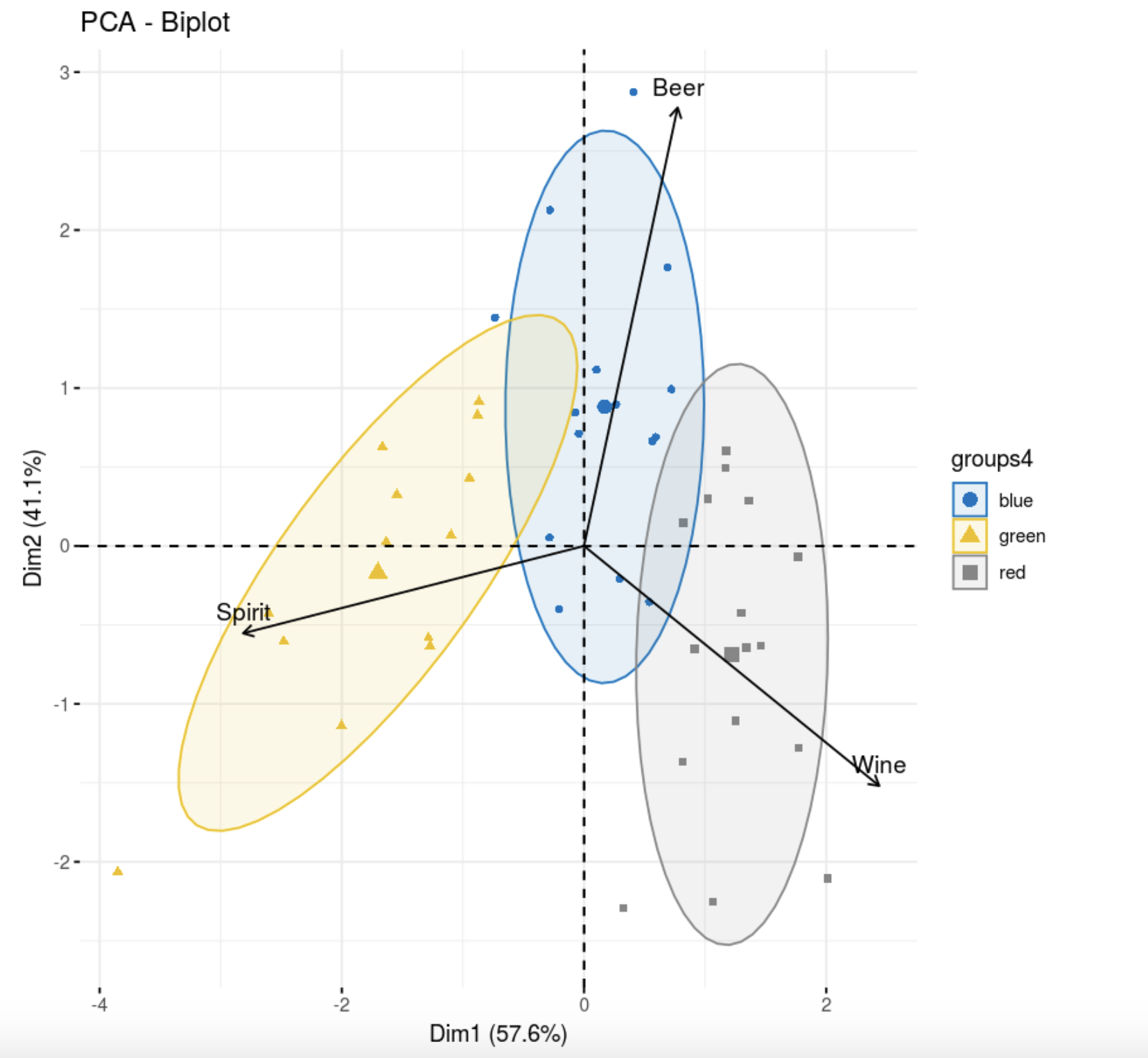
, , . , , , , . , , , k-++.
library(flexclust)
data.kk <- kcca(data, k=3, family=kccaFamily("kmeans"),
control=list(initcent="kmeanspp"))
fviz_pca_biplot(res.pca,
col.ind =as.factor(data.kk@cluster), palette = "jco",
label = "var",
ellipse.level = 0.8,
addEllipses = T,
col.var = "black", repel = TRUE,
legend.title = "clusters")
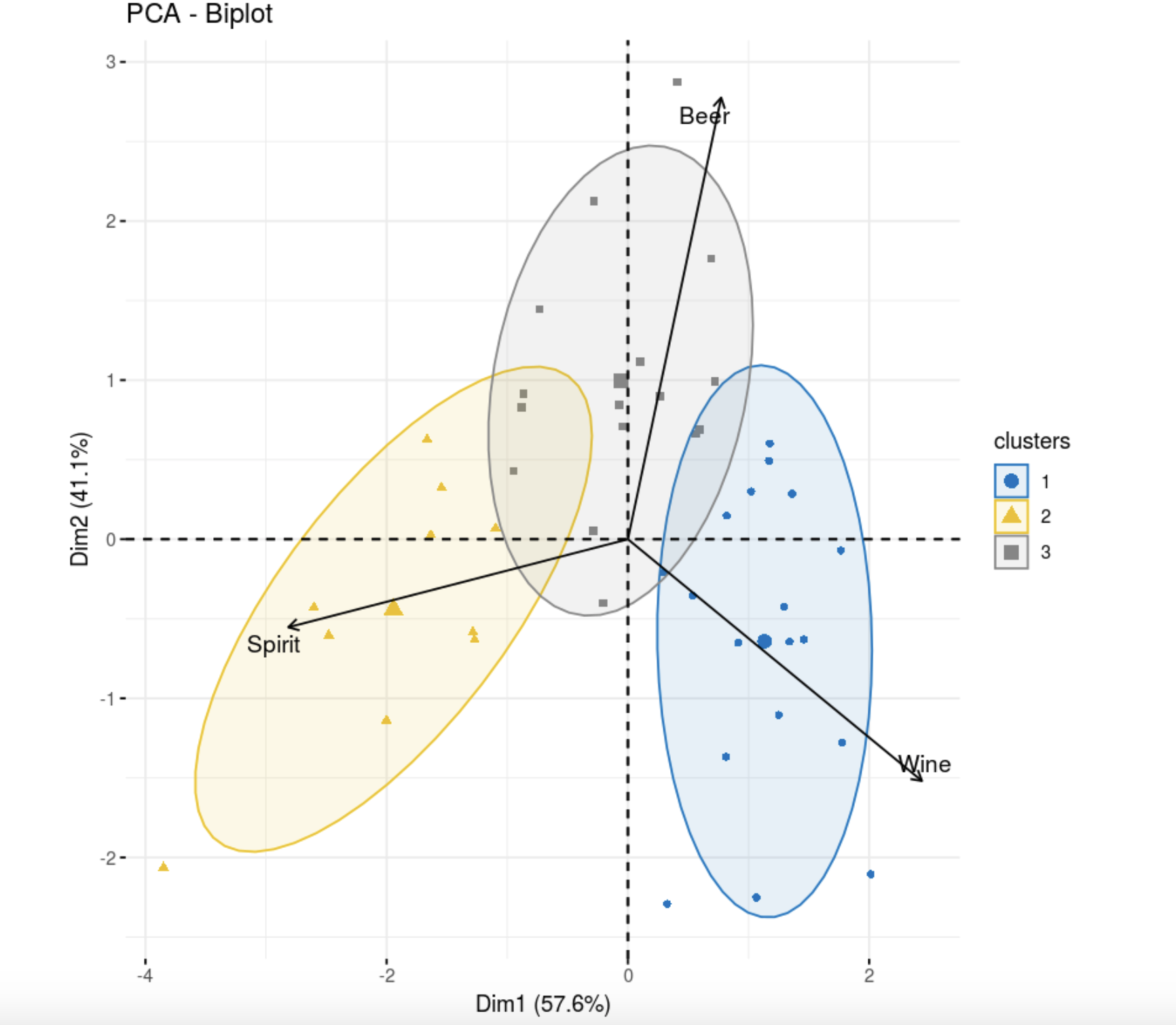
, k- . , , .
, , hclust. .

, , . . , .
. . , , , . , , . , .
سيكون من الممكن تنفيذ التجميع بناءً على افتراض النماذج العنقودية باستخدام معايير المعلومات ( هنا هو الوصف ) ، وتجربة أيضًا التحليل التمييزي الكلاسيكي لمجموعة البيانات هذه. إذا كانت هذه المقالة مفيدة ، أخطط لنشر تكملة.